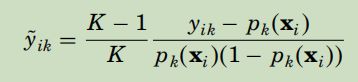sklearn源码解读:1.10 Decision Trees & 1.11 Ensemble methods
本文讨论sklearn源码中的树模型,其中包括 Decision Trees 与 Ensemble methods 两篇,源码文件夹在 sklearn/tree 与 sklearn/ensemble 下。
本文涉及到的模型参数解释请先自行翻阅官方文档,如非必要本文将不再列出。本文将简要介绍全部模型源码(工具源码不介绍),建议在看本文时自己打开源码对照查看,本文不再附大片源码来灌水。涉及到Cython语法细节本文将不详细讲解,因为我也看不懂,用着自己仅有的C++和Python那点知识勉强的在Cython的海洋中狗刨。
基于Cython底层架构
首先树模型的底层代码均由Cython编写,核心文件是为 _tree.pyx, _splitter.pyx,_criterion.pyx 。_utils.pyx 是源代码所使用的的工具文件,就不细说了。
先大致看一下架构:
-
_tree.pyx 是树模型的主架构,负责树的level生成,由Cython类Tree封装。生成树的结点有两种方法:DepthFirstTreeBuilder类与BestFirstTreeBuilder类。它们均继承自TreeBuilder类,其实就是为了继承它的_check_input方法,检查输入类型,并转化为占用连续内存储存形式(目的是加速计算与索引)如np.asfortranarray和np.ascontiguousarray格式。
-
_splitter.pyx 是负责叶节点分裂的架构,有四种分裂方式,两种Sparse处理,两种Dense处理。Dense处理中分为BestSplitter和RandomSplitter两种分裂方式,其实很容易理解,一种是正常的挑最好的特征和特征点分裂,一种是像随机分裂树那样瞎分裂。他们均继承自BaseDenseSplitter,继承它的初始化方法,而BaseDense继承自Splitter类,这是基类,提供了对接分裂准则方法的方法等。
-
_criterion.pyx 是负责叶节点分裂准则的架构,有N多种准则计算方法,有分类ClassificationCriterion和连续RegressionCriterion两种计算准则。这两种类均继承自Criterion基类,继承他的对接Splitter接口。就拿默认的分类准则基尼指数举例子。Gini类继承自ClassificationCriterion,Gini类仅提供了自己的计算准则方法(算基尼指数)。
_tree.pyx
主要有两种构造树的方式,DepthFirstTreeBuilder和BestFirstTreeBuilder,当不限制叶子结点数的时候就会使用DepthFirstTreeBuilder,使用栈的方式递归先在左子树分裂;限制叶子结点数的时候就会BestFirstTreeBuilder,使用优先级队列的方式将得分最大叶子结点进行分裂;堆和栈都定义在util.pyx中,比较常规的定义,就不说了。
DepthFirstTreeBuilder&BestFirstTreeBuilder类
-
_check_input :检查输入类型并转化为占用连续内存储存形式;
-
build:建立树;
-
_add_split_node(BestFirstTreeBuilder):添加树节点
Tree类
-
_resize、_resize_c:设置内存缓冲区;
-
_add_node:添加树节点;
-
apply:返回叶子索引;
-
_apply_dense、_apply_sparse_csr:返回dense和sparse树叶子节点索引;
-
_get_value_ndarray:返回所有树节点各类别权重和;
-
_get_node_ndarray:返回节点;
-
predict:返回当前节点各类别权重和权重和;
-
decision_path:返回决策路径;
-
_decision_path_dense、_decision_path_sparse_csr:返回dense和sparse树决策路径;
-
compute_feature_importances:计算特征重要度,(当前节点的样本权重数*损失数-左右节点的样本权重数*损失数)/样本权重数,然后再归一化即可得,计算方法如下所示;
cpdef compute_feature_importances(self, normalize=True):
...
with nogil:
while node != end_node:
if node.left_child != _TREE_LEAF:
# ... and node.right_child != _TREE_LEAF:
left = &nodes[node.left_child]
right = &nodes[node.right_child]
importance_data[node.feature] += (
node.weighted_n_node_samples * node.impurity -
left.weighted_n_node_samples * left.impurity -
right.weighted_n_node_samples * right.impurity)
node += 1
importances /= nodes[0].weighted_n_node_samples
if normalize:
normalizer = np.sum(importances)
if normalizer > 0.0:
# Avoid dividing by zero (e.g., when root is pure)
importances /= normalizer
return importances_splitter.pyx
有两类四种分裂方式,BestSplitter是最优分裂,跟正常的分裂方式一样,RandomSplitter是随机分裂。
BestSplitter&RandomSplitter类
-
init :初始化,有效样本(样本权重>0)samples,所有样本权重和 weighted_n_samples,预排序特征指针X_idx_sorted_ptr,预排序特征内存间隔 X_idx_sorted_stride,有效样本标志位 sample_mask;
-
node_reset: 初始化ClassificationCriterion类的样本区间,得到区间样本权重和 weighted_n_node_samples;
-
node_impurity:计算当前节点得分数;
-
node_split:分裂节点,使用了Fisher-Yates随机方法选取特征列,记录常量特征剔除出运算;
-
node_value:保存得分,将类别权重和 sum_total 复制到树的 value 内存,当做树得分;
对于稀疏类BestSparseSplitter和RandomSparseSplitter,他们相比于Dense类来说仅仅多了一个排序过程,由于稀疏类的特性,对他们进行如Dense类的预排序性能是非常差的:Python内置sort函数是基于快速排序,对于稀疏问题快排时间复杂度直接飙升到O(n方),使用插入排序效率最高,但还是O(n)的复杂度,所以sklearn源码中选择了只sort排序非0特征,时间复杂度只有O(m),其中m<
-
extract_nnz:提取非零特征;
-
extract_nnz_index_to_samples:通过样本索引值 提取非零特征;
-
extract_nnz_binary_search:通过二分法查找 提取非零特征;
-
sparse_swap:交换样本位置,使正特征值样本在零特征值样本之后;
-
_partition:分割样本;
-
binary_search:二分法查找当前样本行索引以确定对应特征是否为非零;
注:extract_nnz_index_to_samples 和 extract_nnz_binary_search 两函数使用的判断条件为:
(1 - is_samples_sorted[0]) * n_samples * log(n_samples) +n_samples * log(n_indices) < EXTRACT_NNZ_SWITCH * n_indices)_criterion.pyx
ClassificationCriterion&RegressionCriterion类
-
init:初始化,类别权重和 sum_total , 区间样本权重和 weighted_n_node_samples,调用reset方法;
-
reset:初始化分裂结构,左子树为空,右子树为全部样本;
-
reverse_reset:初始化分裂结构,右子树为空,左子树为全部样本;
-
node_impurity:计算当前节点得分数;
-
children_impurity:计算左右子节点得分数;
-
update:更新分割点,计算分割点左右节点类别数与权重和;
-
proxy_impurity_improvement:计算节点总得分
-
node_value:保存得分,将类别权重和 sum_total 复制到树的 value 内存,当做树得分;
基于Python的Tree架构
在介绍完Cython底层架构后,介绍基于Python的树模型架构,主要代码位于sklearn/tree/tree.py 和 sklearn/base.py ,在sklearn对外接口有DecisionTreeClassifier,DecisionTreeRegressor,ExtraTreeClassifier,ExtraTreeRegressor四种,他们全部继承于BaseDecisionTree类和ClassifierMixin/RegressorMixin基类,具体接口内容文档上都有,业务代码比较简单,不介绍了。
BaseDecisionTree继承于BaseEstimator基类,BaseEstimator提供了所有预测器的基本操作接口,例如get_params操作等。
ClassifierMixin/RegressorMixin基类则提供了score接口。
自此1.10节的 Decision Trees 的内容就全部介绍完毕了, 1.11节的 Ensemble methods 的代码继承自sklearn/tree 文件夹下的代码。
基于Python的Ensemble架构
只要了解了Tree文件夹内的Cython代码,Ensemble架构就很好理解,本文所讲的Ensemble内使用的基学习器都是Tree模型(bagging包含其他模型),在Ensemble架构中我们需要了解的就是并行化训练操作。
bagging.py&forest.py
RandomForest的代码和bagging的代码高度相似,本文只讲解随机森林的代码。
随机森林有4个接口(RandomTreesEmbedding忽略):RandomForestClassifier、RandomForestRegressor、ExtraTreesClassifier、ExtraTreesRegressor;他们的基学习器都在Tree内,遂本节仅以RandomForestClassifier为主。
RandomForestClassifier继承于ForestClassifier、BaseForest和BaseEnsemble,其中BaseEnsemble提供了_make_estimator的方法,用来制作estimator。
BaseForest提供了与底层对接的基础训练接口,通过_parallel_build_trees对基学习期进行并行训练。
随机森林选择使用将所有基学习器预测概率相加,取概率和最大类别为样本输出类别(即文档中所说的平均概率),_accumulate_prediction函数定义了这一操作。
在随机性方面,bagging写的通俗易懂,在max_features和max_samples上处理的非常清晰,bagging提供了随机样本和随机特征,_generate_indices,_generate_bagging_indices两个函数直接定义了随意抽取;相反RandomForest就写的特别迷,按理说直接把bagging这一套继承来就可以了,可是这代码接口写的,特征不能随机化了,样本也跟总样本数一样不能变,唯一能变的就是靠重复提取样本来压缩不同样本数,特别特别迷。。
注:在实际使用时如果想用跟理论相同的随机森林,那就使用bagging方法,基学习器使用DecisionTree。
weight_boosting.py
AdaBoost的代码实现,只有AdaBoostClassifier和AdaBoostRegressor两个接口,都是对基础AdaBoost的扩展,使之可以进行多分类和回归;他们两个都继承于BaseWeightBoosting和BaseEnsemble。本节代码都是按照论文敲公式,没啥可说的,多分类的参考可参考论文,本文仅给出一个连接:https://www.cnblogs.com/yeluzi/p/7117075.html 。
值得注意的是,Adaboost需要保证基学习器有大于50%(二分类)的准确率,如果不能保证那么其理论推导是要出错的,所以在源代码中直接限制准确率,如果小于阈值则报错,报错提示也很简单粗暴:“你这预测器还不如瞎编来的准呢,老子不干了”。
gradient_boosting.py
梯度提升树(GBDT),来到了最重要的一节,本节将附源代码,简单讲解原理。
GBDT源自于Friedman的《GREEDY FUNCTION APPROXIMATION:A GRADIENT BOOSTING MACHINE》,GBDT与AdaBoost一同属于加法模型,GBDT是对上一轮预测器结果与真实label的残差进行梯度拟合,至于为什么不直接对残差进行拟合,网上也有观点如下:
-
负梯度方向可证,模型优化下去一定收敛
-
对于一些损失函数来说最大的残差方向,并不是梯度下降最好的方向,倒是损失函数最小与残差最小两者目标不统一
-
梯度可以拟合一些并不能直接最小化的复杂损失函数
个人感觉梯度拟合就像学习率一样,通过多个加法模型结合减少过拟合的发生。
在拟合一个回归树后还需要计算叶子结点得分来更新树的叶子结点权重。
接下来先看拟合过程,附备注的fit函数源代码如下:
def fit(self, X, y, sample_weight=None, monitor=None):
X = check_array(X, accept_sparse=['csr', 'csc', 'coo'], dtype=DTYPE) #检查格式
n_samples, self.n_features_ = X.shape
sample_weight_is_none = sample_weight is None
if sample_weight_is_none:
sample_weight = np.ones(n_samples, dtype=np.float32)
else:
sample_weight = column_or_1d(sample_weight, warn=True)
sample_weight_is_none = False
check_consistent_length(X, y, sample_weight)
y = check_array(y, accept_sparse='csc', ensure_2d=False, dtype=None)
y = column_or_1d(y, warn=True)
y = self._validate_y(y, sample_weight)
#重定义特征类别:original_y = [7,8,9,5,5], modify_y=[0,1,2,3,3]
if self.n_iter_no_change is not None:
#如设置早停,则需分离验证集
stratify = y if is_classifier(self) else None
X, X_val, y, y_val, sample_weight, sample_weight_val = (
train_test_split(X, y, sample_weight,
random_state=self.random_state,
test_size=self.validation_fraction,
stratify=stratify))
if is_classifier(self):
if self.n_classes_ != np.unique(y).shape[0]:
# We choose to error here. The problem is that the init
# estimator would be trained on y, which has some missing
# classes now, so its predictions would not have the
# correct shape.
raise ValueError(
'The training data after the early stopping split '
'is missing some classes. Try using another random '
'seed.'
)
else:
X_val = y_val = sample_weight_val = None
self._check_params() #对参数检查赋值
if not self._is_initialized():
#如无初始化模型,则有两种选择,当 self.init_ == 'zero'时,认为预测结果全部为0
#self.init_ != 'zero' 时,源代码会使用dummy模型来进行预测拟合,保存预测结果
#如有初始化模型即 warm_start == True 时使用之前储存的模型进行预测
# init state
self._init_state()
# fit initial model and initialize raw predictions
if self.init_ == 'zero':
raw_predictions = np.zeros(shape=(X.shape[0], self.loss_.K),
dtype=np.float64)
else:
# XXX clean this once we have a support_sample_weight tag
if sample_weight_is_none:
self.init_.fit(X, y)
else:
msg = ("The initial estimator {} does not support sample "
"weights.".format(self.init_.__class__.__name__))
try:
self.init_.fit(X, y, sample_weight=sample_weight)
except TypeError: # regular estimator without SW support
raise ValueError(msg)
except ValueError as e:
if "pass parameters to specific steps of "\
"your pipeline using the "\
"stepname__parameter" in str(e): # pipeline
raise ValueError(msg) from e
else: # regular estimator whose input checking failed
raise
raw_predictions = \
self.loss_.get_init_raw_predictions(X, self.init_)
begin_at_stage = 0
# The rng state must be preserved if warm_start is True
self._rng = check_random_state(self.random_state)
else:
# add more estimators to fitted model
# invariant: warm_start = True
if self.n_estimators < self.estimators_.shape[0]:
raise ValueError('n_estimators=%d must be larger or equal to '
'estimators_.shape[0]=%d when '
'warm_start==True'
% (self.n_estimators,
self.estimators_.shape[0]))
begin_at_stage = self.estimators_.shape[0]
# The requirements of _decision_function (called in two lines
# below) are more constrained than fit. It accepts only CSR
# matrices.
X = check_array(X, dtype=DTYPE, order="C", accept_sparse='csr')
raw_predictions = self._raw_predict(X)
self._resize_state()
if self.presort is True and issparse(X):
raise ValueError(
"Presorting is not supported for sparse matrices.")
presort = self.presort
# Allow presort to be 'auto', which means True if the dataset is dense,
# otherwise it will be False.
if presort == 'auto':
presort = not issparse(X)
X_idx_sorted = None
if presort:
X_idx_sorted = np.asfortranarray(np.argsort(X, axis=0),
dtype=np.int32)
# fit the boosting stages
n_stages = self._fit_stages(
X, y, raw_predictions, sample_weight, self._rng, X_val, y_val,
sample_weight_val, begin_at_stage, monitor, X_idx_sorted)
# change shape of arrays after fit (early-stopping or additional ests)
if n_stages != self.estimators_.shape[0]:
self.estimators_ = self.estimators_[:n_stages]
self.train_score_ = self.train_score_[:n_stages]
if hasattr(self, 'oob_improvement_'):
self.oob_improvement_ = self.oob_improvement_[:n_stages]
self.n_estimators_ = n_stages
return self业务逻辑比较通俗易懂,首次拟合时会出现两种状况,备注里也都写了,在暖启动预测时会调用_gradient_boosting.pyx文件,这里面函数是预测叶子结点得分的,与Tree_.pyx内的predict功能一样,我不是很懂为什么要单独写一个,注释写着加速,可能就是加速吧,我也运行不了Cython。。。拟合fit函数会调用_fit_stages对estimator进行逐个拟合,_fit_stages函数源代码如下:
def _fit_stages(self, X, y, raw_predictions, sample_weight, random_state,
X_val, y_val, sample_weight_val,
begin_at_stage=0, monitor=None, X_idx_sorted=None):
n_samples = X.shape[0]
do_oob = self.subsample < 1.0
sample_mask = np.ones((n_samples, ), dtype=np.bool)
n_inbag = max(1, int(self.subsample * n_samples)) #计算训练样本数
loss_ = self.loss_
# Set min_weight_leaf from min_weight_fraction_leaf
if self.min_weight_fraction_leaf != 0. and sample_weight is not None:
min_weight_leaf = (self.min_weight_fraction_leaf *
np.sum(sample_weight))
else:
min_weight_leaf = 0.
if self.verbose:
verbose_reporter = VerboseReporter(self.verbose)
verbose_reporter.init(self, begin_at_stage)
X_csc = csc_matrix(X) if issparse(X) else None
X_csr = csr_matrix(X) if issparse(X) else None
if self.n_iter_no_change is not None:
loss_history = np.full(self.n_iter_no_change, np.inf)
# We create a generator to get the predictions for X_val after
# the addition of each successive stage
y_val_pred_iter = self._staged_raw_predict(X_val)
# perform boosting iterations
i = begin_at_stage
for i in range(begin_at_stage, self.n_estimators):
# subsampling
if do_oob:
sample_mask = _random_sample_mask(n_samples, n_inbag,
random_state) #样本标志位
# OOB score before adding this stage
old_oob_score = loss_(y[~sample_mask],
raw_predictions[~sample_mask],
sample_weight[~sample_mask])
# fit next stage of trees
raw_predictions = self._fit_stage(
i, X, y, raw_predictions, sample_weight, sample_mask,
random_state, X_idx_sorted, X_csc, X_csr)
# track deviance (= loss)
if do_oob:
self.train_score_[i] = loss_(y[sample_mask],
raw_predictions[sample_mask],
sample_weight[sample_mask])
self.oob_improvement_[i] = (
old_oob_score - loss_(y[~sample_mask],
raw_predictions[~sample_mask],
sample_weight[~sample_mask]))
else:
# no need to fancy index w/ no subsampling
self.train_score_[i] = loss_(y, raw_predictions, sample_weight)
if self.verbose > 0:
verbose_reporter.update(i, self)
if monitor is not None:
early_stopping = monitor(i, self, locals())
if early_stopping:
break
# We also provide an early stopping based on the score from
# validation set (X_val, y_val), if n_iter_no_change is set
if self.n_iter_no_change is not None:
# By calling next(y_val_pred_iter), we get the predictions
# for X_val after the addition of the current stage
validation_loss = loss_(y_val, next(y_val_pred_iter),
sample_weight_val)
# Require validation_score to be better (less) than at least
# one of the last n_iter_no_change evaluations
if np.any(validation_loss + self.tol < loss_history):
loss_history[i % len(loss_history)] = validation_loss
else:
break
return i + 1虽然是boosting,但由于有subsample的存在,所以是可以求oob_score的,没有注释是因为没什么好注释的,这里的_random_sample_mask就是无放回抽样,开始选样本了,GBDT的训练过程很有意思的是,subsample这个参数在bagging内是直接选择其中max_samples个样本进行分裂的,而GBDT中是用全部sample分裂,然后在计算叶子结点权重时使用subsample,很迷。。
接下来看最重要的_fit_stage函数:
def _fit_stage(self, i, X, y, raw_predictions, sample_weight, sample_mask,
random_state, X_idx_sorted, X_csc=None, X_csr=None):
assert sample_mask.dtype == np.bool
loss = self.loss_
original_y = y
# Need to pass a copy of raw_predictions to negative_gradient()
# because raw_predictions is partially updated at the end of the loop
# in update_terminal_regions(), and gradients need to be evaluated at
# iteration i - 1.
raw_predictions_copy = raw_predictions.copy()
for k in range(loss.K):
if loss.is_multi_class:
y = np.array(original_y == k, dtype=np.float64)
residual = loss.negative_gradient(y, raw_predictions_copy, k=k,
sample_weight=sample_weight)
# induce regression tree on residuals
tree = DecisionTreeRegressor(
criterion=self.criterion,
splitter='best',
max_depth=self.max_depth,
min_samples_split=self.min_samples_split,
min_samples_leaf=self.min_samples_leaf,
min_weight_fraction_leaf=self.min_weight_fraction_leaf,
min_impurity_decrease=self.min_impurity_decrease,
min_impurity_split=self.min_impurity_split,
max_features=self.max_features,
max_leaf_nodes=self.max_leaf_nodes,
random_state=random_state,
presort=self.presort)
if self.subsample < 1.0:
# no inplace multiplication!
sample_weight = sample_weight * sample_mask.astype(np.float64)
X = X_csr if X_csr is not None else X
tree.fit(X, residual, sample_weight=sample_weight,
check_input=False, X_idx_sorted=X_idx_sorted)
# update tree leaves
loss.update_terminal_regions(
tree.tree_, X, y, residual, raw_predictions, sample_weight,
sample_mask, learning_rate=self.learning_rate, k=k)
# add tree to ensemble
self.estimators_[i, k] = tree
return raw_predictionsGBDT总体业务逻辑是根据你设定的n_estimator进行循环,在每次循环中都会按照类别数n_class建立n_class个数,对每一类别用一棵树进行单独拟合。回归树拟合后再更新叶子结点权重。以分类为例,所需工具函数如下所示:
def update_terminal_regions(self, tree, X, y, residual, raw_predictions,
sample_weight, sample_mask,
learning_rate=0.1, k=0):
# compute leaf for each sample in ``X``.
terminal_regions = tree.apply(X)
# mask all which are not in sample mask.
masked_terminal_regions = terminal_regions.copy()
masked_terminal_regions[~sample_mask] = -1
# update each leaf (= perform line search)
for leaf in np.where(tree.children_left == TREE_LEAF)[0]:
self._update_terminal_region(tree, masked_terminal_regions,
leaf, X, y, residual,
raw_predictions[:, k], sample_weight)
# update predictions (both in-bag and out-of-bag)
raw_predictions[:, k] += \
learning_rate * tree.value[:, 0, 0].take(terminal_regions, axis=0)
def _update_terminal_region(self, tree, terminal_regions, leaf, X, y,
residual, raw_predictions, sample_weight):
"""Make a single Newton-Raphson step. """
terminal_region = np.where(terminal_regions == leaf)[0]
residual = residual.take(terminal_region, axis=0)
y = y.take(terminal_region, axis=0)
sample_weight = sample_weight.take(terminal_region, axis=0)
numerator = np.sum(sample_weight * residual)
numerator *= (self.K - 1) / self.K
denominator = np.sum(sample_weight * (y - residual) *
(1 - y + residual))
# prevents overflow and division by zero
if abs(denominator) < 1e-150:
tree.value[leaf, 0, 0] = 0.0
else:
tree.value[leaf, 0, 0] = numerator / denominator从以上两个函数可以看出叶子结点值由如下公式计算出来了:
然而我很困惑的是GBDT的实现跟论文中的不太一样,上面那个公式还需要乘以权重才对,可是代码实现的时候却没有写,很奇怪的说。。至此GBDT就结束了。
voting.py
投票器,只有VotingClassifier和VotingRegressor两个接口,都继承于_BaseVoting、_BaseComposition和BaseEnsemble。_BaseComposition就是对BaseEnsemble进行了一些扩展,因为投票器中的预测器是要有名称的,组合成tuple格式,所以底层处理时加了点操作,就让_BaseComposition继承BaseEnsemble来实现。而且这代码一看就不是一拨人写的,例如在tree.py内对lable处理是用的unique,而这里是用LabelEncoder;并行处理各个代码也都不一样。。。
主要方法就是软投票和硬投票两种,软投票就是各预测器输出概率的加权平均值,硬投票就是看哪个类别的数量多,最后输出的类别就是谁。
_hist_gradient_boosting
sklearn在0.21后增加了基于LightGBM的接口HistGradientBoostingClassifier和HistGradientBoostingRegressor。这个接口不稳定,随时会改,但是原理都是固定的,主要更改如下:
-
首先在特征分裂点上使用了integer-valued bins,具体操作代码在binning.py中的_find_binning_thresholds函数。这种机制是先定义最多256个桶(可自定义),选取最多2e5个样本,将特征按分位数分在256个桶中(如特征小于256则按特征数),并记录特征裁剪点。在树结点分裂时就会按照裁剪点分裂,而不会遍历所有特征,会使训练过程大大加速。在构建完integer-valued bins后,X的特征即以完成bin排序了(X的特征现在是记录着对应bin的序号)。
-
sklearn的多分类树是one-to-all类型,所以每次一棵树都是当前类别和其他类别的二分类结构;特征直方图是一个结构体,shape=(n_features,max_bins),记录着每个特征当前类别的bin的梯度和与hessen和,再根据xgboost论文上的公式,计算loss以得到分裂得分。
-
分裂的操作全都在splitting.pyx中,在分裂时使用多线程查找分裂节点左右样本,并切分其样本索引值;
在看过Tree的Cython源码后这个代码就很简单了,写的特别通俗易懂,在此就不细讲了,我估计这个迟早会写成Cython版本来加速。
其他
总结一下GBDT、Xgboost和LightGBM的异同。
GBDT:基学习器是回归树,残差是损失函数一阶导数,不支持自定义损失函数,有shrink,不支持特征采样,特征切分使用预排序后贪婪切分算法,子树分裂有level-wise(深度优先)和leaf-wise(最优值优先)两种,使用全部样本。
Xgboost:基学习器是任意,残差是损失函数二阶泰勒展开,支持自定义损失函数,有shrink,支持特征采样,特征切分使用预排序后分位数算法,子树分裂有level-wise和leaf-wise两种,使用全部样本。
LightGBM:基学习器是回归树,残差是损失函数二阶泰勒展开,不支持自定义损失函数,有shrink,支持特征采样,使用Exclusive Feature Bundling(EFB)进行特征捆绑(合并),特征切分使用特征直方图算法,子树分裂是leaf-wise,使用Gradient-based One-Side Sampling (GOSS)算法进行样本选取。
总结
-
看源码时从接口出发,sklearn的源码普遍都是从fit出发,这样一条龙下来就都看完了,既然选择了看源码就要把底层操作都看了,否则你光看那点接口代码我觉得毫无用处,徒增“自豪感”,源码有用的东西都在底层;
-
在设计底层时,将需要操作的数据变换为内存中的连续内存块,这样通过指针操作会大大加快IO速度,对数据操作尽量以操作其索引为主,操作到最后再由索引读取内存块内的数据所有底层操作只对上层操作提供接口,返回原始记录数据索引,不需要进行过多处理;
-
在编写大型代码结构时,要充分考虑输入的规范性,同时对外接口的容错性要高,不要把麻烦都留给调用者;
-
在规划架构时尽量风格统一,这样在看完一节代码后其他代码都轻松很多,在基类写出可供所有子类调用的接口,这样子类就不用重复写了;
-
在sklearn中底层已进行了许多优化,如Tree通过特征预排序加速特征选择(在底层源码中特征得分计算并未实现XgBoost之类的并行化)、排除常特征列,在bagging中并行化训练基学习器等;
-
细节方面,随机选取特征是Fisher-Yates随机方法,随机样本选择是使用randint或permutation方法;
-
sklearn所有树模型都不支持处理None特征,使用前必须填充缺失值,而字符型特征则无需处理,底层np.unique直接就编码了;