关于正则表达式
一定要看完,收藏就是雪藏。
如果我们要在一段mRNA序列里查找起始密码子“ATG”的位置,在word中使用查找就可以,也可以使用快捷键Ctrl+F,输入ATG就行。这是常规操作,人人都会。
但是如果我们要寻找一段CDS区(蛋白编码序列),他通常是以ATG起始,以TGA,TAG,TAA三个中的任意一个结尾,起始和终止中间出现的字符要是3的倍数(三联密码子)。上述描述的规律无法直接输入,需要用一个通用的具有代表性式子来表示,可以是这样的
ATG([ATCG]{3})+(TGA|TAG|TAA)
现在看不懂很正常,说明还不懂,需要往下看,相信我,看完了你就懂了(我不骗你)。
这种表示匹配模式的式子就是传说中的正则表达式。
正则表达式名字很奇怪,我觉得是被翻译给耽误了,他的英文是regular expression,翻译成通用表达式,或者规则表达式会更加合理。
R语言的正则表达式是让我头疼的,所以我干脆就没有去学。一件事情,只要不是刚需,就有拖延的可能性。后来男神Hadley Wickham 写了一个R包stringr使得流程简化,里面规范了正则表达式相关函数的名称。
但是我一直分不清他们之间的关系,直到有一天我遇到了下面这张小抄。
正则表达式的匹配模式用pattern来表示,他把正则表达式在字符串的功能分为四个方面,分别是
- 查找:Detect pattern,确定这个模式有没有
- 定位:Locate pattern, 返回模式起止位置
- 取回:Extract pattern, 返回模式匹配到的条目
- 替换:Replace pattern,替换匹配的模式,返回替换后的结果
理解了这个小抄后,我一下就进去了,有一种愉快的突破感。
总结一下就是一下几个函数,都是以str_开头:
学习字符串处理的R包stringr
我们以此讲一下这几个函数的使用:
首先创建一个需要练习的字符串,
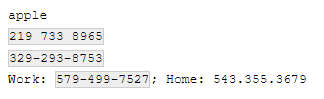
strings <- c(
"apple",
"219 733 8965",
"329-293-8753",
"Work: 579-499-7527; Home: 543.355.3679"
)
我们看到里面有一些电话号码,但是格式不一样,现在我们创建一个可以匹配电话号码的模式
1.正则表达式中的三个括号
在这里先介绍正则表达式中的三个括号
[]
{}
()
中括号[]表示选项,代表内部数据任意选择,比如[ATCG],表示A,T,C,G四个字符随意选择。
如果是数字也是一个意思[1356],表示有1,2,5,6这个四个选项
如果嫌麻烦,可用-连接起始代表范围,
比如[A-Z],代表大写的26个字母,
比如[a-z],代表小写的26个字母,
比如[0-9],代表从0到9的10个数字,
还可以混写
比如[0-9a-zA-Z],代表数字和字母
大括号{}代表重复次数,比如[ATCG]{3}表示从A,T,C,G四个字符中选择3个,那么这时候会产生ATC,ATG,ACG,TCG4种组合,在这里是为了形成三联密码子。
如果是两个数,用逗号隔开,代表范围,[0-9]{4,10} 表示产生4位数到10位数都可以。
如果两个数中的第二个缺失,比如[0-9]{4,},代表4位数及以上。
圆括号()代表成组,跟我们平常理解的意思一样,代表分组。实际上他的用处很多,但是分组,是核心功能。
2.编写第一个正则表达式
这时候,用前面的的知识,已经可以构建一个识别电话号码的模式了。
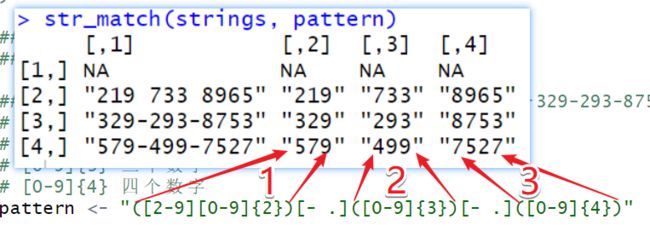
pattern <- "([1-9][0-9]{2})[- .]([0-9]{3})[- .]([0-9]{4})"
首先有三个括号(), 里面的内容分别是
[1-9][0-9]{2}
[0-9]{3}
[0-9]{4}
看一下[1-9][0-9]{2},这表示有三个数字,第一个数字不为零
[0-9]{3}和[0-9]{4}分别表示3位数和四位数
这三个大的内容用[- .]来间隔,分别是-和.号.
这时候回看这个表达式,是不是好很多了
pattern <- "([1-9][0-9]{2})[- .]([0-9]{3})[- .]([0-9]{4})"
3.用str_view函数学习正则表达式
这个函数是用来学习正则表达式的
str_view(strings, pattern)
返回的结果,以图示化展示模式匹配的结果
以下仅仅通过更改函数名称,即可返回不同的结果
1.查找
str_detect,从strings中检测能否和pattern匹配,返回的是逻辑值,有点像grepl
str_detect(strings, pattern)
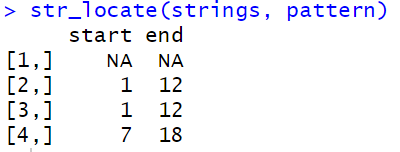
结合str_view的结果,表示,2,3,4确实匹配到了结果。
2.定位
str_locate,从strings中检测能否和pattern匹配,返回匹配的起止位置
str_locate(strings, pattern)
3.取回
这个又分为三个层面, 层层递进
第一,
str_subset返回的是匹配到的原始条目
str_subset(strings, pattern)
第二,
str_extract返回的是匹配到的模式
str_extract(strings, pattern)
第三,
str_match返回的是数据框
第一列是str_extract的数据,后面依次是括号中的内容,模式中有多少个
(),就返回多少列。本例中有三对小括号。
str_match(strings, pattern)
4.替换
str_replace从strings中能够精确匹配pattern的内容,并替换为''XXX-XXX-XXXX''(此处自定义)
str_replace(strings, pattern, "XXX-XXX-XXXX")
现在,我们就多了一个新技能,我最最喜欢str_match。
4.使用正则表达式来提取GEO基因symbol
(这部分内容以前写过,我复制了过来,复制自己已发布的内容,也算是抄袭)
以下这个GEO的样本名称,处理和对照夹杂,如何获取样本名称呢?
在很长一段时间内,我用的是tidyr包中的分列separate,因为作为R语言讲师,我想传授的是自我更新的能力,所以,函数越少越好,虽然我特别推荐R数据科学这本书,认为他拯救了我,但是他里面我也就学习了10个函数,而且我也只教这10个函数。
在这里,我只要不断分和取舍,就能获得想要的列,也就是隐藏着的case和control。
那我们就操作一下:
复制信息粘贴为txt,
读入R语言,并且命名。
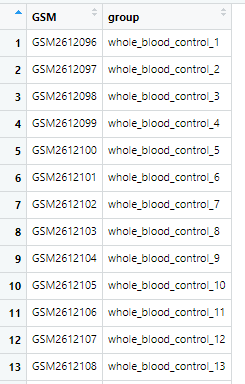
data <- data.table::fread("gsm_group.txt",data.table = F,header = F)
names(data) <- c("GSM","group")
现在数据是这个样子的:
切割两次,第一次以_blood_作为分隔符,一分为二,留取后面的内容,再以 _切割一次,获取前面的内容就可以了
library(dplyr)
library(tidyr)
metadata <- data %>%
separate(group,into = c("drop","group"),sep = "_blood_") %>%
select(-drop) %>%
separate(group,into = c("group","drop"),sep = "_") %>%
select(-drop)
现在我们就获得了想要的数据:
现在我们多了一个选择,正则表达式,而且,这是强项!!
在那之前我们再介绍三个表示匹配次数的符号
?
+
*
问号?表示0次或者是1次,因为这是一个生存或是毁灭的问题
加号+表示1次或者多次,把加号和1联系起来,用医(1)院来记忆
星号*表示0次或者多次,把星号和零联系起来,用零星来记忆
这样我们就得到了六种表示次数的方法
现在我们可以来编写一个正则表达式了
我们用我最喜欢的stringr这个包里面的str_match函数来处理,因为他可以返回括号中的内容,做到精确匹配和返回。
library(stringr)
group <- str_match(data$group,"whole_blood_(.*)_.*")
metadata <- data.frame(GSM=data$GSM,group=group[,2])
下面解释一下这里面涉及到的正则表达式,
whole_blood_(.*)_.*
以"whole_blood_control_33"为例,看看这段表达式的意义:
他表示有这么一段字符串,
- 先出现
whole_blood_ - 再出现一串字符
control - 后面跟着下划线
_ - 最后跟着一串字符结束
33
正则表达式,代表的是我们观察世界的后提取的内在规律,我们观察了group那一列,发现,他可以用一个通用的式子来表达。
正则表达式用点号.代表任何字符,用*号代表出现0次或者多次,那么刚才的特例就可以写成whole_blood_.*_.*
他表示,我要匹配一段字符串,这个字符串由四部分组成,
- 第一部分是,
whole_blood_, - 第二部分是任意个数的字符
.*, - 第三部分是下划线
_, - 第四部分是任意个数的字符
.*,
因为我们需要的是两个下滑线中间的部分,也就是第二部分的内容,所以给他一个括号(),这样str_match函数就会识别这个模式并返回括号()中的内容。此时模式就写出了这样whole_blood_(.*)_.*。
你看,结果是一模一样的,而且十分高效。
再比如,GEO中有的gene symbol是小括号内的
正则表达式处理起来,也是十分顺手,具体看这个
正则表达式是我们认识世界的哲学
本篇帖子,也算是对那个帖子的填坑。
5.匹配编码区域CDS
这个就是我们帖子一开始的那个需求,我们来回忆一下:
如果我们要寻找一段CDS区(蛋白编码序列),他通常是以
ATG起始,以TGA,TAG,TAA三个中的任意一个结尾,起始和终止中间出现的字符要是3的倍数(三联密码子)。
上面这段话,我们就总结了了CDS的规律,正则表达式可以展示出这个规律,可以是这样的
ATG([ATCG]{3})+(TGA|TAG|TAA)
解释如下:
首先是以起始密码子ATG开头,
接着出现三联密码子,用[ATCG]{3}, 出现的次数是1次或者多次,所以整体给个加号+
那现在就是这个样子的
ATG([ATCG]{3})+
接着要出现终止密码子,有三种,分别是TGA,````TGA,TAA,正则表达式中竖线|表示或的意思,那么(TGA|TAG|TAA)```就代表,三个终止密码子任意选择一个。
最终CDS区就表示成这个样子啦
ATG([ATCG]{3})+(TGA|TAG|TAA)
6.转义
这个有点特别,有没有想过,如果一个字符串出现以下的符号,该怎么匹配
'
*
?
+
|
这些字符我们都已经碰到过,他们比较特别,因为他们除了字符串本身外,还有其他意义,称为元字符。正常情况下出现的时候都是特殊意义,如果想要他代表字符本身的意思,比如点号.就代表点号,就需要给他转义,转义的符号是斜杠\,那么点号就应该写作\.。
但是,说话就怕但是,这里的斜杠\也属于元字符,需要另外一个斜杠来转义,那么在R语言中,如果想要表示匹配点号本身,就应该是这个样子的\\.,也就是说R语言里面的转义是两个斜杠\\。
那么如果特殊一点我要匹配一对括号怎么办?括号也是元字符,那就得转义\\(\\)
如果要匹配两个//,那就应该写成\\/\\/,十分有趣吧?
举个例子哈,现在有个能裂解字符串的函数,也是来自于stringr这个包,叫做str_split
我们用点号.来裂解一个字符串a.b.c
str_split("a.b.c",pattern = ".")
这种情况下结果是这样的,显然不对
[[1]]
[1] "" "" "" "" "" ""
点号应该用转义符号去转义
str_split("a.b.c",pattern = "\\.")
此时结果就正常了。
[[1]]
[1] "a" "b" "c"
那么stringr中还有哪些比较有用的函数呢?其实不多,可以参考这个帖子自行学习。
https://cran.r-project.org/web/packages/stringr/vignettes/stringr.html
这样借助stringr这个包,我们就讲完了R语言中的正则表达式,但是实际上,这只是入门。我们题目写的很夸张,30分钟的帖子写了13年。并不是指的是我现在这个帖子,我说的是这个。
这个帖子写的真好,作者的名字叫陆超,软件开发者,自由职业者,父亲。最后的阅读原文可以直接跳转过去,
从他的更新记录我们可以看到,第一版是2006年
这都2019年了,还在更新,十分不容易,我还给他赞赏了一下。
我在想,13年后,我40几岁的时候,大概率还活跃在生信领域,继续更新果子学生信公众号。