matlab自带SVM算法例子(附函数详解)
一、程序
SVM理论的学习,见文章:http://blog.csdn.net/ckzhb/article/details/68941695
load fisheriris %fisheriris 是一个数据集。
X = [meas(:,1), meas(:,2)]; %提取前两个特征。
Y = nominal(ismember(species,'setosa')); %只能识别两类,获取Setosa类数据在species中的标记,Y中存储的相应位置的出现或不出现(True or False)。
P = cvpartition(Y,'Holdout',0.20); %将Y中20%作为测试数据,其余为训练数据。
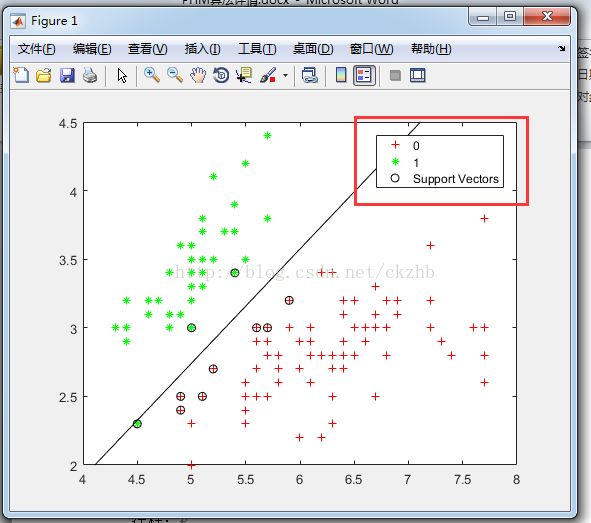
svmStruct =svmtrain(X(P.training,:),Y(P.training),'showplot',true);
C =svmclassify(svmStruct,X(P.test,:),'showplot',true);
errRate = sum(Y(P.test)~=C)/P.TestSize %统计分类错误率
conMat = confusionmat(Y(P.test),C) % t计算混淆矩阵
注:(1)matlab中关于SVM的函数有:svmtrain和svmclassify。
(2)适用于两类问题。多类问题可以参考论文文献。
二、详解
(1)ismember():
[tf index]=ismember(a,b); %判断a中的元素有没有在b中出现。
tf返回一个和a同样大小的矩阵,a的元素在b中出现,tf上的相应位置元素值为1,没有出现则为0;index也是返回一个和a同样大小的矩阵,其相应位置的值为a相应位置的元素在b中出现的第一次标号,若某元素在b中出现多次,则记录的是第一次出现的标号,若该位置上的a的元素没有在b中出现,则返回0.
(2)nominal():
具体见帮助文档。实际上,这里可以不用的。
比较:
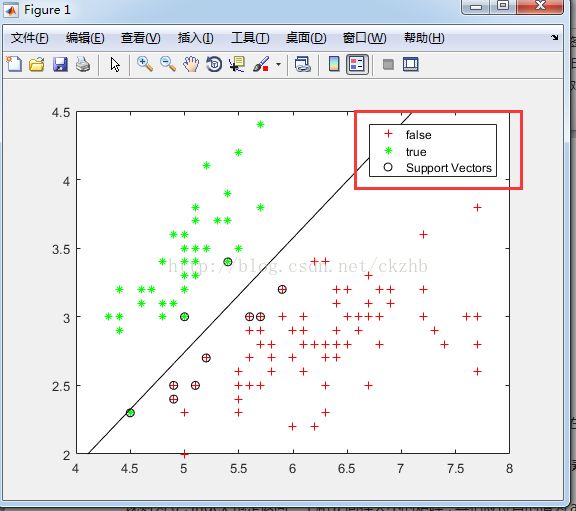
Y = ismember(species,'setosa');
svmStruct =svmtrain(X(P.training,:),Y(P.training),'showplot',1);
与
Y = nominal(ismember(species,'setosa'));
svmStruct =svmtrain(X(P.training,:),Y(P.training),'showplot',true);
结果:
两者分类识别对象不同,一个是“1”,另一个是“true”,最终影响的只是plot中的标签。
(3) C = cvpartition(N,'HoldOut',P):
数据分类,取N乘P为测试集,其余为训练集,进行交叉验证。
所谓交叉验证,即例如10折交叉验证(10-fold cross validation),将数据集分成十份,轮流将其中9份做训练1份做验证,10次的结果的均值作为对算法精度的估计。
Holdout 验证并非一种交叉验证,因为数据并没有交叉使用。 随机从最初的样本中选出部分,形成交叉验证数据,而剩余的就当做训练数据。
cvpartition properties:
TestSize:测试集的大小。这里是30。(150*20%=30)
cvpartition methods:
test:交叉验证分区的测试集。返回一个逻辑向量。
training:交叉验证分区的训练集。返回一个逻辑向量。
详细:P.training是150x1 logical,而且P.test也是150x1logical,两者逻辑相反。即P.training中有120行是逻辑1,30个是逻辑0。P.test与之相反。
则X(P.training,:)——将X中选出的训练集挑选出来。120行2列。
(4)SVMSTRUCT = svmtrain(TRAINING, Y,'PARAM1',val1, 'PARAM2',val2, ...)
TRAINING:预测数据。行表示数据个数,列表示特征的个数。
Y:针对TRAINING的数据的类别。即已知的类别。
TRAINING和Y的行数必须一致,每一行代表一组数据及其结果。(这与matlab中BP算法程序正好相反)。
'PARAM1',val1, 'PARAM2',val2, ... :具体可见帮助文档。
这里使用参数'showplot':A logical value specifying whether or not to show a plot. When thevalue is true, svmtrain creates a plot of the grouped data and the separatingline for the classifier, when using data with 2 features (columns). Default isfalse.
注意:The display option can only plot 2D training data.所以前面X只提取两个特征。
(5)svmclassify(SVMSTRUCT, TEST)
C =svmclassify(svmStruct,X(P.test,:),'showplot',true);
使用之前的训练数据训练出来的支持向量机,对测试数据进行分类。要求TEST的列数与前面训练支持向量机的数据的列数一致。
(6)~= 是 “不等于”的意思。
如a ~= b 判断a和b是否不等,若不等,值为1。
Y(P.test)表示测试集的实际分类结果;C表示测试集经支持向量机分类后的结果。
(7)混淆矩阵
它是一种特定的矩阵用来呈现算法性能的可视化效果,通常是监督学习(非监督学习,通常用匹配矩阵:matching matrix)。其每一列代表预测值,每一行代表的是实际的类别。
混淆即一个class被预测成另一个class,对两类问题,显然是一个2X2矩阵。