Pathway enrichment analysis and visualization of omics data using g:Profiler, GSEA, Cytoscape and EnrichmentMap 使用g:Profiler,GSEA,Cytoscape和EnrichmentMap对组学数据进行通路富集分析和可视化 发表在nature protocols。在生信星球公众号聊天窗口回复“121”可获取原文pdf。对文章进行了简单理解和翻译,由于我是跨专业,没有什么背景知识积累,有不准确的地方欢迎批评指正。
摘要
通路富集分析有助于研究人员获得从基因组规模(组学)实验产生的gene list的机制洞察。该方法鉴定的gene list中富含的生物通路比偶然预期的更多。我们解释了通路富集分析的程序,并提供了一个实用的逐步指导,以帮助解释由RNA-seq和基因组测序实验产生的gene list。该方案包括三个主要步骤:从组学数据定义gene list,确定统计学上富集的通路,以及结果的可视化和解释。我们描述了如何将该方案与差异表达基因和突变癌基因的已发表实例一起使用; 但是,这些原则可以应用于各种类型的组学数据。该流程描述了创新型可视化技术,提供了全面的背景和故障排除指南,并使用免费提供和经常更新的软件,包括g:Profiler,Gene Set Enrichment Analysis(GSEA),Cytoscape和EnrichmentMap。完整的流程可在约4.5小时内完成,设计用于没有事先生物信息学培训的生物学家。
Introduction
现在,生物样品中DNA,RNA和蛋白质的全面定量1⃣已成为常规。由此产生的数据是呈指数式增长,他们的分析有助于研究人员发现新的生物学功能,基因型-表型关系和疾病机理1,2。然而,对这些数据的分析和解释是许多研究人员面临的主要挑战。分析经常导致需要不切实际的大量手工文献搜索解释的长gene list。解决该问题的标准方法是通路富集分析,其将大gene list概括为更容易解释的通路的较小列表。通过几个常见的统计检验,考虑实验中检测到的基因数量,相对排名以及注释到通路的基因数量,对实验gene list中相对于偶然预期的过度表达进行统计检验。例如,含有40%细胞周期基因的实验数据令人惊讶地富集,因为只有8%的人类蛋白质编码基因参与了这一过程。
在最近的一个例子中,我们使用通路富集分析来帮助识别多梳抑制复合物(PRC2)的组蛋白和DNA甲基化作为室管膜瘤的第一个合理治疗靶点,室管膜瘤是最常见的儿童脑癌之一3。该通路可通过可用的药物如5-氮杂胞苷来靶向,其在患有终末病的患者中以富有同情心的方式使用并且停止快速转移性肿瘤生长3。在另一个例子中,我们分析了自闭症罕见的拷贝数变异(CNV的),并确定受基因缺失影响的重要通路,而通过对单个基因或基因座的病例对照关联试验,几乎没有发现显著的影响4,5。这些实例说明了使用通路富集分析可以实现的生物学机制的有用见解。
Development of the protocol
该流程涵盖了源自基因组规模(组学)技术的大gene list的通路富集分析。该流程适用于对解释其组学数据感兴趣的实验生物学家。它只需要一个学习和使用“单击”计算机软件的能力,尽管高级用户可以从我们提供的补充流程自动分析脚本中获益1 - 4。我们分析先前公布的人基因的表达和体细胞突变的数据作为实例6,7,8; 然而,我们的概念框架适用于分析来自大规模数据的任何生物的基因或生物分子列表,包括蛋白质组学,基因组学,表观基因组学和基因调控研究。我们广泛使用的通路富集分析了许多项目,并已评估了许多可用的工具9,10,11,12。我们在这里介绍的软件包因其易用性,免费访问,高级功能,大量文档和最新数据库而被选中,它们是我们在研究中每天使用的软件包,并向合作者和学生推荐。此外,我们还向这些工具的开发人员提供了反馈,使他们能够实现我们在已发布分析中所需的功能。这些工具是g:Profiler 13,GSEA 14,Cytoscape 15和EnrichmentMap 16,所有这些都可以在线免费获得:
g:Profiler(https://biit.cs.ut.ee/gprofiler/)
GSEA(http://software.broadinstitute.org/gsea/)
Cytoscape(http://www.cytoscape.org/)
EnrichmentMap(http://www.baderlab.org/Software/EnrichmentMap)
Overview of the procedure
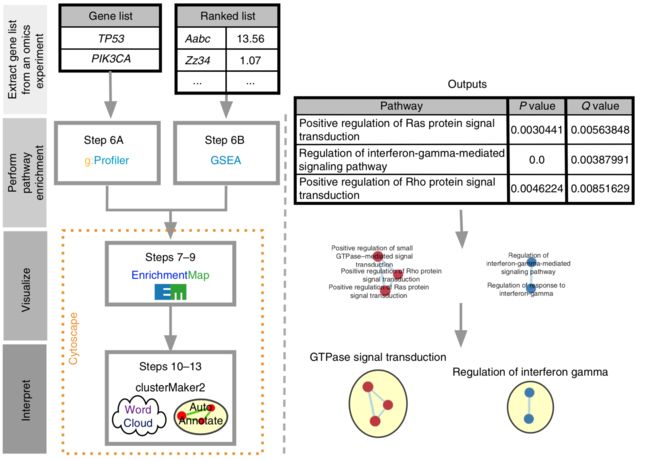
本节概述了通路富集分析的主要阶段。下面的过程中提供了详细的逐步流程。通路富集分析涉及三个主要阶段(图1 ;参见box1的基本定义)。
使用组学数据定义感兴趣的gene list。组学实验在实验环境中全面测量基因的活性。考虑到实验设计,得到的原始数据集通常需要计算处理,例如归一化和评分,以识别感兴趣的基因。例如,可以从RNA-seq数据17导出两组样品之间差异表达的gene list。从其它类型的组学实验,如基因表达芯片得到的gene list18,定量蛋白质组学19,20,种系和体细胞的基因组测序21,22,23和总体DNA甲基化测定法24,25,可以在该流程中使用; 但是,每种类型的数据都可能需要特定的预处理步骤(参见“与替代方法的比较”部分)。
通路丰富分析。统计方法用于鉴定第1阶段gene list中富集的通路,相对于偶然预期的通路。检验给定数据库中的所有通路以在gene list中富集(参见box2以获得通路数据库列表)。可以使用几种已建立的通路富集分析方法,并且选择使用哪种方法取决于gene list的类型(参见“对替代方法的比较”部分)。
通路富集分析结果的可视化和解释。在阶段2中可以鉴定许多富集通路,通常包括相同通路的相关版本。可视化有助于确定主要的生物学主题及其关系,以进行深入研究和实验评估。
图1:流程概述。
fig1|来自不同组学数据的gene list经历通路富集分析,使用g:Profiler或GSEA,以鉴定在实验中富集的通路。使用其EnrichmentMap,AutoAnnotate,WordCloud和clusterMaker2应用程序在Cytoscape中可视化和解释通路富集分析结果。流程概述显示在左侧,从gene list输入开始,每个阶段的示例输出显示在右侧。
Box 1 | Definitions
Pathway。共同实现生物过程的多个基因。
Gene set。一组相关的基因。“通路基因集”包括通路中的所有基因。基因组可以基于基因之间的各种关系,例如细胞定位(例如,核基因)或酶功能(例如,蛋白激酶)。蛋白质相互作用等细节不包括在内。
Gene list of interest。来自组学实验的gene list,其输入到通路富集分析中。
Ranked gene list。在许多组学数据(例如,来自用于基因表达的RNA-seq的数据)中,可以根据一些评分(例如,差异表达水平)对基因进行分级,以提供用于通路富集分析的更多信息。富集在ranked list顶部的基因的通路得分高于如果通路基因随机分散在ranked list中的情况。
Pathway enrichment analysis。一种统计技术,用于鉴定在gene list或排序的目标gene list中显著表示的通路。
Multiple testing correction。可以单独检验数以千计的通路进行富集,这可能导致显著的富集P值单独出现。多重检测校正是一种统计技术,用于校正个体富集检验中的P值以解决该问题并减少假阳性富集的机会(box3)。
Leading-edge gene.。在GSEA分析中在最大ES处或之前的排名中发现的基因子集。这个基因子集通常可以解释被定义为富集的通路。
Box2 通路富集分析资源
Pathway databases
我们列出了一系列大型,开放获取且便于访问的通路数据库,这些数据库为通路富集分析提供了最大价值。数百个通路数据库可用于多种目的82。
Gene set databases
基因本体论(GO)57:GO为生物过程,分子功能和细胞组分提供数千个标准化术语的分层组织,以及基于这些术语的多种物种的策划和预测基因注释。生物学过程GO注释是通路富集分析中最常用的资源。
分子签名数据库(MSigDB)80,81:MSigDB是基于GO基因集的数据库,通路,治疗,个体组学研究,序列基序,染色体位置,致癌性和免疫学表达特征,以及由GSEA团队维护的各种计算机分析的基因组数据库(http://www.msigdb.org)。可以获得相对非冗余的'标志'基因集。该数据可与许多通路富集方法一起使用。
Detailed biochemical pathway databases:这些数据库由管理团队维护,他们手动收集详细的通路信息,包括生化反应,基因调控事件和其他基因相互作用。信息可以导出或转换为基因集格式。
Reactome 58:最活跃的人类通路通用公共数据库(http://www.reactome.org)。
Panther 38:人类信号通路(http://pantherdb.org/pathway)。
NetPath 60:人类信号通路,重点是癌症和免疫学(http://www.netpath.org/)。
HumanCyc 59:人类代谢通路(http://humancyc.org/)。
国家癌症研究所(NCI)通路相互作用数据库(PID):人类癌症相关的信号传导通路; 此数据库不再更新。
KEGG 83:KEGG数据库是最有用的,因为有直观的通路图。它包含多种类型的通路,其中一些不是正常通路,而是与疾病相关的基因集,例如“癌症中的通路”(http://www.genome.jp/kegg/)。由于数据许可限制,KEGG路径的最新GMT文件目前无法免费提供。
Pathway meta-databases
这些数据库收集自多个源通路数据库的详细通路描述。
Pathway Commons 45:从其他通路数据库收集信息并以标准化格式提供。(http://www.pathwaycommons.org)。
WikiPathways 48:共同驱动的通路集合,其中还包括来自其他数据库的通路(http://www.wikipathways.org/)。
Box3 Multiple testing correction
在典型的通路富集分析中使用的重复统计检验将意外产生一些极显著的P值。为了纠正这种情况,多重检验校正方法系统地降低了从一系列检验中得出的每个P值的重要性。在该流程中,g:Profiler和GSEA自动对P值应用多重检验校正。最常用的method是BH-FDR(或只有FDR)34。它基于降压程序,使用未校正的P值阈值和检验次数估计富集通路上的错误富集通路的评分。例如,假设100个通路的P值<0.05时,并且在p值<0.05时FDR为5%,意味着有五个通路富集错误。另一个可选method是经典的Bonferroni多重检验校正,通过将其除以检验次数来调整显著性阈值。实际上,该方法将每个未校正的P值乘以进行的检验的数量,设好显著性临界值(例如,如果已经检验了100个路径,则P值0.001将变为非显著的Q值0.1)。该技术确保选择至少一个错误富集通路的概率低于校正的临界P值。对于差异基因表达和通路富集分析,Bonferroni校正通常被认为过于保守,因为可以容忍一些假阳性结果。重要的是,Bonferroni和BH-FDR都假设检验是独立的,而由于基因重叠和串扰,通路通常不是独立的。因此,BH-FDR对通路分析的估计可能是不准确的,但实际上它们仍然可用于过滤和假设生成,因此是常规使用的。
stage1:使用组学数据定义感兴趣的gene list
基因组规模的实验产生原始数据,必须对其进行处理以获得适合于通路富集分析的基因水平信息(补充流程1和2)。特定的处理步骤适用于特定的组学实验类型,并且可以是标准化的,因此要么可以直接执行,要么就不适用,在这种情况下,数据处理可能需要高级计算技能。标准处理方法可以用于已建立的组学技术,由生成数据的核心设备执行数据处理会是最方便的。
从组学数据中定义gene list有两种主要方式:list或ranked list。某些组学数据自然地产生gene list,例如通过外显子组测序鉴定的肿瘤中的所有体细胞突变基因,或者在蛋白质组学实验中与诱饵相互作用的所有蛋白质。这样的列表可以使用g:Profiler直接输入通路富集分析(step6A)。其他组学数据自然产生排名列表。例如,可以通过全基因组CRISPR筛选中的差异基因表达评分或灵敏度对gene list进行排序。一些通路富集分析方法分析通过特定阈值(例如,FDR调整的P)过滤的分级gene list值<0.05和倍数变化> 2)。替代方法,例如GSEA,旨在分析所有可用基因的ranked list,并且不需要阈值。全基因组ranked list适合使用GSEA输入通路富集分析(step6B)。部分(非全基因组)排序的gene list应使用g:Profiler分析。
例如,我们描述了卵巢癌样本中原始RNA-seq数据的分析,以定义排序gene list7。对reads进行质控和过滤(例如去除低质量碱基)并比对到全基因组范围的参考转录本,以对每个转录本比对到的reads进行计数。reads计数在基因水平上汇总(对每个基因计数)。通常,可获得多个实验条件(两个或更多个,例如治疗和对照)中的每一个的多个生物学重复(三个或更多个)的RNA-seq数据。不同样品对应的每个基因的reads计数进行标准化,以去除样品之间不必要的技术性差异(例如,由于测序lane或每测序获得的总reads数的差异)26,27,28。接下来,检验每个基因的reads数是否在样品分组之间存在差异表达(例如,处理与对照)(RNA-seq和芯片数据分别对应补充流程1和2)。R包如edgeR 29,DESeq 30,LIMMA / VOOM 31,32和Cufflinks 33等,用于RNA-seq数据标准化和差异表达分析。差异基因表达分析结果包括:(i)描述差异表达显著性的 P值; (ii)相关 Q值(又称校正后的 P.值,是对所有基因的多重检验进行校正(例如,通过使用BH-FDR程序34(box3)); (iii)表达变化的效应大小和方向,上调的基因是阳性的,在列表的顶部;下调的基因是阴性的,在列表的底部,通常表示为log-transformed fold-change。然后依据一个或多个值对gene list进行排序(例如-log10 P值乘以log-transformed fold-change),进行通路富集分析研究。
stage 2A:使用g:Profiler的gene list的通路富集分析(step6A)
在g:Profiler和类似的基于网络的工具的默认分析流程35,36,37,38,在固定的目的基因列表中搜索基因显著富集(即过表达)的通路,而不是搜索基因组中的所有基因(step6A)(box4)。使用Fisher精确检验计算通路富集的P值,并应用多重检验校正(box3)。
g:Profiler还包括一个有序的富集检验,它适用于按评分排序的多达几千个基因的列表,而其余的基因组中基因缺乏有意义的排序方式。例如,可以通过cancer driver预测方法6的评分对显著突变的基因进行排序。该分析重复修正的Fisher精确检验,输入递增的较大的基因子列表,并返回每个通路的富集P值最强的子列表39。g:Profiler搜索基因集的集合,包括基因本体论(GO)术语,通路,网络,调节基序和疾病表型的基因集合的集合。可以选择主要类别的基因集来定制搜索。
使用Fisher精确检验或相关检验的通路富集方法需要定义背景基因以进行比较。所有注释的蛋白质编码基因通常用作默认值。如果实验直接测量所有基因的子集,将导致P值的不适当膨胀和假阳性结果。例如,设置自定义背景对于分析来自靶向测序或磷酸化蛋白质组学实验的数据非常重要。好的定制背景会分别包括测序仪panel中所有基因或所有已知的磷蛋白。
stage2B:使用GSEA对ranked gene list进行通路富集分析(step6B)
ranked gene list的通路富集分析使用GSEA软件14(step6B)(box4)。GSEA是一种无阈值方法,可根据其差异表达排序或其他评分对所有基因进行分析,无需事先进行基因过滤。GSEA特别适用于基因组中的所有或大多数基因(例如,RNA-seq数据)可排序的情况。但当只有小部分基因可排序时(例如,在鉴定显著突变的癌症基因的实验中)(stage2A;步骤6A),GSEA不适用。
GSEA主要搜索其基因在ranked gene list的顶部或底部富集的通路。例如,如果最顶端的差异表达基因参与细胞周期,这表明细胞周期通路在实验中受到调节。相反,如果细胞周期基因在整个ranked list中随机分散,则细胞周期通路可能没有受到明显的调节。为了计算通路的富集评分(ES),GSEA逐个检查ranked list的顶部到底部的基因,如果基因是通路的一部分则增加ES,否则降低ES。对这些计算的总和值进行加权,以便放大顶部(和底部)排序基因的富集,而没有放大排序在中间的基因的富集。ES评分是计算总和的最大值,再相对于通路大小标准化,得出归一化的富集评分(NES),反映了list中通路的富集。正NES值和负NES值分别表示列表顶部和底部的富集情况。最后,基于排序计算并校正P值以进行多重检验以产生FDR Q值,其范围从0(高度显著)到1(不显著)(box3)。从ranked gene list的底部开始进行相同的分析,以鉴定在列表底部富集的通路。使用FDR Q值的阈值(例如,Q <0.05)选择所得到的通路并使用NES进行排序。此外,GSEA分析的“leading edge”部分确定了对检测到的通路富集信号贡献最大的基因。
GSEA有两种确定ES的统计显著性(P值)的方法(基因集排序和表型排序)。基因集排序检验需要ranked list,并且GSEA将观察到的通路ES与重复分析随机取样获得的同样大小的基因集(例如1,000次)获得的评分分布进行比较。表型排序检验需要所有样品的表达数据(例如,生物学重复)、“表型”的样品分组(例如,病例与对照;肿瘤与正常样品)。将获得到的通路的ES与通过在所有样品随机打乱并重复分析(例如1,000次)获得的评分分布进行比较,包括ranked gene list和获得的通路的ES。对于具有有限变异性和生物学重复的研究(即每种条件下2至5次),推荐基因集排序。在这种情况下,差异基因表达值应该在GSEA之外计算,使用包括方差稳定的方法(例如edgeR),DESeq和LIMMA / VOOM)并在通路分析之前导入GSEA软件。表型排序应使用大量重复(例如,每种条件至少10次)。表型排序方法的主要优点在于,与基因集排序方法相比,它在排序期间维持具有生物学上重要的基因相关性的基因集的结构。该流程仅涵盖基因集排序,因为它适用于通路富集分析的最常见用法。表型排序需要消耗大量计算资源,并且对于当前版本的GSEA,需要定制编程以分别计算数千个表型随机化的ES和差异表达统计。对于高级用户,我们为此程序提供补充流程(补充流程4).
默认情况下,GSEA桌面软件搜索MSigDB基因集数据库,其中包括通路,已发表的基因特征,microRNA靶基因和其他基因集类型(box2)。用户还可以提供自定义数据库作为基于文本的GMT(Gene Matrix Transposed)文件,其中每一行定义一个通路,包括名称,标识符和它包含的gene list。GMT文件中的基因ID必须与输入gene list中的基因ID相对应。
stage3:通路富集分析结果的可视化和解释(step7-13)
通路信息本质上是冗余的,因为基因通常参与多种通路,且数据库包括具有许多共有基因的一般和特定通路(例如,'细胞周期'和'细胞周期的M期')来分级地编组通路。因此,通路富集分析通常突出显示相同通路的几个版本。将冗余通路折叠成单一的生物学主题可以简化注释。我们建议使用可视化方法(如EnrichmentMap,ClueGO等)来解决此类冗余问题。 “富集图”是表示丰富路径之间重叠的网络可视化(图1),而“EnrichmentMap”是指创建可视化的Cytoscape应用程序。如果通路共享许多基因,则通路显示为与线连接的节点。节点根据ES着色,边线根据连接通路共享的基因数量确定大小。网络布局和聚类算法自动将类似的通路按照主要的生物学主题分组。EnrichmentMap软件将包含通路富集分析结果的文本文件和包含原始富集分析中使用的通路基因集的另一文本文件作为输入。通路ES(过滤节点)和通路(过滤边缘)之间的连接可以交互式探索(step9A(xii和xiii)和9B(xiii和xiv))。多个富集分析结果可以在单个富集图中同时可视化,在这种情况下,每个富集在节点上使用不同的颜色。如果任选加载基因表达数据,点击通路节点将显示该通路中所有基因的基因表达热图。
EnrichmentMaps有助于识别目的通路和主题。首先应确定主题,以帮助验证通路富集分析结果(阳性对照)。例如,分析癌症基因组数据集,可能会鉴定出与生长相关的通路和癌症的其他标志物。其次,将先前未与实验环境联系起来的通路作为潜在的发现,进行更深入的评价。应首先研究具有最大ES的通路和主题,然后逐渐减弱信号(step12)。第三,更详细地检查感兴趣的通路,检查通路内的基因(例如,表达热图和GSEA前沿基因)。此外,如果图标可用,基因表达值可以用PathVisio等工具从 Pathway Commons,Reactome ,KEGG 47或WikiPathways 等数据库添加到通路图上。如果该图不可用,可以使用STRING或GeneMANIA等工具与Cytoscape 一起定义通路基因之间的相互作用网络,用于添加表达值。这有助于可视化鉴定在实验中改变最多(例如,差异表达)的通路组分(例如,单基因或整个信号级联)。此外,可以用EnrichmentMap后分析工具整合miRNA 52或转录因子53的基因集,搜索富集通路的主要调节因子。最后,可以发表通路富集分析结果以支持科学结论(例如,两种癌症亚型的功能差异),或用于假设生成或实验计划以支持新通路的鉴定。http://www.pathwaycommons.org/guide/提供了更多通路丰富分析实例和核心概念的更深入解释。
优点和局限
与单基因,转录产物或蛋白质的分析相比,组学数据的通路富集分析具有几个优点。首先,它以两种方式提高统计效力:(i)它汇总了给定细胞机制中涉及的所有基因和基因组区域的突变计数,提供了更多的计数,这使统计分析更可靠; (ii)它将数万个基因或数百万个基因组区域(例如SNP)降维到少数“系统”或“通路”,从而降低了多重假设检验的成本。其次,结果通常更容易解释,因为分析是在熟悉的概念水平(如“细胞周期”)上描述的。第三,该方法可以帮助确定潜在的因果机制和药物靶向。第四,从相关但不同的数据中获得的结果更具可比性,因为结果被映射到较小的共享特征空间(即,有限数量的通路)。第五,该方法有助于整合各种数据类型,例如基因组学,转录组学和蛋白质组学,它们都可以映射到相同的通路。因此,将疾病数据与已知机制结合,会更有统计方面的说服力。
在解释通路富集分析结果时,通常需要考虑以下限制,包括本流程涵盖的那些。其他限制的适用情况取决于具体的组学数据类型(请参阅“应用于各种组学数据”部分)。指定和可选的通路富集分析方法的优点和缺点在“与可选方法的比较”部分中给出。
- 富集分析对于多个基因具有强生物信号(例如,差异表达)的通路更有效。例如,在转录组学实验中,假设细胞经过进化,仅在需要时表达通路,而且通路激活或失活的状态受通路中基因的共同作用调节。活性仅由少数基因控制或不受基因表达控制(例如,通过翻译后调节)的通路不会被富集。一些通路分析方法通过使用激活和抑制基因相互作用来构建通路活性的定量模型来解决这个问题,所述通路活性包括未差异表达但仍起到重要调节作用的基因。但是,但是,这些方法需要用到详细生化和调节基因相互作用的途径模型,而模型是由针对性实验得到的,因此数量不多。
- 通路边界往往是任意的,给定的通路包含哪些基因,不同的数据库给出的结果不一致。使用多个数据库分析多个通路定义,在解释实验数据时,有的数据库表现更好。
- 一些通路富集方法(例如基于Fisher精确检验的方法)在统计学上更可能将较大(lager)的通路鉴定为显著的。用户可以通过选择分析中考虑的基因组大小的上限来解决这种限制。
- 在gene list中排名很高的多功能基因可能导致许多不同通路的富集,其中一些与实验无关54。排除这些基因后重复分析可揭示其富集过度依赖于其存在的通路或确认通路富集的稳定性。
- 根据通路数据库分析的富集结果偏向于众所周知的通路。事实上,通路富集分析忽略了没有通路注释的基因,有时被称为“基因组的暗物质”,这些基因应该分开研究。例如,非编码RNA基因目前缺乏系统注释,不能直接用于通路富集分析。
- 大多数富集分析方法对基因和通路之间的统计独立性做出了不符合实际情况的假设。某些基因可能总是共表达(例如,蛋白质复合物中的基因),某些通路包含共同的基因。因此,在检验之间假设统计独立性的标准FDR可能比理想的保守。但还是应该用它校正多重检验和富集通路排序,用于探索性分析和假设生成。自定义排序检验可以更好地评估错漏情况。(参见“与替代方法的比较”部分)。
实验设计
通路富集分析从仔细的实验设计中获益。否则,分析可能会揭示由实验偏差或其他混杂因素引起的明显有意义的结果。本节介绍在执行此流程之前必须考虑的一系列实验方面。
实验条件
必须明确实验条件,以便观察到的主要变化是实验者想要监测的反应并且与感兴趣的生物学问题相关(例如,肿瘤与正常,治疗与未治疗,四种疾病亚型的比较,时间系列)。
重复次数
生物学重复是从不同生物或细胞系获得的独立处理样品,这些样品是测量样品间变异性所需的,并计算统计学显著性(P值)。缺乏复制(即,每组一个样本)将不允许稳健地估计信号的重要性。复制不足可能导致数据中缺乏信号(例如,没有显著差异表达的基因)。样品组的变化越大,精确测量信号所需的生物学重复越多。对于具有较低可变性的系统(即,在受控实验室条件下具有相同遗传背景的模式生物,或来自相同克隆的稳定细胞系),对于具有方差稳定归一化的差异分析,推荐每种条件至少三至四个生物重复。方差稳定化使用全局统计模型来“稳定”基因方差估计,以减少由于很少重复而导致的不准确性。对于具有较高变异性的实验(例如,肿瘤样品),需要更多的重复; 理想情况下,试验性实验接着进行正式的统计功效计算应使用 55(有时称为灵敏度检验)来确定鉴定差异表达基因或富集通路的信号所需的最小重复数。对于成熟的实验技术(例如RNA-seq),通常不需要包括相同样品的重复实验的技术重复,其具有低技术可变性,但可以有助于新技术。
混淆因素
应该避免或者至少在不同条件下平衡与实验问题无关的因素的差异,以便诸如广义线性模型之类的统计技术可以校正每个因素。常见因素包括测序批次,核酸提取方案,受试者年龄和许多其他因素。否则,可能无法准确地将来自实验响应的实验信号与混杂因素分开。提前了解重要因素有助于正确的实验设计。统计探索性分析,如聚类或主成分分析(PCA)可以帮助识别未知因素。例如,期望案例和控件分别进行集群,而不是通过处理批处理。
离群值
由于主要的实验或技术问题,例如污染或样品混淆,异常值样品可能与其他样品有很大不同。或者,它们可能呈现极端的生物学特征,例如具有异常侵袭性表型的肿瘤样品。使用诸如PCA或聚类的统计技术可以无偏见地识别异常值样本。应在有和没有异常值的情况下进行通路富集分析,以确保稳健的结果。系统地去除异常值可能是合理的,以减少实验中的可变性。
实验灵敏度
可以调整一些实验方法或多或少地敏感。例如,RNA-seq实验中的读数数量影响下游分析。为了在具有适度变异性的生物系统中量化基因表达并检验具有方差稳定性的差异表达,需要至少三到五次重复和1000万次映射读数56。需要基本上更大的测序深度,例如50-100百万个定位读数,以研究剪接异构体,检测表达不良的基因或具有复杂细胞混合物的样品,例如手术切除标本。
通路基因组数据库的选择
我们建议仅首先搜索通路基因集的富集,因为这些捕获了易于解释的熟悉的正常细胞过程。来自Reactome 58,Panther 38,HumanCyc 59和NetPath 60的 GO 57生物过程术语和手动策划的分子通路是人类通路的良好资源(box2)。GO生物过程注释包括手动策划和电子推断源的混合。
通过证据代码过滤GO通路
GO中的大部分基因注释源于自动数据分析,未经人类策展人验证。这些证据代码“从电子注释中推断出来”(IEA)。早期的文献警告不要分析和解释IEA标注的注释61,而最近的研究表明,这些注释通常与人类策展人指定的注释一样可靠62。对于具有许多手动策划注释的人类和常见模型生物的数据的高可信度分析,我们通常建议在有和没有过滤IEA注释的情况下比较分析的版本以验证稳健性。然而,IEA注释构成了研究较少的物种中的大部分信息,在这些情况下应默认使用。删除IEA编码的注释可能会使分析偏向于经过充分研究的生物过程。
使用非通路基因组
不同类型的基因集有助于回答各种问题。例如,对应于微小RNA和转录因子的目标非通路基因组可以用来发现重要调节52,53。然而,同时分析所有可用类型的基因集降低了数据的可解释性。它也可能导致假阴性,因为增加的传导检验数量增加了多重检验校正的效果,并降低了单个通路的多重检验调整显著性。因此,我们建议分别进行非通路和通路基因组的分析。
基因集大小考虑因素
排除许多小通路通常是有益的,因为它们是冗余的,具有较大的通路和复杂的解释,并且它们的丰度使得多重检验校正更加严格。也应该排除大的通路,因为它们过于笼统(例如,“代谢”),它们不会有助于结果的可解释性,并且当使用某些统计富集方法(例如,Fisher精确检验)时,它们的统计显著性可以被夸大。为了分析人类基因表达数据,我们经常建议排除具有<10-15个基因和> 200-500个基因的通路基因组,尽管在文献中可以找到200-2,000个基因的上限。然而,对于可能具有不同基因集大小分布的非人生物和其他类型的基因集,可能需要包括更大的集合。过滤通路取决于实验背景,因为不同的生物学领域在通路数据库中具有可变的覆盖范围。通过检查预期与实验相关的几种感兴趣通路的大小,可以确定通路大小的下限和上限。
使用更新的通路基因集的重要性
通路富集分析取决于分析中使用的基因集和数据库,许多最近使用通路富集分析的研究受到过时资源的强烈影响11。为了提高研究的可重复性和透明度,研究人员应在出版物中报告使用的通路富集分析软件和基因集数据库的分析日期和版本,以及所有分析参数。除了富集图谱,作者还应考虑将他们研究的gene list和完整的富集通路表添加为补充信息。
选择基因ID
基因与许多不同的数据库标识符(ID)相关联。我们建议使用明确,唯一且稳定的ID,因为一些ID会随着时间的推移而过时。对于人类基因,我们建议使用Entrez Gene数据库ID(例如,4193对应于MDM2)或基因符号(MDM2是HUGO基因命名委员会推荐的官方符号)。随着基因符号随时间的变化,我们建议保留基因符号和Entrez Gene ID。g:Profiler和相关的g:转换工具支持将多种ID类型自动转换为标准ID。
意外的通路结果和实验设计
通路分析中揭示的意外生物学主题可能表明实验设计,数据生成或分析存在问题。例如,细胞凋亡通路的富集可能表明实验方案存在问题,导致样品制备过程中细胞死亡增加。在这些情况下,在进一步的数据解释之前,应仔细审查实验设计和数据生成。
应用于各种组学数据
该流程使用RNA-seq数据7和体细胞突变数据6作为示例,因为经常遇到这些数据类型。然而,我们提出的通路富集分析的一般概念适用于可以产生gene list的许多类型的实验,例如单细胞转录组学,CNV 5,蛋白质组学63,磷酸化蛋白质组学64,DNA甲基化65和代谢组学66。大多数数据类型都需要流程修改,我们在此仅简要讨论。对于某些数据类型,需要专门的计算方法来产生适合于通路富集分析的gene list,而对于其他数据类型,需要专门的通路富集分析技术。必须考虑特定于数据类型和实验方法的问题,包括:
- 对于某些数据类型,建议使用不同的基因标识 我们建议蛋白质的UniProt登录号(例如,MDM2的Q00987)和代谢物的人代谢组数据库ID(例如,ATP表示为HMDB00538)。
- 通过设计的某些类型的组学实验仅捕获基因或蛋白质的子集。为了解决这种有限的覆盖范围,通路富集分析必须定义可在实验中测量的基因的定制背景基因集。例如,磷酸化蛋白质组学实验仅测量具有一个或多个磷酸化位点的蛋白质,因此必须使用编码磷蛋白的基因组作为定制背景基因组。否则,通路富集分析将揭示一般过程(例如激酶信号传导和蛋白质磷酸化)的膨胀P值。
- 来自ChIP-seq实验的短非编码基因组区域(例如转录因子结合位点)的通路富集分析需要额外考虑。必须将基因组区域定位到蛋白质编码基因并校正偏差,例如较长基因中的信号增加。GREAT 67等工具可自动执行这两项任务。
- 跨越多个基因的大基因组间隔(例如,来自全基因组关联,CNV和差异甲基化区域)需要专门的富集检验,例如PLINK CNV基因组负荷检验68或INRICH 69。标准富集检验经常揭示聚集在基因组中的基因,其信号强烈统计膨胀,因为每个基因被错误地计为独立信号。与基因组位置相关的基因类型包括嗅觉受体,组蛋白,主要组织相容性复合物(MHC)成员和同源框转录因子。解决通路中基因的基因组聚类的简单解决方案涉及在富集分析之前从每个功能上均一的基因组簇中仅选择一个代表性基因。
- 对于罕见的遗传变异,病例对照通路“负担”检验是最合适的通路富集分析方法(参见“替代方法比较”部分)。
与替代方法的比较
通路富集分析方法
该流程建议使用g:Profiler和GSEA软件进行通路富集分析。G:探查13,39分析使用Fisher精确检验和有序的gene list使用改进的Fisher检验gene list。它提供了图形Web界面,并通过R和Python编程语言进行访问。该软件经常更新,基因集数据库可以下载为GMT文件(http://biit.cs.ut.ee/gprofiler)。GSEA 14使用基于排序的检验分析排序的gene list。该软件通常作为桌面应用程序运行(http://software.broadinstitute.org/gsea)。存在数百种通路富集分析工具(参见参考文献70)),尽管许多人依赖于过时的通路数据库或缺乏与最常用工具相比的独特功能; 因此,我们不在此处覆盖它们。以下是替代免费通路富集分析软件工具。虽然我们的流程中没有涵盖这些工具,但我们建议根据其易用性,独特功能或高级编程功能进行以下操作。
- Enrichr 37:这是一个基于网络的富集分析工具,用于基于Fisher精确检验的非ranked gene list。它易于使用,具有丰富的交互式报告功能,包括> 100个基因集数据库(称为库),包括多个类别中的> 180,000个基因集。功能类似于本流程中描述的g:Profiler Web服务器的功能。
- 相机71:该R Bioconductor软件包分析gene list并校正基因间相关性,例如基因共表达数据中明显的基因间相关性。该软件作为Bioconductor中的limma软件包的一部分提供(https://bioconductor.org/packages/release/bioc/html/limma.html ;这是一种需要编程专业知识的高级工具;补充流程3)。
- GOseq 72:该R Bioconductor软件包通过校正用户选择的协变量(如基因长度)来分析RNA-seq实验的gene list(https://bioconductor.org/packages/release/bioc/html/goseq.html ;这是一个需要编程专业知识的高级工具)。
- 基因组区域富集注释工具(GREAT)67:与分析gene list的常用方法相比,GREAT分析基因组区域,如DNA结合位点,并将这些区域与附近的基因连接,用于通路富集分析(http://bejerano.stanford.edu / great / public / html /)。请参阅“应用于各种组学数据”部分。
可视化工具
该流程建议使用EnrichmentMap进行通路富集分析可视化以帮助解释。EnrichmentMap 16是一个Cytoscape 15应用程序,可通过通路富集分析显示结果,并通过将通路显示为网络来简化解释,其中重叠通路聚集在一起以识别结果中的主要生物学主题(http://www.baderlab.org/软件/ EnrichmentMap)。另外两种有用的可视化工具是:
- ClueGO 40:此Cytoscape应用程序在概念上类似于EnrichmentMap,并提供基于网络的可视化,以减少通路富集分析的结果冗余。它还包括一个通路富集分析功能,用于使用Fisher精确检验分析GO注释。但是,它目前仅支持GO基因组。
- PathVisio 49:这个桌面应用程序为EnrichmentMap和ClueGO提供了一种互补的可视化方法。PathVisio使用户能够在感兴趣的通路中在基因和蛋白质相互作用的背景下可视地解释组学数据。PathVisio根据用户提供的组学数据(https://www.pathvisio.org)对通路基因进行着色。与EnrichmentMap和ClueGO相比,这是PathVisio的主要优势。
拓扑感知通路分析方法
大多数通路富集分析方法均匀地处理通路中的所有基因并忽略基因相互作用。相比之下,拓扑感知方法明确地模拟基因之间的相互作用。CePa 73,GANPA 74和THINK-Back 75使用物理基因相互作用或共表达网络为每个通路中的每个基因分配权重。权重可以来自网络中基因重要性的度量,例如程度,基因连接数和介于中心性,并且可以整合到传统通路富集分析方法如GSEA中。方法如SPIA 76,Pathway-Express 77和EnrichNet 78为整个通路产生ES,其考虑通路调节相互作用,例如激活和抑制。尽管有用且可能更准确,但与物理相互作用网络和共表达相比,调节和生物化学基因相互作用可用于更少的基因和通路。我们预计这些方法将变得更有用,因为在通路中更多的基因相互作用在详细的分子实验中被表征。然而,从文献中收集和策划高质量和生物化学详细的通路数据目前是复杂且昂贵的。因此,在可预见的未来,本流程中描述的通路富集分析方法可能仍然是最广泛使用的方法。
未来的观点
目前的通路富集分析方法提供了对基因组学实验中活性通路的有用的高水平概述。然而,这些方法考虑仅涉及基因集的简化通路视图。下一代通路分析方法将整合更多的生物通路细节,基于多种样本中测量的多种基因组学数据构建通路模型,并考虑数据中的正负调控关系。例如,用单细胞RNA-seq数据参数化的定性数学建模可能有一天能够准确预测能够治疗研究中的给定疾病的药物组合。