【深度学习 SS】Selective Search原理剖析
项目地址: http://disi.unitn.it/~uijlings/MyHomepage/index.php#page=projects1
这是一篇2013年发表的文章,应该是下面2011年ICCV会议论文的扩展:
Van, de Sande, Koen E A, et al. "Segmentation as selective search for object recognition. " IEEE International Conference on Computer Vision, ICCV 2011, Barcelona, Spain, November 2011:1879-1886.
1 介绍
如何定位一张图像上的目标(比如"牛")? 处理的流程可以是这样的:
第一步: 将图像划分成很多的小区域(regions);
第二步: 判定每个区域是属于"牛"的还是"非牛",将属于"牛"的区域进行合并,就定位到牛了!
解释:
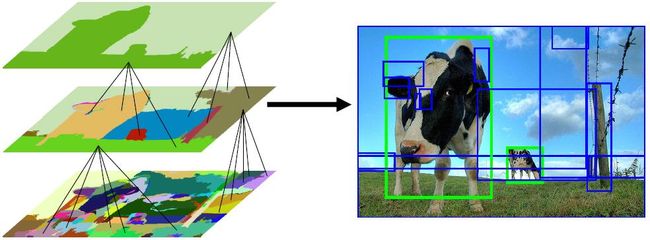
第一步解释: 如何将图像划分成很多的小区域? 划分的方式应该有很多种,比如: 1)等间距划分grid cell,这样划分出来的区域每个区域的大小相同,但是每个区域里面包含的像素分布不均匀,随机性大;同时,不能满足目标多尺度的要求(当然,可以用不同的尺度划分grid cell,这称为Exhaustive Search, 计算复杂度太大)! 2)使用边缘保持超像素划分; 3)使用本文提出的Selective Search(SS)的方法来找到最可能的候选区域;
其实这一步可以看做是对图像的过分割,都是过分割,本文SS方法的过人之处在于预先划分的区域什么大小的都有(满足目标多尺度的要求),而且对过分割的区域还有一个合并的过程(区域的层次聚类),最后剩下的都是那些最可能的候选区域,然后在这些已经过滤了一遍的区域上进行后续的识别等处理,这样的话,将会大大减小候选区域的数目,提供了算法的速度.
下面放一张图说明目标的多尺度:
第二步解释: 第一步中先生成,后合并得到了那些最可能的候选区域,这一步将对每个区域进行判别,也就是判别每个区域到底是"牛"还是"非牛"! 流程的话,无非是在每个区域上提取特征,然后训练一个分类器(Kernel SVM);
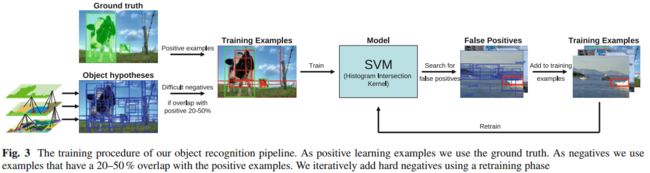
2 训练和测试流程图
2.1 本文模型训练流程图
下面我将按照自己的理解一步一步地对此训练流程图进行讲解: 这个流程图我认为应该分成四个部分:
第一部分: 训练集构造
负样本: 给定一张训练图像 --> 形成原始的过分割区域 --> 使用本文SS方法对区域进行融合,形成一系列最可能的候选区域 --> 计算每个候选区域与真实标记区域GT之间的重合度,如果区域A与GT的重合度在20-50%之间,而且A与其他的任何一个已生成的负样本之间的重合度不大于70%,则A被采纳为负样本,否则丢弃A,继续判别下一个区域;
正样本: 就是那些手工标记的GT区域作为正样本;
(问题1: 会不会负样本很多而正样本很少? 从而出现类不均衡问题)
下图展示了区域合并的过程: 对于此图而言,正样本是两个绿色框框圈出来的区域;负样本为蓝色框框圈出来的区域;正样本是人手工标记的,负样本是SS方法得到的!
第二部分: 提取每个正/负样本(都是一个个不同大小的区域)的特征
第一部分中将正样本区域和负样本区域都提取出来了,现在就需要提取每个区域的特征了.本文主要采用了两种特征: HOG特征 + bag-of-words特征,同时辅助性地增加了SIFT,two colour SIFT,Extended OpponentSIFT,RGB-SIFT这四种特征,这样特征加起来的维度达到了惊人的360,000.
(问题2:每个区域的大小都是不相同的,如何保证提取到的每个区域的特征向量维度相同?)
第三部分: 分类器
第二部分中,每个区域的特征提取出来了,真实类别标签也知道,那这就是一个2分类问题;分类器这里采用了带有Histogram Intersection Kernel的SVM分类器进行分类;这里没有对分类器本身做什么改进,我们可能会质疑一下他这种分类器的选择是否对这种场合是最好的,其他的没什么好讲的.
(问题3:选这种分类器的原因是不是它适用于处理高维度数据?)
第四部分: 反馈
第三部分将分类器训练好了,训练好了就完了吗? NO! 现在流行一种反馈机制,SVM训练完成了,将得到每个训练图像每个候选区域的软分类结果(每个区域都会得到一个属于正样本的概率),一般如果概率大于0.5将被认为是目标,否则被认为是非目标,如果完全分类正确,所有的正样本的SVM输出概率都大于0.5,所有负样本的SVM输出概率都小于0.5,但是最常见的情况是有一部分的负样本的输入概率也是大于0.5的,我们会错误地将这样样本认为是目标,这些样本就称之为"False Positives".
我们这里就是想把这些"False Positives"收集起来,以刚才训练得到的SVM的权值作为其初始权值,对SVM进行二次训练,经过二次训练的SVM的分类准确度一般会有一定的提升;
2.2 测试过程
测试的过程基本和训练过程相同: 首先用SS方法得到测试图像上候选区域 --> 然后提取每个区域的特征向量 --> 送入已训练好的SVM进行软分类 --> 将这些区域按照概率值进行排序 --> 把概率值小于0.5的区域去除 --> 对那些概率值大于0.5的,计算每个区域与比它分数更高的区域之间的重叠程度,如果重叠程度大于30%,则把这个区域也去除了 --> 最后剩下的区域为目标区域.
(问题4:重叠程度如何计算,如果计算A与B之间的重叠程度,分子是A与B的交集,分母是A还是B?)
总结
1. 本文最大的卖点在于它的Selective Search策略,这个策略其实是借助了层次聚类的思想(可以搜索了解一下"层次聚类算法"),将层次聚类的思想应用到区域的合并上面;作者给出了SS的计算过程:
总体思路:假设现在图像上有n个预分割的区域,表示为R={R1, R2, ..., Rn}, 计算每个region与它相邻region(注意是相邻的区域)的相似度,这样会得到一个n*n的相似度矩阵(同一个区域之间和一个区域与不相邻区域之间的相似度可设为NaN),从矩阵中找出最大相似度值对应的两个区域,将这两个区域合二为一,这时候图像上还剩下n-1个区域; 重复上面的过程(只需要计算新的区域与它相邻区域的新相似度,其他的不用重复计算),重复一次,区域的总数目就少1,知道最后所有的区域都合并称为了同一个区域(即此过程进行了n-1次,区域总数目最后变成了1).算法的流程图如下图所示:
2. 除了SS这个卖点之外, 本文还用较大的篇幅讲述了"Diversification Strategies(多样化策略)"这个东西,在我看来,这就是一个模型选择问题,讲不讲都没有多大的关系.我认为,面对一个具体的问题,有很多的超参需要调节.何为超参?我认为超参从小到大应该分成三类:
第一类: 一个既定模型里面可以调节的参数.比如在CNN里面有学习率,卷积核尺寸,卷积maps数目等等参数,面对不同的问题,这些参数的设置可能是不相同的,不同的参数带来不同的结果,因此我们需要对这些参数进行调节;再比如本文SS算法里面的相似度度量,度量方法不止一种,你可能需要面对实际的任务对其进行实验调整.
第二类: 一个既定模型里面可以调节的模块.比如说本文的模型,它采用了SVM分类器,你可以把它换成其他的分类器;提取了HOG等特征,你也可以把它换成深度特征;利用了RGB图像,你也可以把RGB转化到其他的色彩空间进行;采用这种初始区域初始化方法,当然也可以换成其他的,只要最适合你的任务就行.
第三类: 模型方法超参.比如同样是解决目标检测问题,本文的方法算是一种,但是还有千千万万种其他的不同的目标检测算法,从这个角度来说,模型方法整体上可以看做是一种超参,当然,在实际进行中我们可能只专注于自己的算法,对这个超参的调节表现在实验部分对不同方法之间的对比.
原文"3.2 Diversification Strategies"这一节提供了调参的一些思路,分析了不同颜色空间各通道的特点,这有利于我们在面对自己的实际任务时明白要尝试的方向.还有就是给出了SS算法里面相似度度量的几种方法,比如颜色相似度,纹理相似度,尺寸相似度,填充相似度,作者肯定没有对所有的相似度进行穷举,也没有说哪种相似度适合哪种任务,我觉得更重要的还是一种思路的借鉴,提供了一种思路和思考方向,我觉得这篇论文的价值就达到了.
3. 计算速度
相比于以往的对图像上的区域进行穷举的方法,本文SS方法只生产一小部分的最可能的候选区域,这样对后续的处理以及整体计算有效性确实有所提高.但是我认为对每个区域都要提取高达36万维度的特征向量,这可能是本文算法最耗时的地方,如此高纬度的特征向量是否有必要?(给人的感觉就是一股脑把那些典型的手工特征HOG,SIFT不管好坏都提取出来了)
本文难念有一些误解或者不当之处, 敬请留言指教, 谢谢!
参考文献:
[1]Selective Search for Object Recognition: http://blog.csdn.net/charwing/article/details/27180421
[2]Selective Search for Object Recognition解读: http://blog.csdn.net/mao_kun/article/details/50576003
[3]MATLAB源代码SelectiveSearchCodeIJCV.zip: https://pan.baidu.com/s/1bncWrQR
[4]官网:https://ivi.fnwi.uva.nl/isis/publications/bibtexbrowser.php?key=UijlingsIJCV2013&bib=all.bib