- 相关文章:LIDC-IDRI肺结节公开数据集Dicom和XML标注详解
一、数据源
训练数据源为LIDC-IDRI,该数据集由胸部医学图像文件(如CT、X光片)和对应的诊断结果病变标注组成。该数据是由美国国家癌症研究所(National Cancer Institute)发起收集的,目的是为了研究高危人群早期癌症检测。
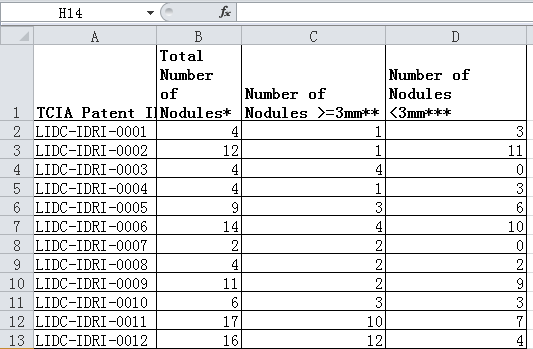
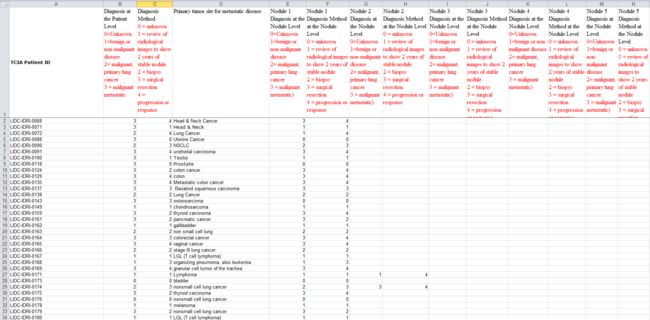
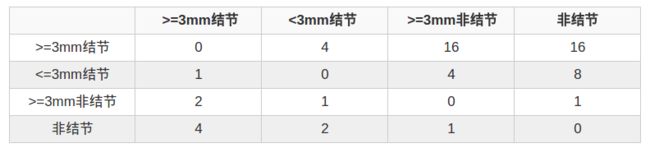
该数据集中,共收录了1018个研究实例。对于每个实例中的图像,都由4位经验丰富的胸部放射科医师进行两阶段的诊断标注。在第一阶段,每位医师分别独立诊断并标注病患位置,其中会标注三中类别:1) >=3mm的结节, 2) <3mm的结节, 3) >=3mm的非结节。在随后的第二阶段中,各位医师都分别独立的复审其他三位医师的标注,并给出自己最终的诊断结果。这样的两阶段标注可以在避免forced consensus的前提下,尽可能完整的标注所有结果。
文件位置: LIDC-IDRI -> lidc-idri nodule counts (6-23-2015).xlsx
文件位置 : LIDC-IDRI -> tcia-diagnosis-data-2012-04-20.xls
二、图像文件格式
1. 图像Dicom格式
图像文件为Dicom格式,是医疗图像的标准格式,其中除了图像像素外,还有一些辅助的元数据如图像类型、图像时间等信息。
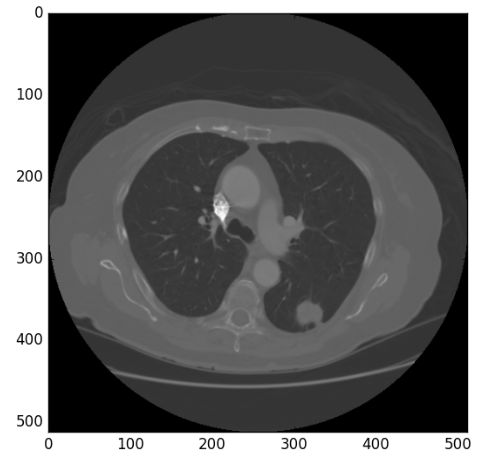
一张CT图像有 512x512 个像素点,在dicom文件中每个像素由2字节表示,所以每张图片约512KB大小。图像中每个像素都是整数,专业名称为 Hounsfield scale 或 CT Number,是描述物质的放射密度的量化值(参考Wikipedia)。下表为常见物质的HU值。
由于图片为单通道,画图渲染出来为黑白图,放射密度越高的位置越亮。
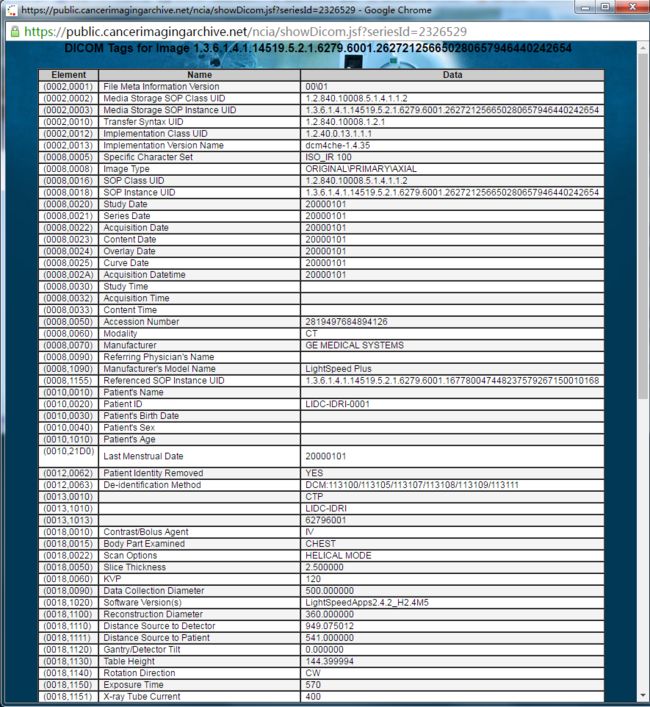
除了像素图以外,元数据中有一些其它主要Tag (参考DICOM的常用Tag分类和说明)
在LIDC-IDRI上面可以直接网页上搜索图像数据信息,通过Dicom里面的tag可以对比上述tag描述,我们在实际过程中只取上述tag使用,其他tag暂时不管:
2. HDF文件格式
在之后的数据处理中可能还会用到hdf格式的数据,下面介绍一下hdf文件格式:
HDF是用于存储和分发科学数据的一种自我描述、多对象文件格式。HDF是由美国国家超级计算应用中心(NCSA)创建的,以满足不同群体的科学家在不同工程项目领域之需要。HDF可以表示出科学数据存储和分布的许多必要条件。HDF被设计为:
- 自述性:对于一个HDF文件里的每一个数据对象,有关于该数据的综合信息(元数据)。在没有任何外部信息的情况下,HDF允许应用程序解释HDF文件的结构和内容。
- 通用性:许多数据类型都可以被嵌入在一个HDF文件里。例如,通过使用合适的HDF数据结构,符号、数字和图形数据可以同时存储在一个HDF文件里。
- 灵活性:HDF允许用户把相关的数据对象组合在一起,放到一个分层结构中,向数据对象添加描述和标签。它还允许用户把科学数据放到多个HDF文件里。
- 扩展性:HDF极易容纳将来新增加的数据模式,容易与其他标准格式兼容。
- 跨平台性:HDF是一个与平台无关的文件格式。HDF文件无需任何转换就可以在不同平台上使用。
除了NCSA在用它,我想还有国家卫星气象中心因为国家卫星气象中心提供了一个中文版的翻译HDF5.0 使用简介 - 国家卫星气象中心!
注:hdf具体介绍详见下述链接:
关于HDF文件的一点概述(HDF4,HDF5)
HDF5 小试——高大上的多对象文件格式
三、诊断标注
诊断标注以XML格式提供,目前已有XML parser (DeepLearning:medical_image/src/data/annotation.py)
- XML文件头部ResponseHeader:
1.7
1152727
2006-06-05
17:08:37
removed
removed
Second unblinded read
removed
1.3.6.1.4.1.14519.5.2.1.6279.6001.340202188094259402036602717327
1.3.6.1.4.1.14519.5.2.1.6279.6001.584233139051825667176600857752
2006-06-05
17:08:37
1 - Reading complete
Merged, reader anonymized, unblinded responses
- XML文件ReadingSession (
标注 ):
3.12
anonymous
16448
-181.0
1.3.6.1.4.1.14519.5.2.1.6279.6001.156388264492529846911221229696
TRUE
61 198
16446
-187.0
1.3.6.1.4.1.14519.5.2.1.6279.6001.270634182491671360557327079123
373 225
- XML文件ReadingSession (
标注 ):
3.12
anonymous
12468
-70.0
1.3.6.1.4.1.14519.5.2.1.6279.6001.350028248790971951374528707491
TRUE
398 252
12453
3
1
6
2
4
1
1
5
1
-250.0
1.3.6.1.4.1.14519.5.2.1.6279.6001.100791367364990316173567441168
TRUE
49 316 50 315 51 314 51 313 51 312 51 311 51 310 51 309 51 308 51 307 50 306 49 305 48 306 48 307 49 308 48 309 48 310 48 311 49 312 48 313 49 314 48 315 49 316
12461
-253.0
1.3.6.1.4.1.14519.5.2.1.6279.6001.150739457477763063347777523734
TRUE
202 398
12471
-199.0
1.3.6.1.4.1.14519.5.2.1.6279.6001.267363953457812811093234965009
372 153
四、数据处理代码分析
- github:zhwhong/lidc_nodule_detection/pylung
1. nodule_structs.py
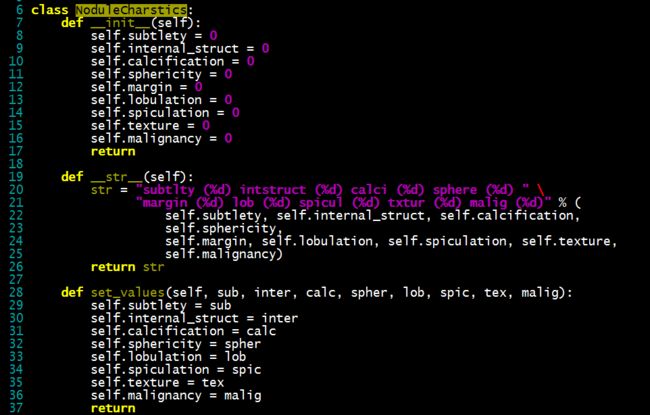
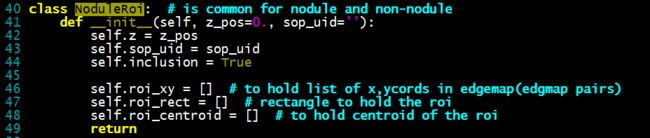
【基本结构】
- NoduleCharstics
- NoduleRoi
- Nodule(所有结节类型的基类)
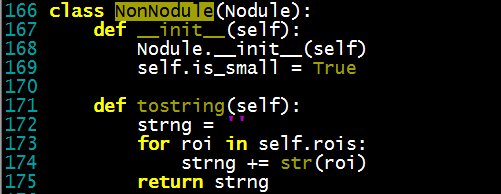
【结节分类 -> 普通结节,小结节,非结节】
- NormalNodule
- SmallNodule
- NonNodule
【结节标注】
- RadAnnotation
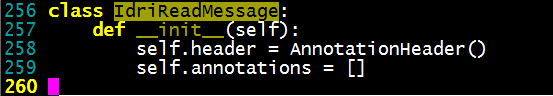
【结节头】
- AnnotationHeader
【读取XML信息】
- IdriReadMessage
2. annotation.py(部分)
- 【分析XML】
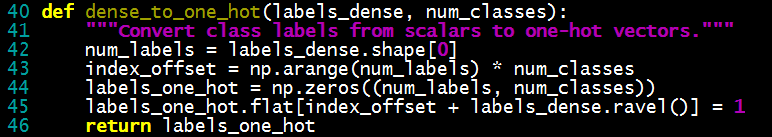
def parse(xml_filename):
logging.info("Parsing %s" % xml_filename)
annotations = []
# ET is the library we use to parse xml data
tree = etree.parse(xml_filename)
root = tree.getroot()
# header = parse_header(root)
# readingSession-> holds radiologist's annotation info
for read_session in root.findall('nih:readingSession', NS):
# to hold each radiologists annotation
# i.e. readingSession in xml file
rad_annotation = RadAnnotation()
rad_annotation.version = \
read_session.find('nih:annotationVersion', NS).text
rad_annotation.id = \
read_session.find('nih:servicingRadiologistID', NS).text
# nodules
nodule_nodes = read_session.findall('nih:unblindedReadNodule', NS)
for node in nodule_nodes:
nodule = parse_nodule(node)
if nodule.is_small:
rad_annotation.small_nodules.append(nodule)
else:
rad_annotation.nodules.append(nodule)
# non-nodules
non_nodule = read_session.findall('nih:nonNodule', NS)
for node in non_nodule:
nodule = parse_non_nodule(node)
rad_annotation.non_nodules.append(nodule)
annotations.append(rad_annotation)
return annotations
- 【分析结节头】
def parse_header(root):
header = AnnotationHeader()
print root.findall('nih:*', NS)
resp_hdr = root.findall('nih:ResponseHeader', NS)[0]
header.version = resp_hdr.find('nih:Version', NS).text
header.message_id = resp_hdr.find('nih:MessageId', NS).text
header.date_request = resp_hdr.find('nih:DateRequest', NS).text
header.time_request = resp_hdr.find('nih:TimeRequest', NS).text
header.task_desc = resp_hdr.find('nih:TaskDescription', NS).text
header.series_instance_uid = resp_hdr.find('nih:SeriesInstanceUid', NS).text
date_service = resp_hdr.find('nih:DateService', NS)
if date_service is not None:
header.date_service = date_service.text
time_service = resp_hdr.find('nih:TimeService', NS)
if time_service is not None:
header.time_service = time_service.text
header.study_instance_uid = resp_hdr.find('nih:StudyInstanceUID', NS).text
return header
- 【分析结节,非结节】
具体详见源代码
3. dicom_set.py(部分)
CT图像有三种,分别是CT,CR,DX,这个也可以查看LIDC-IDRI中的Modality,如下图所示:
【基类DcmImage】
【三种子类 -> CTImage, CRImage, RXImage】
- CTImage
- CRImage
- DXImage
4. lidc_xml_parser.py(部分)
主要就是分析XML文件,提取所有需要用到的信息,详见源代码。
5. prepare_data.py(部分)
【判断指定位置是否有结节】
【One-hot编码】
【处理结节】
五、数据特征分析
由于数据具有很多不同的特征,有的对后期训练比较有利,有的却不利于后期的训练,需要进行各种预处理之类的。
【有利点】
- CT图像的连续性(等间隔多次断层扫描,可以更有利于对结节的判别)
- 三维CT图像(不同轴切方向扫描)
- 进行预处理(如去噪,增强,平滑等处理)
【不利点】
- 数据不平衡,不对称(带标注的数据点所占的比例太低,不到1/1000),可以考虑对positive进行旋转,增加数据源,对negtive进行削减,只取部分等;
- 图像的模糊性(灰度模糊性,几何模糊性,和不确定性等),存在伪影(被测者自主或者不自主的运动所致)
- 原始图像的格式采用的是Dicom格式,需要进行各种预处理才能拿来用,前期比较麻烦,如果处理好了,就相对比较好用了
【数据切割方法】
- 顺序切割
切割方法:CT图片大小为 512x512,按32个像素为步长,截取 64x64 大小的块,每张原始图可切出225张训练图。
标注方法:若64x64图片中,存在结节的中心点,则标注为True,否则标注为False。- 仅取标注点
由于在原始标注中描述了所有的结节和非结节,他们在数量上相当,所以只采集所有有标注的图片来进行识别。
切割方法:读取结节的坐标,以结节中心点为切割中心点截取64x64大小的图片。
标注方法:若图像为结节点,则标注为True,否则标注为False。- 标注图+其他非标注切割
由于仅取标注点的图片来训练,无法让分类器识别影像边缘位置,会造成大量误判,因此再重新调整数据集,将有标注的图和没有标注的图都加入训练集。
切割方法:对于所有标注的点,采用 #仅取标注点# 的方法截取。对于其它所有没有标注的方格,按 #顺序切割# 的方式获得。
标注方法:将数据分为4类:1) >=3mm结节, 2) <3mm结节, 3) >=3mm非结节, 4) 无标注。有原始标注的图片会组成1, 2, 3类,其它顺序截取的图片会构成第4类。
注:
【Data Imbalance】
对于医疗图像来说,大部分图像是正常的,非标记的数据只占非常小的比例。以本例中,标记数据与非标记数据的比例至少为 1:1000 以上。在先验概率如此小的情况下,按照正确率而言,即使将所有数据都判定为正常非病患,正确率也可以高达99.9%以上。然而对于病患诊断,不能漏诊则是重中之重。因此,需要调整数据集的分布,放大Positive数据的比例,减小Negative数据的比例,让分类器可以有效识别positive。
在实验中,按以下方法调整数据集:
- 旋转有标记的图片,每次旋转27度,每次旋转后形成一张新的图片。这样所有有原始标记的图片则扩大了10倍。
- 所有没有原始标记的图片,只取2%,剩下则全部舍弃。
Loss Function:
对于医疗诊断而言,漏诊的代价更大,即将positive判定为negative的代价是更大的更严重的错误。
判定错误的代价定义为:
六、短期总结
1. 知识储备
- 做出一个checklist,逐步完善
- 将自己学的,做的的东西写出来,总结成文,发出去
- 能够将自己掌握的知识讲出来,并且让别人能够听懂
2.经验积累(项目)
- 对于简单的问题,能够稳定到一个比较好的结果,比较高的准确率
- 对于没有接触过的项目,遇到新问题时,能够快速定位,及时解决(比如服务器出现问题,能够半小时定位,三小时解决恢复服务)
Reference:
[1] LIDC-IDRI 官网
[2] Lung Image Database Consortium (LIDC) Nodule Size Report
[3] 肺部CT图像分析及特征提取研究
[4] 肺结节CT图像特征提取及SVM分类方法研究
[5] Computer-aided classification of lung nodules on computed tomography images via deep learning technique (paper)
[6] Computerized Detection of Lung Nodules in Thin-Section CT Images by use of Selective Enhancement Filters and an Automated Rule-Based Classifier (paper)
[7] DICOM的常用Tag分类和说明
[8] 肺结节CT征象及显示
[9] CT影像的识别
[10] 基于分类技术的肺部CT图像识别
[11] 关于HDF文件的一点概述(HDF4,HDF5)
[12] HDF5 小试——高大上的多对象文件格式
[13] 基于ct影像孤立性肺结节识别与分析
[14] 肺结节的影像诊断和鉴别诊断 (ppt)
[15] 肺部结节的CT鉴别诊断 (ppt)
(注:感谢您的阅读,希望本文对您有所帮助。如果觉得不错欢迎分享转载,但请先点击 这里 获取授权。本文由 版权印 提供保护,禁止任何形式的未授权违规转载,谢谢!)