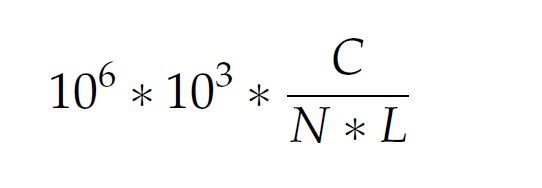转自:http://blog.sciencenet.cn/home.php?mod=space&uid=635619&do=blog&id=884213
用tophat和cufflinks分析RNAseq数据
人的基因组一共有两万多个基因,但是这些基因不是每时每刻都在表达,在不同发育时期和不同组织中,基因的表达是不同的,一个检测这些表达的有效的方法就是RNA-seq,它结合了下一代测序的技术来对细胞整个的mRNA进行测序,从而确定每一个基因的表达量和表达区段,主要用在分析不同条件下细胞内基因表达差异和分析基因表达的不同可变剪接上。
RNAseq分析大致分下面几个步骤,首先要把测到的序列map到基因组上,然后根据map到的区段对细胞构建转录本,然后比较几种细胞的转录本并且合并,最后衡量差异和可变间接和其他的分析。
1. Mapping
所有的序列分析的第一步,都是把测到的序列map到基因组上,这样就能知道序列原来是在基因组的什么地方。mapping一般基于两种快速索引算法,一种是哈希,MOSAIK,SOAP,SHRiMP用的就是这种算法,在对参照基因组建好哈希表之后,可以在常数次的运算里查找到给定序列的位置,虽然高效,但是由于基因组有些区段重复性很高,所以查找次数虽是常数,但有时会变得非常大,降低效率;还有一种叫Burrows-Wheeler变换,BWA,Bowtie和SOAP2都是用它,Burrows-Wheeler变换的设计比哈希更加巧妙,它最开始是一种文本压缩算法,文本重复性越高,它的压缩比就越大,这正好克服了基因组重复性高的问题,而且对于一个精确的序列查找,最多在给定序列的长度的次数里就能找到匹配,所以说基于Burrows-Wheeler变换的软件在mapping里用得更加广泛。可是RNAseq的map还有另外一个问题,那就是要允许可变剪接的存在,因为一条RNA不一定是一个外显子表达出来的,也有可能是几个外显子结合在了一起,原来基因里的内含子被空了出来,这些内含子的长度从五十到十万个碱基不等,如果直接用DNAseq的方法的话去在基因组里寻找,有些正好在两个exon连接处的序列就会有错配,而且有些在进化过程中遗漏下来的假基因是没有intron的,这样就导致有些序列会被map到假基因上去,使假基因的表达变得很高,所以,传统的bwa和bowtie在RNAseq里都不是最好的选择。
更加适合RNA mapping的软件需要克服上面的两个问题,Tophat,subread, STAR, GSNAP, RUM,
MapSplice都是为RNA测序而开发的
我只用过Tophat,它的新版Tophat2,在map的过程中分三个步骤:
如果基因注释文件存在的话,它会先用注释文件的转录组来map;
然后再对剩下的序列用bowtie进行普通的map;
最后再用bowtie里用过的所有的序列做剪接map,所以跟其他的软件比起来会有比较高的正确率。
在运行tophat之前,要对参考基因组作index,samtools可以轻松搞定。
samtools faidx hg38.fasta
如果用的是tophat的话,还需要用bowtie2做index.
bowtie2-build genome.fa genome
hg38.fasta是人类的参考基因组,对参考基因组做index是为了提高mapping时查找的效率.
(对懒人来说,在这里可以直接下载到Bowtie打包做好的index文件,这样就可以省略掉做index的步骤了 :D )
然后用Tophat开始mapping:
path/tophat
-p 8
-G $path_ref/Homo.GTF
-o tophat_output
$path_ref/hg38
single_end.fastq;
-p指定用几个线程来工作,-G指定注释文件的位置,-o指定输出文件的路径和文件名,最后两个参数分别告诉参考基因组的位置和要map的fastq文件。在参考基因组里不用加.fa的后缀,因为程序还要去寻找其他后缀的index文件。
这步的运行时间是根据fastq文件的大小和设定的线程数来决定的,一般单端8个线程需要每G一个小时,双端各4G,线程数设为16的话需要五六小时,运行完之后会在fastq文件的目录里产生一个-o命令指定的文件夹,这个文件夹里有几个bam文件和bed文件,还有一个summary,在下一步需要用到的是accepted_hits.bam这个文件。
注释文件的后缀是GTF,它包含了所有已知的基因的外显子在基因组中的位置。(hg38的注释文件可以在这里下载)所以对于已经map在基因组上的序列,我们可以直接根据它的位置从注释文件里查找它是不是属于一个外显子,或者是一个转录本。对于要不要在这一步提供注释文件,各有各的看法,我用单端测序的序列用两种方法做了实验,发现他们有些差别:
有注释的时候:
没注释的时候:
Mapping的最后一步是去除map到基因组中多于一处的序列,如果出现好几个序列都map在完全相同的一个区段,那么就应该只保留一个这样的序列,所以,只保留匹配最高的那一个。而且这样的序列占很大一部分,这步也很简单,samtools里的rmdup可以轻松解决:
samtools rmdup -s input.bam output.bam
-s小写是告诉samtools,bam文件是单端测序的结果,不指定-s的话默认是双端。
2 构建转录本
Mapping完了以后,cufflinks就可以把map到基因组里的序列组装成一个转录组了,这个转录组理论上包含了所有当时细胞里的所有mRNA,组装好的转录组包含了可能的剪切信息和所有转录的表达量,这个表达量是根据map到基因组的序列的总数和每个转录片断的长度进行归一化的,听起来比较难懂,它是对于在转录片断里的每一千个碱基对,在每一百万个成功map的序列中,map在这一千个碱基对上的序列的比例,fragments
per kilobase of transcript per million mapped fragments (FKPM)。
FPKM是这么算出来的:
在公式里,C代表的是map在这一千个碱基对上的序列的个数,N是所有成功map的序列的个数,L是转录片断的长度。
命令:
~/software/cufflinks-2.2.1/cufflinks
-g /wrk/ref/Homo_sapiens.GRCh38.78.gtf
-o ../cufflinks_sample1
-p 8
accepted_hits.bam;
然后在输出的cufflinks_sample1文件夹里会产生四个文件,genes.fpkm_tracking, isoforms.fpkm_tracking, skipped.gtf 和 transcripts.gtf,下一步需要用到的就是transcripts.gtf这个文件,transcripts.gtf就是这个样品的转录组。
3。合并转录组
为了比较不同样本间的差异,需要把实验组和对照组的转录组合并起来,cuffmerge不仅可以用来合并两个或者多个转录组,还能把注释过后的基因组的信息也合并起来,从而找到新的基因可变剪接,提高合并转录组的质量。有人说需要在合并之前用cuffcompare,但从官网的说法来看没有是必要的。它们最大的区别是,cuffcompare不改变原有样本里的转录片段,只是将他们的位置作比较,输出的combined文件也只是包含了所有的小转录片段,而cuffmerge会寻找几个样本间的不同,试着把几个样本里的转录片段从头开始尽可能拼接成更长更完整的片段,所以cuffmerge的输出merge文件比cuffcompare输出的combined文件更有说服力。两个小工具的作用都是为了生成一个合并的注释文件给接下来要用的cuffdiff。
命令:
~/software/cufflinks-2.2.1/cuffmerge
-g /wrk/ref/Homo_sapiens.GRCh38.78.gtf
-s /wrk/ref/hg38.fa
-p 16
assembly_list.txt
在最后输入的assemby_list.txt文件里,要写上所有需要合并的transcripts.gtf文件的路径,两个和多个都可以,然后cuffmerge就会生成一个merged文件夹,里面有一个merged.gtf,这个文件就是合并好的转录组。
4. 基因差异表达分析
最后一步就是分析可变剪接和差异表达了,用到的小工具叫cuffdiff,这个的输入比较复杂,不仅需要上一步的merge文件,还需要每个样本的mapping结果的bam文件,最后还需要对每一个bam文件对应的样本按顺序起一个名字作为标签,标签之间记得用逗号。
~/software/cufflinks-2.2.1/cuffdiff
-o diff22rv1/
-labels vap,vcap_neg_control
-p 32
-u merged_asm/merged.gtf
vcap/accepted_hits.bam vcapneg/accepted_hits.bam
在这个例子里只有一个case和一个control,所以我们只要两个标签,在最后按顺序输入bam文件。
diff的输出比较多,他会对每个基因,每个转录片段,每个编码序列,和每个基因的不同剪接体进行FPKM,个数和样本间差异进行分析,最后生成几组不同的文件,按照不同的分析需求,就可以试着往下分析了。
到现在结果就基本都差不多了,剩下的主要是作图了,发现新的基因的可变剪接,发现差异表达的基因,对差异表达的基因做富集分析等等。作图也是非常重要的环节,再好的结果也需要有好的图表示出来,可变剪接我也没做过,但是如果做差异表达的话,CummRbund是一个非常兼容cufflinks的作图工具。
CummRbund是R里的一个包,用来分析cuffdiff的结果非常方便,在安装好这个包之后,要做的只是把路径改在cuffdiff生成结果的文件夹里,然后在R里运行这两行代码就好了。
library(cummeRbud) #这一行需要在R里加载已经安装好的cummeRbud包
cuff <- readCufflinks() #这一行就告诉R把所有cuffdiff生成的结果读入cuff这个变量里。
下一步就可以作差异表达的基因的热图了,这里稍微复杂一点:
#取前100个差异最显著的基因,或者取多少个随你便,标准是t检验的p值,p值越小差异就越大。
gene.diff <- diffData(genes(cuff))
gene.diff.top <- gene.diff[order(gene.diff$p_value),][1:100,]
#找到这前100个差异基因的ID
myGeneIds <- gene.diff.top$gene_id
# 然后根据基因ID来得到基因的名字
myGenes <- getGenes(cuff, myGeneIds)
# 是画图
csHeatmap(myGenes, cluster="both")
作出的图基本上是这个样子的:
接下来就可以进行富集分析了,有很多种方法,可以直接把基因的名字导出来后上传到david来分析,也可以用bioconductor里的Goseq包来分析,详细的goseq用法的代码有点长,我以后会写,如果需要的话可以直接去这里下载我使用Goseq时的代码。
Cuff系列的分析流程是这篇文章介绍的,它里面也有非常详细的命令和例子,可以去这里看看。Differential
gene and transcript expression analysis of RNA-seq experiments with
TopHat and Cufflinks