- C51填坑记:中断处理导致主程序函数参数改变
albert_812
C51C51DataOverlay中断参数异常改变
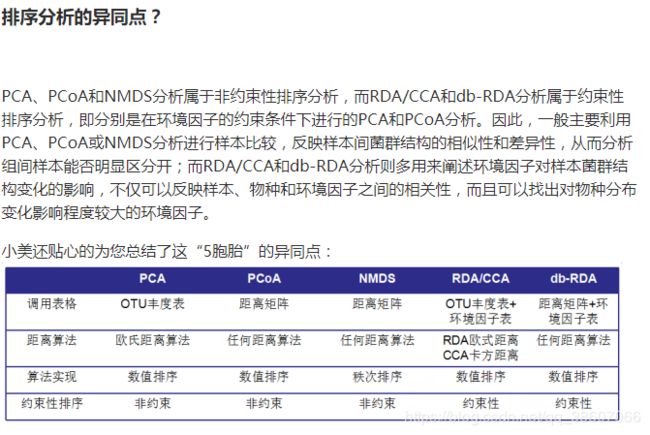
1.现象平台:keilc51,中颖SH79F7019A现象:在增加了一个中断处理逻辑后,发现主程序异常,断点调试发现某个函数的参数被改变了,程序使用了错误的数据导致逻辑出错。2.排查初步分析,可能原因如下:1.参数寄存器(R0-R7)的值,被中断函数改变。2.堆栈溢出。2.1参数寄存器首先排查参数寄存器(中断里面调用了函数,有参数传递)。通过仿真器观察中断函数汇编代码,发现在进入中断之前是对R0-
- python正则匹配11个数字_python正则表达式re.match()匹配多个字符方法的实现
小馬锅
python正则匹配11个数字
1.*表示匹配任意多个字符\d*表示匹配任意多个数字字符importretext="123h1elloworld"text1="123Helloworld456"text2="helloworld"res=re.match("\d*",text)res1=re.match("\d*",text1)res2=re.match("\d*",text2)print(res.group())print(r
- STM32+ESP8266连接网络的几种方法
op:)
stm32stm32嵌入式
1、SmartConfig上篇文章提到的2、连接固定的路由在程序中写死;例如:voidESP8266_Init_Fixed_WiFi(void){ESP8266_Clear();/*AT测试*/printf("0.AT\r\n");ESP8266_SendCmd("AT\r\n","");delay_ms(500);/*重启*/printf("1.RST\r\n");ESP8266_SendCmd
- 达人评测 锐龙9 8940HX和r9 7940HX差距大不大
ThantZinHtay
cpu
R97940HX采用了Zen4架构,5nm制作工艺8核16线程,主频2.4GHz,最高睿频5.2GHz三级缓存64MB功耗55w选R97940HX还是锐龙98940HX这些点很重要http://www.adiannao.cn/dyR98940HX采用Zen5架构和5nm生产工艺16核心32线程基础频率为2.3GHz,最高加速频率5.3GHz三级缓存为64MB热设计功耗(TDP)55W
- Winrar X64_V6.24_win10/win11
Malwolf
winrar
SW_WinRAR_W11_64_V6240_20231108R.zipwinrar解压工具,免费版,无需破解,也不是破解版的,国内硬件大厂提供的官方版本。
- Mariadb-Server的二进制安装
linux土老帽
linux土老帽mariadblinuxcentos
搭建环境:OS:centos7.6版本mariadb:mariadb-10.2.25-linux-x86_64.tar.gz#下载地址:DownloadMariaDBServer-MariaDB.org1.创建用户组useradd-r-s/sbin/nologin-d/data/mysqlmysql#创建用户-r系统账户-s指定shell/sbin/nologin-d指定家目录但是不创建原因是系统
- linux服务器上创建一个文件需要授权一次的问题根源:umask
qq_30024063
linux运维服务器
umask命令用于设置文件的默认权限掩码。文件的权限掩码决定了新建文件的默认权限。umask命令的语法如下:umask[-S][模式]其中,-S选项用于以符号方式显示当前的权限掩码。模式表示要设置的新的权限掩码,可以使用八进制或者符号两种方式。在Linux系统中,每个文件都有三个属性:所有者权限、所属组权限和其他用户权限。每个属性有读、写和执行三个权限,分别用r、w和x表示。对于每一个属性,权限可
- Python_计算两个省市之间的直线距离_2506
夏天里的肥宅水
PYTHONpythonspring开发语言
更新代码上一版链接importpandasaspdimporttimeimportpickleimportosimportsysfromgeopy.geocodersimportNominatimfromgeopy.distanceimportgeodesicfromtqdmimporttqdm#ConfigurationINPUT_FILE=r"距离.xlsx"#输入文件路径OUTPUT_FIL
- 使用Docker部署mysql8
小莫分享
dockeradb容器
1、拉取mysql8的镜像:1[root@i-zgn6som8~]#dockerpullmysql:8.02、创建配置和数据文件夹并修改权限:1234mkdir-p/data/mysql8/confmkdir-p/data/mysql8/datachmod-R755/data/mysql8/3、配置一个自定义的配置文件my.cnf:1vim/data/mysql8/conf/my.cnf文件内容如
- 右移的错误使用 造成 超出时间限制 解决方案
aPurpleBerry
LeetCode做题总结算法力扣二分查找
题目链接:https://leetcode.cn/problems/guess-number-higher-or-lower/?envType=study-plan-v2&envId=leetcode-75最开始的代码varguessNumber=function(n){letl=1,r=n;while(l>1;if(guess(m)===-1){r=m-1;}elseif(guess(m)===
- 安卓开发 手动构建 .so
XCZHONGS
android
手动构建.so(兼容废弃ABI)下载旧版NDK(推荐r16b)地址:https://developer.android.com/ndk/downloads/older_releases下载NDKr16b(最后支持armeabi、mips、mips64的版本)使用ndk-build手动构建(不使用Gradle)在源文件目录下执行D:\ideal\androidstudio\sdk\ndk\16.1.
- 莫队算法 —— 将暴力玩出花
秒啦
算法
莫队算法——将暴力玩出花一、为什么需要莫队?——暴力法的瓶颈我们已经学会了用分块处理一些在线的区间问题。现在,我们来看一类特殊的离线区间查询问题。“离线”意味着我们可以把所有查询先读进来,再按我们喜欢的顺序去处理它们。思考一个问题:给定一个长度为N的数组,M次询问。每次询问一个区间[l,r],问区间内有多少种数字至少出现了2次?那我们回到最朴素的暴力。纯暴力:对于每个询问(l,r),都for一遍,
- 关于 webpack 打包结构 详解
shenyan~
webpack前端node.js
一、整体结构概览Webpack打包代码示例:(function(t,n){...i=function(){returnfunction(t){vare={};//模块缓存functionn(r){...}//模块加载器__webpack_require__//工具函数定义↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓↓n.m=t;//模块表(每个模块对应一个函数)n.c
- 【lua】Linux上安装lua和luarocks包管理工具
果壳~
lualinux开发语言
目录安装lua安装luarocksluarocks其他命令安装lua首先打开lua官网https://lua.org点击download就可以看到安装脚本新建一个目录将压缩包下载到这个目录里curl-L-R-Ohttps://www.lua.org/ftp/lua-5.4.8.tar.gztarzxflua-5.4.8.tar.gzcdlua-5.4.8makealltest#最后还得加上make
- vue el-date-picker 直接赋值时控件失效
梓暮
ITvue.js前端elementui
项目场景:前端vueel-date-picker控件无故失效问题描述本人是主打后端,新进的公司要求前后端全干,然后又因为前端做得少,所以经常碰到一些奇怪的问题,比如以下操作,是给vue前端el-date-picker这个时间控件赋值,但是发现,数据是赋值上去了,但是控件失效了,怎么点都没用if(resData.batchEntity.manage_scene_start_time!=null&&r
- Tiktok App 登录账号、密码、验证码 XOR 加密算法
抖音App登录账号、密码、验证码XOR加密算法%E9n+z,\&R1a4b.^流程分析登录TiktokAPP时,通过抓包发现账号密码是非明文传输的。getUserProfile($userId,$secUid);echo"\n\n视频列表:\n";echo$tiktok->getMixList($userId);//示例:加密后的密码hex字符串$encrypted_hex="7472607771
- python做生物信息学分析_Python从零开始第五章生物信息学①提取差异基因
吴敬欣
python做生物信息学分析
目前来说,做生物信息学的人越来越多,但是我觉得目前而言做生信的主要有三类人:老本行是做实验的,做生信可能是为了辅助研究或者是为了发paper(有非常多的临床生选择趟生信这波水)主要是做生信的,主要涵盖高通量测序数据分析,组学数据分析等等,专门从事生物学数据分析的这群人,其大部分也是本科生物狗作为强大的生力军,以调包写R,python为主。那么这群人就要熟悉看各种包的tutorial以及如何进行常规
- 实例化Bean对象的三种方式
越来越无动于衷
javasql开发语言
默认是无参数的构造方法(默认方式,基本上使用)静态工厂实例化特点:工厂方法属于静态方法,可直接通过类名调用,无需先创建工厂类的对象。优势:调用起来更为简便,性能方面也稍占优势。Spring配置:class属性指向静态工厂类,factory-method属性指向静态方法。示例代码:publicclassStaticFactory{publicstaticUserServicecreateUs(){r
- Python打卡训练营day31
weixin_70153780
Python打卡训练营python开发语言
规范的文件命名目标:将文件夹中的文件按规则重命名(如添加前缀、序号等)。#示例importos#定义文件夹路径folder_path=r'C:\Users\Name\Documents\Project\Files'#遍历文件夹中的文件forindex,filenameinenumerate(os.listdir(folder_path)):#拆分文件名和后缀file_ext=os.path.spl
- Go Web开发框架实践:模板渲染与静态资源服务
程序员爱钓鱼
golang开发语言后端网络
Gin不仅适合构建API服务,也支持HTML模板渲染和静态资源托管,使其可以胜任中小型网站开发任务。一、模板渲染基础1.加载模板文件使用LoadHTMLGlob或LoadHTMLFiles方法加载模板:r := gin.Default()r.LoadHTMLGlob("templates/*") // 支持通配符或:r.LoadHTMLFiles("templates/index.tmpl", "
- STM32 CAN 单个设备回环通信及三个设备互相通信
2401_88885971
网络
一、基本流程基础配置分为三大块:1.CAN外设的初始化RCC时钟初始化(GPIO时钟和CAN1的时钟)GPIO初始化(CAN_TX复用推挽输出模式,CAN_RX上拉输入模式)整个CAN外设的初始化(结构体配置,模式,波特率,各种其余小功能......)单独对过滤器初始化(结构体配置,过滤器位宽、模式、R1R2的值、FIFO关联、使能的参数)最后若是使用中断再加上ITConfig函数使能中断输出,再
- 如何制作一份E-R图
菜汪本汪
数据库
ER模型,全称为实体联系模型、实体关系模型或实体联系模式图(ERD)(英语:Entity-relationshipmodel)由美籍华裔计算机科学家陈品山发明,是概念数据模型中高层描述所使用的数据模型或模式图。ER模型常用于信息系统设计中。比如,在概念结构设计阶段,ER模型用来描述信息需求和/或要存储在数据库中的信息类型,但是数据建模技术可以用来描述特定论域(感兴趣的区域)的任何本体(对使用的术语
- 计算机毕业设计Springboot农副产品线上商场系统 基于Spring Boot的农产品电商交易平台设计与实现 Spring Boot架构下的农产品线上商城系统开发
路可程序设计
课程设计springboot后端
计算机毕业设计Springboot农副产品线上商场系统r7duh7er(配套有源码程序mysql数据库论文)本套源码可以先看具体功能演示视频领取,文末有联xi可分享随着互联网技术的飞速发展,电子商务已经成为人们生活中不可或缺的一部分。尤其是在农产品销售领域,传统的线下销售模式面临着诸多限制,如销售渠道狭窄、信息不对称、销售成本高等问题。为了打破这些限制,提升农产品的销售效率和市场覆盖范围,开发一个
- 【力扣 中等 C】912. 排序数组
黑听人
c语言leetcode数据结构算法开发语言
目录题目解法一:快速排序解法二:归并排序题目解法一:快速排序voidswap(int*a,int*b){inttmp=*a;*a=*b;*b=tmp;}voidpartition(int*nums,intleft,intright,int*leftMax,int*rightMin){intrandomNum=nums[left+rand()%(right-left+1)];intl=left,r=
- SVN的项目文件夹不显示图标问题解决
泥巴客
svn
文章目录一、问题阐述二、解决方案1.打开注册编辑表:windows+R(键盘按键)2.找到Tortoise相关项3.添加空格调整排序4.验证排序结果一、问题阐述安装了SVN,发现从服务器拉取文件到本地仓库后,对应的文件没有绿色红色的小钩图标,然后解决后,总结一下二、解决方案1.打开注册编辑表:windows+R(键盘按键)输入:regedit2.找到Tortoise相关项导航到路径:HKEY_LO
- 认识Jacobian
一碗姜汤
统计学习线性代数矩阵
Jacobian(雅可比矩阵)是数学中用于描述多元函数在某一点处导数的重要概念,广泛应用于微积分、微分几何、数值分析等领域。以下从定义、数学表达、几何意义、应用场景等方面详细解析:一、定义与数学表达1.基本定义若有一个从欧式空间Rn\mathbb{R}^nRn到Rm\mathbb{R}^mRm的多元函数:f:Rn→Rmf:\mathbb{R}^n\to\mathbb{R}^mf:Rn→Rm,其分量
- Linux 下安装MySQL 5.7与 8.0详情
我姓谭却弹不死你
LiunxMySQLmysqlcentoslinux
官网资源下载1、检查/tmp临时目录权限,将/tmp目录及所有子目录和文件的权限设置为所有用户可读、写和执行[root@localhost~]#chmod-R777/tmp2、安装前检查相关依赖2.1、查找是否安装了libaio包。libaio是Linux下异步I/O库的一个实现,用于提供异步I/O支持。[root@localhost~]#rpm-qa|greplibaiolibaio-0.3.1
- pymilvus.exceptions.MilvusException: <MilvusException: (code=0, message=attempt #0: channel=by-dev-r
Langchain连接AI大模型连接milvus数据库,向milvus同步数据时报错如下:ERROR:ExceptioninASGIapplicationTraceback(mostrecentcalllast):File"/home/devops/.local/lib/python3.10/site-packages/uvicorn/protocols/http/httptools_impl.p
- (线性代数最小二乘问题)Normal Equation(正规方程)
音程
数学线性代数机器学习人工智能
NormalEquation(正规方程)是线性代数中的一个重要概念,主要用于解决最小二乘问题(LeastSquaresProblem)。它通过直接求解一个线性方程组,找到线性回归模型的最优参数(如权重或系数)。以下是详细介绍:1.定义与数学表达式给定一个超定方程组(方程数量多于未知数):Ax=bA\mathbf{x}=\mathbf{b}Ax=b其中:A∈Rm×nA\in\mathbb{R}^{m
- Ubuntu中使用netcat发送16进制网络数据包
张太行_
ubuntu网络linux
在Ubuntu中,使用netcat(nc)发送16进制数据,可以通过echo、printf或xxd等工具将十六进制数据转换为二进制格式,再通过管道传递给nc。以下是几种方法:方法1:使用echo+xxd(推荐)xxd是一个十六进制转二进制的工具,可以方便地将十六进制字符串转换为原始二进制数据。示例(发送UDP十六进制数据):echo"48656c6c6f20576f726c64"|xxd-r-p|
- Java实现的简单双向Map,支持重复Value
superlxw1234
java双向map
关键字:Java双向Map、DualHashBidiMap
有个需求,需要根据即时修改Map结构中的Value值,比如,将Map中所有value=V1的记录改成value=V2,key保持不变。
数据量比较大,遍历Map性能太差,这就需要根据Value先找到Key,然后去修改。
即:既要根据Key找Value,又要根据Value
- PL/SQL触发器基础及例子
百合不是茶
oracle数据库触发器PL/SQL编程
触发器的简介;
触发器的定义就是说某个条件成立的时候,触发器里面所定义的语句就会被自动的执行。因此触发器不需要人为的去调用,也不能调用。触发器和过程函数类似 过程函数必须要调用,
一个表中最多只能有12个触发器类型的,触发器和过程函数相似 触发器不需要调用直接执行,
触发时间:指明触发器何时执行,该值可取:
before:表示在数据库动作之前触发
- [时空与探索]穿越时空的一些问题
comsci
问题
我们还没有进行过任何数学形式上的证明,仅仅是一个猜想.....
这个猜想就是; 任何有质量的物体(哪怕只有一微克)都不可能穿越时空,该物体强行穿越时空的时候,物体的质量会与时空粒子产生反应,物体会变成暗物质,也就是说,任何物体穿越时空会变成暗物质..(暗物质就我的理
- easy ui datagrid上移下移一行
商人shang
js上移下移easyuidatagrid
/**
* 向上移动一行
*
* @param dg
* @param row
*/
function moveupRow(dg, row) {
var datagrid = $(dg);
var index = datagrid.datagrid("getRowIndex", row);
if (isFirstRow(dg, row)) {
- Java反射
oloz
反射
本人菜鸟,今天恰好有时间,写写博客,总结复习一下java反射方面的知识,欢迎大家探讨交流学习指教
首先看看java中的Class
package demo;
public class ClassTest {
/*先了解java中的Class*/
public static void main(String[] args) {
//任何一个类都
- springMVC 使用JSR-303 Validation验证
杨白白
springmvc
JSR-303是一个数据验证的规范,但是spring并没有对其进行实现,Hibernate Validator是实现了这一规范的,通过此这个实现来讲SpringMVC对JSR-303的支持。
JSR-303的校验是基于注解的,首先要把这些注解标记在需要验证的实体类的属性上或是其对应的get方法上。
登录需要验证类
public class Login {
@NotEmpty
- log4j
香水浓
log4j
log4j.rootCategory=DEBUG, STDOUT, DAILYFILE, HTML, DATABASE
#log4j.rootCategory=DEBUG, STDOUT, DAILYFILE, ROLLINGFILE, HTML
#console
log4j.appender.STDOUT=org.apache.log4j.ConsoleAppender
log4
- 使用ajax和history.pushState无刷新改变页面URL
agevs
jquery框架Ajaxhtml5chrome
表现
如果你使用chrome或者firefox等浏览器访问本博客、github.com、plus.google.com等网站时,细心的你会发现页面之间的点击是通过ajax异步请求的,同时页面的URL发生了了改变。并且能够很好的支持浏览器前进和后退。
是什么有这么强大的功能呢?
HTML5里引用了新的API,history.pushState和history.replaceState,就是通过
- centos中文乱码
AILIKES
centosOSssh
一、CentOS系统访问 g.cn ,发现中文乱码。
于是用以前的方式:yum -y install fonts-chinese
CentOS系统安装后,还是不能显示中文字体。我使用 gedit 编辑源码,其中文注释也为乱码。
后来,终于找到以下方法可以解决,需要两个中文支持的包:
fonts-chinese-3.02-12.
- 触发器
baalwolf
触发器
触发器(trigger):监视某种情况,并触发某种操作。
触发器创建语法四要素:1.监视地点(table) 2.监视事件(insert/update/delete) 3.触发时间(after/before) 4.触发事件(insert/update/delete)
语法:
create trigger triggerName
after/before
- JS正则表达式的i m g
bijian1013
JavaScript正则表达式
g:表示全局(global)模式,即模式将被应用于所有字符串,而非在发现第一个匹配项时立即停止。 i:表示不区分大小写(case-insensitive)模式,即在确定匹配项时忽略模式与字符串的大小写。 m:表示
- HTML5模式和Hashbang模式
bijian1013
JavaScriptAngularJSHashbang模式HTML5模式
我们可以用$locationProvider来配置$location服务(可以采用注入的方式,就像AngularJS中其他所有东西一样)。这里provider的两个参数很有意思,介绍如下。
html5Mode
一个布尔值,标识$location服务是否运行在HTML5模式下。
ha
- [Maven学习笔记六]Maven生命周期
bit1129
maven
从mvn test的输出开始说起
当我们在user-core中执行mvn test时,执行的输出如下:
/software/devsoftware/jdk1.7.0_55/bin/java -Dmaven.home=/software/devsoftware/apache-maven-3.2.1 -Dclassworlds.conf=/software/devs
- 【Hadoop七】基于Yarn的Hadoop Map Reduce容错
bit1129
hadoop
运行于Yarn的Map Reduce作业,可能发生失败的点包括
Task Failure
Application Master Failure
Node Manager Failure
Resource Manager Failure
1. Task Failure
任务执行过程中产生的异常和JVM的意外终止会汇报给Application Master。僵死的任务也会被A
- 记一次数据推送的异常解决端口解决
ronin47
记一次数据推送的异常解决
需求:从db获取数据然后推送到B
程序开发完成,上jboss,刚开始报了很多错,逐一解决,可最后显示连接不到数据库。机房的同事说可以ping 通。
自已画了个图,逐一排除,把linux 防火墙 和 setenforce 设置最低。
service iptables stop
- 巧用视错觉-UI更有趣
brotherlamp
UIui视频ui教程ui自学ui资料
我们每个人在生活中都曾感受过视错觉(optical illusion)的魅力。
视错觉现象是双眼跟我们开的一个玩笑,而我们往往还心甘情愿地接受我们看到的假象。其实不止如此,视觉错现象的背后还有一个重要的科学原理——格式塔原理。
格式塔原理解释了人们如何以视觉方式感觉物体,以及图像的结构,视角,大小等要素是如何影响我们的视觉的。
在下面这篇文章中,我们首先会简单介绍一下格式塔原理中的基本概念,
- 线段树-poj1177-N个矩形求边长(离散化+扫描线)
bylijinnan
数据结构算法线段树
package com.ljn.base;
import java.util.Arrays;
import java.util.Comparator;
import java.util.Set;
import java.util.TreeSet;
/**
* POJ 1177 (线段树+离散化+扫描线),题目链接为http://poj.org/problem?id=1177
- HTTP协议详解
chicony
http协议
引言
- Scala设计模式
chenchao051
设计模式scala
Scala设计模式
我的话: 在国外网站上看到一篇文章,里面详细描述了很多设计模式,并且用Java及Scala两种语言描述,清晰的让我们看到各种常规的设计模式,在Scala中是如何在语言特性层面直接支持的。基于文章很nice,我利用今天的空闲时间将其翻译,希望大家能一起学习,讨论。翻译
- 安装mysql
daizj
mysql安装
安装mysql
(1)删除linux上已经安装的mysql相关库信息。rpm -e xxxxxxx --nodeps (强制删除)
执行命令rpm -qa |grep mysql 检查是否删除干净
(2)执行命令 rpm -i MySQL-server-5.5.31-2.el
- HTTP状态码大全
dcj3sjt126com
http状态码
完整的 HTTP 1.1规范说明书来自于RFC 2616,你可以在http://www.talentdigger.cn/home/link.php?url=d3d3LnJmYy1lZGl0b3Iub3JnLw%3D%3D在线查阅。HTTP 1.1的状态码被标记为新特性,因为许多浏览器只支持 HTTP 1.0。你应只把状态码发送给支持 HTTP 1.1的客户端,支持协议版本可以通过调用request
- asihttprequest上传图片
dcj3sjt126com
ASIHTTPRequest
NSURL *url =@"yourURL";
ASIFormDataRequest*currentRequest =[ASIFormDataRequest requestWithURL:url];
[currentRequest setPostFormat:ASIMultipartFormDataPostFormat];[currentRequest se
- C语言中,关键字static的作用
e200702084
C++cC#
在C语言中,关键字static有三个明显的作用:
1)在函数体,局部的static变量。生存期为程序的整个生命周期,(它存活多长时间);作用域却在函数体内(它在什么地方能被访问(空间))。
一个被声明为静态的变量在这一函数被调用过程中维持其值不变。因为它分配在静态存储区,函数调用结束后并不释放单元,但是在其它的作用域的无法访问。当再次调用这个函数时,这个局部的静态变量还存活,而且用在它的访
- win7/8使用curl
geeksun
win7
1. WIN7/8下要使用curl,需要下载curl-7.20.0-win64-ssl-sspi.zip和Win64OpenSSL_Light-1_0_2d.exe。 下载地址:
http://curl.haxx.se/download.html 请选择不带SSL的版本,否则还需要安装SSL的支持包 2. 可以给Windows增加c
- Creating a Shared Repository; Users Sharing The Repository
hongtoushizi
git
转载自:
http://www.gitguys.com/topics/creating-a-shared-repository-users-sharing-the-repository/ Commands discussed in this section:
git init –bare
git clone
git remote
git pull
git p
- Java实现字符串反转的8种或9种方法
Josh_Persistence
异或反转递归反转二分交换反转java字符串反转栈反转
注:对于第7种使用异或的方式来实现字符串的反转,如果不太看得明白的,可以参照另一篇博客:
http://josh-persistence.iteye.com/blog/2205768
/**
*
*/
package com.wsheng.aggregator.algorithm.string;
import java.util.Stack;
/**
- 代码实现任意容量倒水问题
home198979
PHP算法倒水
形象化设计模式实战 HELLO!架构 redis命令源码解析
倒水问题:有两个杯子,一个A升,一个B升,水有无限多,现要求利用这两杯子装C
- Druid datasource
zhb8015
druid
推荐大家使用数据库连接池 DruidDataSource. http://code.alibabatech.com/wiki/display/Druid/DruidDataSource DruidDataSource经过阿里巴巴数百个应用一年多生产环境运行验证,稳定可靠。 它最重要的特点是:监控、扩展和性能。 下载和Maven配置看这里: http
- 两种启动监听器ApplicationListener和ServletContextListener
spjich
javaspring框架
引言:有时候需要在项目初始化的时候进行一系列工作,比如初始化一个线程池,初始化配置文件,初始化缓存等等,这时候就需要用到启动监听器,下面分别介绍一下两种常用的项目启动监听器
ServletContextListener
特点: 依赖于sevlet容器,需要配置web.xml
使用方法:
public class StartListener implements
- JavaScript Rounding Methods of the Math object
何不笑
JavaScriptMath
The next group of methods has to do with rounding decimal values into integers. Three methods — Math.ceil(), Math.floor(), and Math.round() — handle rounding in differen