1. 获取测序数据
Aspera 软件以最快速度传输全球数据,不受文件大小、传输距离或网络条件的影响。
ubuntu下安装Aspera软件
下载Aspera软件包,解压,安装,添加到环境变量
wget https://download.asperasoft.com/download/sw/conne
ct/3.6.2/aspera-connect-3.6.2.117442-linux-64.tar.gz
tar zvxf aspera-connect-3.6.2.117442-linux-64.tar.gz
sh aspera-connect-3.6.2.117442-linux-64.sh
echo 'export PATH=$PATH:[ascp所在目录]' >> ~/.bashrc
source ~/.bashrc
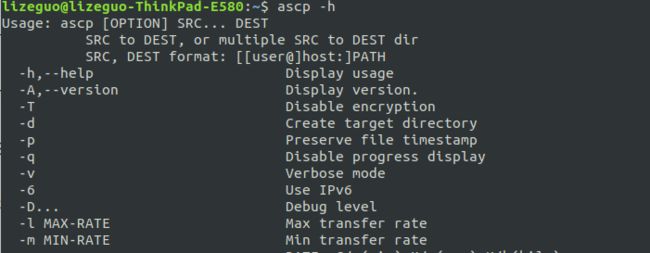
查看Aspera 工具下ascp 的帮助信息
ascp -h
SRA测序数据的下载
例如在NCBI的SRA数据库下载数据,这里选择NCBI 的ftp 服务器
网址为:ftp://ftp.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/
通过Aspera 下的工具ascp下载数据:
~/.aspera/connect/bin/ascp -T -i /home/lizeguo/.aspera/connect/etc/asperaweb_id_dsa.openssh -k 1 -l 200m [email protected]:/sra/sra-instant/reads/ByRun/sra/SRR/SRR620/SRR6208854/SRR6208854.sra ./Seqs/
参数解释:
USAGE:ascp [参数] 目标文件 目的地址
-T :不进行加密。若不添加此参数,可能会下载不了。
-i string :输入私钥,安装 aspera 后有在目录 ~/.aspera/connect/etc/ 下有几个私钥,使用 linux 服务器的时候一般使用 asperaweb_id_dsa.openssh 文件作为私钥。
-l string:设置最大传输速度,比如设置为 200M 则表示最大传输速度为 200m/s。若不设置该参数,则一般可达到10m/s的速度,而设置了,传输速度可以更高。
-k 1 :支持断点续传
--host=string:ftp的host名,NCBI的为ftp-private.ncbi.nlm.nih.gov;EBI的为
fasp.sra.ebi.ac.uk。
--user=string :用户名,NCBI的为anonftp,EBI的为era-fasp。
--mode=string :选择模式,上传为 send,下载为 recv。
--file-list:批量下载SRA文件的路径
2.测序数据的格式转换——SRA toolkit
NCBI开发,用于下载处理SRA文件,各种数据格式转换的工具包
SRA toolkit的下载、解压、安装
wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/2.9.2/sratoolkit.2.9.2-ubuntu64.tar.gz
tar -zvxf sratoolkit.2.9.2-ubuntu64.tar.gz -C ~/Biosofts/
echo 'export PATH=~/Biosofts/sratoolkit.2.9.2-ubuntu64/bin:$PATH' >> ~/.bashrc
source ~/.bashrc
现在不仅可以通过SRA toolkit做格式转换,还可以通过SRA toolkit下的工具prefetch 下载SRA 数据
例如下载SRR6232298.sra文件
prefetch SRR6232298
软件自动建立~/ncbi/public/sra文件夹,下载sra文件
ls ncbi/public/sra/
SRR6232298.sra
查看SRA文件类型和格式转换
我们可以进入NCBI的SRA数据库,搜索SRR6232298
可以看见它的layout为paired,说明是双末端测序,输出的文件为2个,这是我们在做格式转换的时候就需要输出两个文件:
fastq-dump --split-files SRR6232298.sra
ls
SRR6232298_1.fastq SRR6232298_2.fastq SRR6232298.sra
3.测序数据的质量控制——FastQC
用于测序数据质控的软件,下载后直接使用,需配置java环境
java安装包:
链接:https://pan.baidu.com/s/1Z9sS34oYfJl1GTaydrjekw
提取码:j1vu
复制这段内容后打开百度网盘手机App,操作更方便哦
java环境的安装
sudo mkdir /usr/java
sudo tar -zvxf BioSofts/jdk-8u172-linux-x64.tar.gz -C /usr/java/
cd /usr/java
sudo ln -s jdk1.8.0_172 latest
sudo ln -s /usr/java/latest default
sudo vi /etc/profile
在末尾添加这几行
export JAVA_HOME=/usr/java/latest
export PATH=$JAVA_HOME/bin:$JAVA_HOME/jre/bin:$PATH
export CLASSPATH=.:$JAVA_HOME/lib/dt.jar:$JAVA_HOME/lib/tools.jar
source /etc/profile
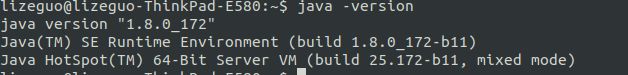
查看java的版本
java -version
FastQC的下载和安装
wget http://www.bioinformatics.babraham.ac.uk/projects/fastqc/fastqc_v0.11.7.zip
unzip fastqc_v0.11.7.zip -d ~/BioSofts/
echo 'export PATH=~/BioSofts/FastQC:$PATH'>>~/.bashrc
source ~/.bashrc
chmod 777 ~/BioSofts/FastQC/fastqc
fastqc -h
fastqc质量评估
fastqc SRR6232298_1.fastq
得到的结果如下
将html文件用浏览器打开就可以查看结果。
测序数据的数据过滤
常用的数据过滤软件有Trimmomatic、seqtk、cutadapt、 bbduk(BBmap),用来切除低质量碱基和接头序列。
Trimmomatic 是一个广受欢迎的 Illumina 平台数据过滤工具。
Trimmomatic的安装
wget http://www.usadellab.org/cms/uploads/supplementary/Trimmomatic/Trimmomatic-0.38.zip
unzip Trimmomatic-0.38.zip -d ~/BioSofts/
java -jar ~/BioSofts/Trimmomatic-0.38/trimmomatic-0.38.jar
Trimmomatic的使用
java -jar SE [-threads ] [-phred33 | -phred64] [-trimlog] 参数详见:http://www.biotrainee.com/thread-1484-1-1.html
mkdir trim_out
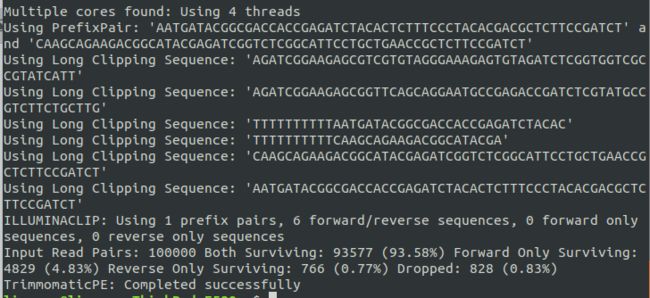
java -jar ~/BioSofts/Trimmomatic-0.38/trimmomatic-0.38.jar PE -phred33 test_7942raw_1.fq.gz test_7942raw_2.fq.gz ./trim_out/output_forward_paired.fq.gz ./trim_out/output_forward_unpaired.fq.gz ./trim_out/output_reverse_paired.fq.gz ./trim_out/output_reverse_unpaired.fq.gz ILLUMINACLIP:/home/lizeguo/BioSofts/Trimmomatic-0.38/adapters/TruSeq2-PE.fa:2:30:10 SLIDINGWINDOW:5:20 LEADING:20 TRAILING:20 MINLEN:75