https://doi.org/10.1016/j.semcdb.2015.11.013
Pre:
基因的转录表达精确复杂,对细胞的生长,发育,分化至关重要!人们对于基因的初步认识是线性的,它上面有编码区,非编码区。有启动子,增强子,绝缘子等元件。在细胞核中,线性的DNA分子会折叠形成的高级结构。在这个折叠的过程中,调控元件区域(如增强子,绝缘子等区域)会靠近启动子区域从而导致基因表达上调。伴随着3D基因组的不断研究,发现空间结构对于基因的表达调控有非常重要的作用。
Q:为什么空间结构对于基因的表达调控很重要呢?
空间构象发生了改变,导致启动子和增强子等调控元件靠近,基因的表达发生了变化
(图片来源:基于Hi-C技术哺乳动物三维基因组研究进展 DOI: 10.16288/j.yczz.18-317 )
在形成DNA折叠的过程中,有很多蛋白质发挥了重要的作用。染色质高度相互的domain(结构域)被称为 TADs(topologically associating domains)。很多的证据表明,这些TADs横跨了很多domain(比如说DNA复制时期Domain,进化重排domian等)。帮助TAD形成的蛋白,被称为架构蛋白(arichitecture protein, AP)当把敲出这些架构蛋白以后,基因的表达将会受到影响。具体一点,基因的表达调控和这些架构蛋白的数量,蛋白的结合方向等有关系呢?这些成为了科学家们的关注重点。
这篇文章主要是先回顾了果蝇和脊椎动物的架构蛋白情况,然后讨论了蛋白缺失后,对染色质的互作和基因转录的影响。最后,讨论了结构蛋白对3D基因组模型的作用(从架构蛋白的量,性质和结构蛋白结合的方向角度出发)
1:架构蛋白(Architectural protein)
在哺乳动物中,CTCF(CCCTC-bing factor)蛋白是主要被研究的架构蛋白。但是在果蝇里面,有很多结构蛋白被研究的很清楚了,如Associated Factor 32 (BEAF-32), Suppressor of Hairy-wing [Su(Hw)], Transcription Factor IIIC (TFIIIC), Z4 (also called Putzig), Insulator binding factor 1 and 2 (Ibf1 and Ibf2), Pita,Zn-finger Protein Interacting with CP190 (ZIPIC) 。当这些结构蛋白结合到蛋白结合位点以后。会招募一系列相关的蛋白如(CP190, Modifier of mdg4 [Mod(mdg4)], Rad21, CAP-H2,等等)。他们会形成一个复合物。被称为APBS(位点+架构蛋白+结合因子)
在哺乳动物中,CTCF和cohesin这两个架构蛋白最主要。除了这两个以外,还有别的比如说YY1,TFIIIC(果蝇)等。还有一些转录因子(POZ-Zn fingre, MAZ,ZNF143等等)对染色质的三维结构有一定的作用。一些锌指结构的蛋白通过识别一些特殊的位点对高级结构也有一定的影响。
2:通过架构蛋白介导远距离互作
通过Hi-C和ChIA-PET实验,可以在不同的细胞系上进行实验发现,CTCF和cohesin在TAD的边缘富集。但是很多证据表明,在哺乳动物中,不仅仅是通过CTCF和cohesin去介导远距离相互作用,在GM12878中,只有30%的互作是由CTCF介导的,54%的互作只有一边有CTCF。此外,还有Rad21/smc1a(cohesin复合物亚基)介导的远距离互作也只有41%、35%。Znf143, YY1, 和 TFIIIC,一些tRNA 基因,SINE元件都可以在远距离调控上发挥作用。
在细胞的分化中,TAD的形成还是很稳定的,但是架构蛋白的结合位点是灵活的。有意思的是CTCF的在全基因组上的位点是保守的。而且,一些多能转录因子。比如说Oct4,Nanog,Klf4等,如果把这些多能转录因子敲除,导致了细胞内多能行的染色质互作减少。说明,多能转录因子对的启动子-增强子的互作有一定的影响,可以帮助招募cohesin,从某种程度上来说,可以稳定interaction。
3:架构蛋白的删除可以导致异位染色质接触(ectopic chromatin interactions )影响转录
通过RNAi的方法,把CTCF敲除,通过Hi-C可以看到TAD内部的互作减少,TAD之间的互作增加了。举个例子,当把X染色体上的CTCF的区域删掉(58Kb)导致了染色质间的相互作用增强了。此外,用CRISPER-Cas9技术,可以特定区域的CTCF敲除,比如把Hoxa6(活跃表达)和Hoxa7(沉默)的CTCF敲除,发现Hoxa7的基因被激活而且通过4C技术发现了一个新的相互作用。此外,还有通过把TAD边界的CTCF删除导致多指症的情况,都说明CTCF作用非常重要。
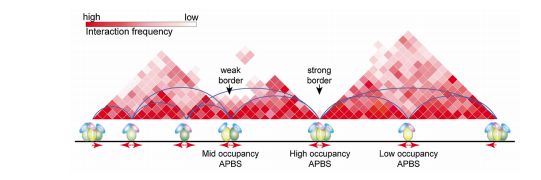
4 占据的位置可能决定了其功能----建立特殊的3D结构
APBS占据比较少的地方,可能是sub-TAD的边界或者是TAD互作弱的地方。当APBS占据比较多的地方(strong border)显示TAD的界限明晰
在TAD的界定上,一直是基于它们的相互作用的强弱来看的。TAD(热图)显示了染色质互作之间的接触频率。为了明确TAD/sub-TAD,界定一个border是非常必要的。TAD的APBS的强度高sub-TAD,除了CTCF,还有一些CTCF的同源蛋白BORIS等,和CFCF会共定位在一些位点上。帮助形成3D结构
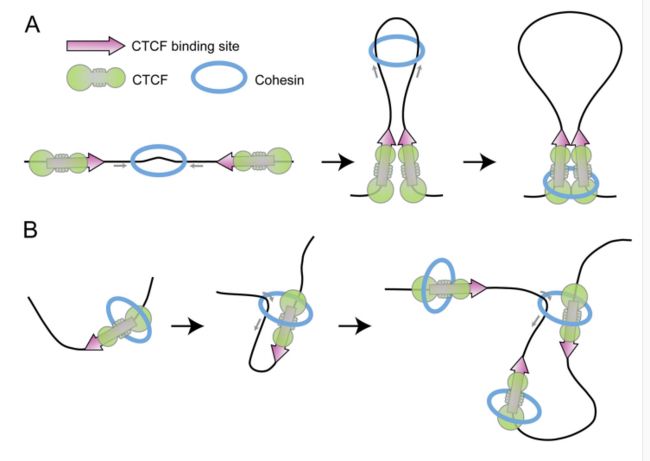
5:架构蛋白的方向指导了loop的形成
哺乳动物中,高级结构的形成依赖于CTCF的结合方向,但是在果蝇中没有这种情况出现。在哺乳动物中,绝大部分的CTCF的方向是相对的。(90%,GM12878数据)而且在其他的细胞系中也观察到了类似的情况。 但是也有同向的CTCF,比如说在小鼠中, Igh 位置上, intergenic control region1 (IGCR1)有反向的CTCF,它需要依赖于其他的同向的CTCF构成loop。这种反向CTCF形成loop的模式在斑马鱼,小鼠等其他物种是保守的。
在人protocadherin(Pcdh)基因簇中,CTCF在调节增强子-启动子选择中起重要作用。因为它随机负责Pcdh的表达情况。(既然CTCF那么重要,如果把一些特定的CTCF敲除会如何?)当把CTCF敲出以后,在用4C去看图谱的时候发现,相向的CTCF会受到影响。(同向和反向的不会)
6:loop 挤压模型解释了CTCF的方向性对LOOP的形成作用
一个比较有意思的问题是这些CTCF是如何作用于3D结构的形成的呢?有2种解释的模型机制。
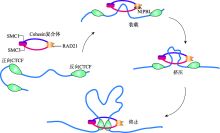
模型一:当cohesin结合上去的时候,会挤压DNA形成环,慢慢伴随着cohesin的移动,这个环会形成。直到遇到了两个相向的CTCF。就停止了
模型二:当CTCF结合到了特定的DNA序列上时就导致了DNA开始扭曲成环,然后慢慢移动,直到碰到了相向的CTCF环,此外,这个模型是不能够去解释同向的CTCF成环情况。
当然,这两个模型未被完全证实,还存在一些缺陷。需要更多的试验机制去证明和实现。
Last:
在哺乳动物中,CTCF和cohesin到底是如何形成高级结构,如何去调控的呢?机制未明,有待发现!