MCScanX是检测基因共线性和进化分析的常用工具之一,2012发表至今引用数200+,作者之一的唐海宝老师是国内植物基因组学生信分析、软件开发领域的大拿,在学习使用MCScanx之前推荐先看看他08年介绍gene synteny和collinearity概念的science文章以及MCScanX软件算法文章。
Tang H, Bowers J E, Wang X, et al. Synteny and Collinearity in Plant Genomes[J]. Science, 2008, 320(5875):486-488.
Wang Y, Tang H, DeBarry JD, Tan X, Li J, Wang X, Lee TH, Jin H, Marler B, Guo H, Kissinger JC, Paterson AH. (2012) MCScanX: a toolkit for detection and evolutionary analysis of gene synteny and collinearity. Nucleic Acids Res, 40(7): e49.
好,接下来具体讲讲MCScanX的使用
1. 进入MCScanX官网http://chibba.pgml.uga.edu/mcscan2/#tm
首先是关于版本的介绍:
可以看到MCScanX是MCScan的升级版本:
1)用法更简单,只需要blastp m8格式的比对文件和经过简单处理的gff作为输入文件即可;2)参数更多元,可以设置gap的阈值;3)输出文件中有html网页版的展示,可以看到第一列是duplicaiton depth,tandem genes用红色标出;4)优化了算法,缓和了物种间不同gene densities的影响
右边是历史版本的优化情况,其中13年发布的MCScanX-transposed是用来检测基因组内或组间的transposed gene重复
2. 下载及安装
官网都给了链接,也可以参考biochen的文章,很详细使用MCScanX分析基因组共线性区块
unzip MCscanX.zip
cd MCScanX
make
Manual http://chibba.pgml.uga.edu/mcscan2/documentation/manual.pdf
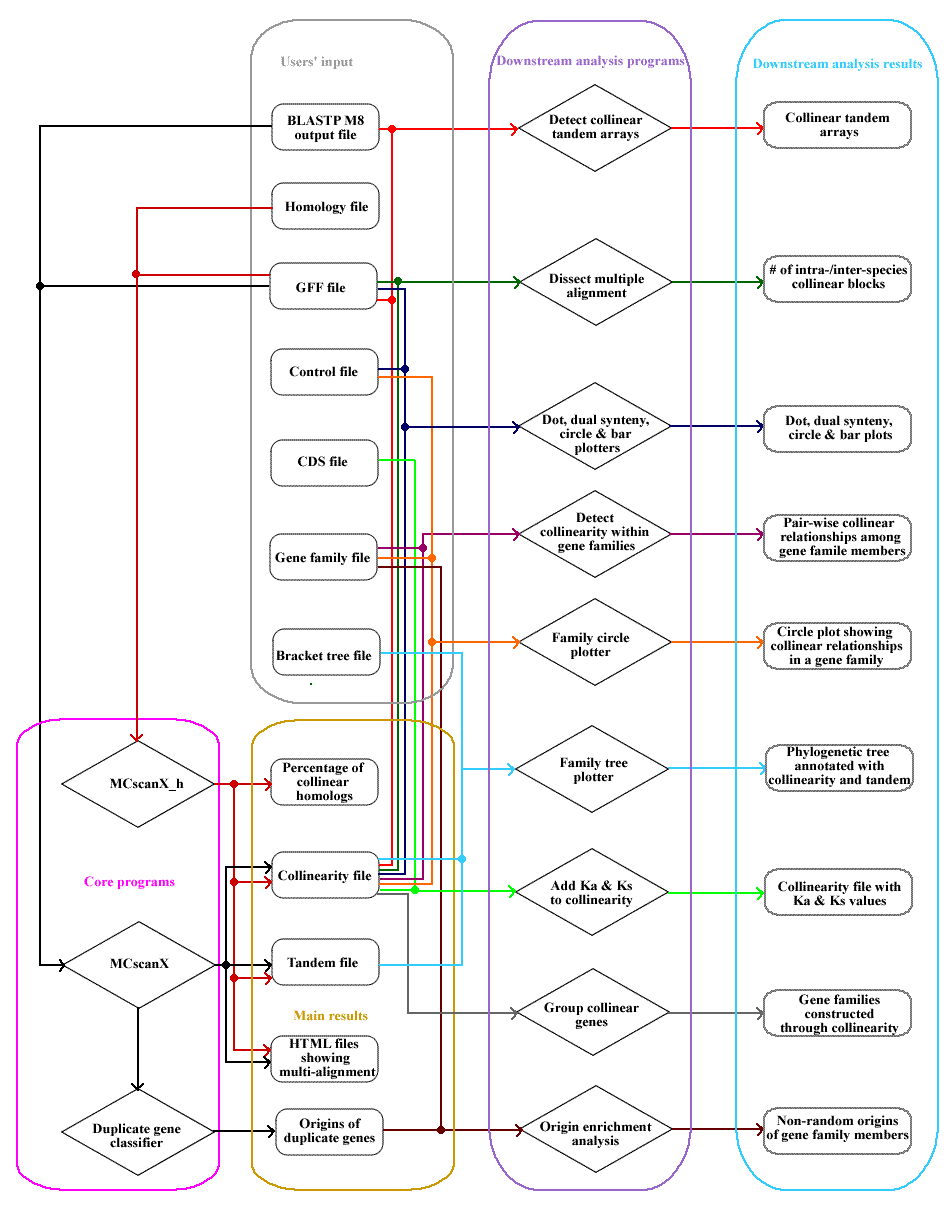
MCScanX、MCScanX_h、duplicate_gene_classifier这三个是核心程序,downstream_analyses中包含12个下游分析程序,可以画图构树,还是很方便很强大的
3.具体使用方法
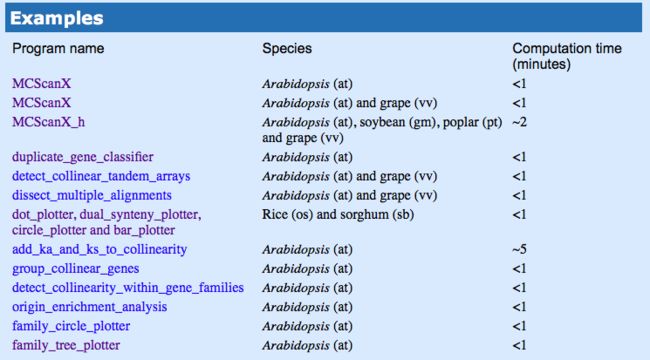
第一个MCScanX是拟南芥单一物种内找共线性gene区块的例子,还可以用 duplicate_gene_classifier在单一基因组内找重复序列,并区分singleton, dispersed, proximal, tandem, WGD/segmental五种类型。第二个MCScanX是拟南芥和葡萄物种之间找共线性,MCScanX_h和MCScanX类似,只是输入文件稍有不同,如果已知物种间的homology关系,可以直接用MCScanX_h。后面是下游分析的java程序,每个程序点击去可以看到实例
下面以最常用的多物种MCScanX找共线性使用为列:
第一步 blastp(protein-protein BLAST)比对
注意!这里是找at和vv两个基因组组内和组间的共线性,因为想同时知道物种内和物种间的共线性,所以在blast之前把at和vv的基因组facat到一起,既做database,又做query,如果只想知道组间的共线性,那么就任取一个基因组为database,另一个做query
合并 cat at.fa vv.fa >>all.fasta
建库 makeblastdb -in all.fa-dbtype prot -parse_seqids -out all (-logfile allpep.log -title all)
蛋白比对 blastp -query all.fa -db all -out at_vv.blast -evalue 1e-10 -num_threads 16 -outfmt 6 -num_alignments 5
小tips:blast这一步是限速步骤,可以把all.fasta文件cut成多份,同时并行跑节省时间
注意!亲自验证该软件最多只能做5个物种的共线性。。。不管输入再多物种结果只有五个!!
第二步 运行MCScanX
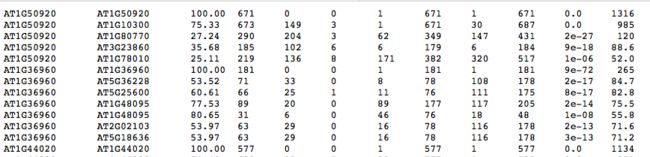
输入文件只有两个,一个是上一步blast得到的at_vv.blast文件,格式如下:
还有一个是at_vv.gff文件,不过跟一般的九列gff文件不同,这里是缩略版本,可以用awk得到,第一列是物种名和染色体编号,第二列是基因号,第三列是起始位置,第四列是终止位置(用tab分割)
准备好这两个文件之后,输入命令行
MCScanX at_vv
注意:at_vv.gff at_vv.blast和at_vv命名需要一致且在同一个文件夹里
其他参数设置
运行速度快(吐槽一下用Sibelia来做植物基因组实在是太慢了。。)
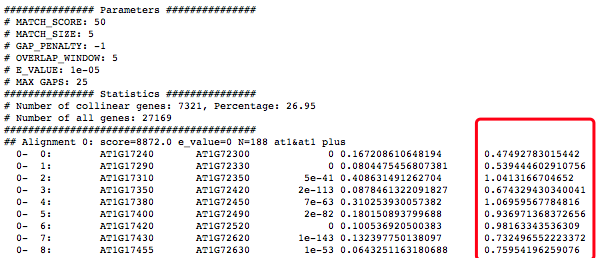
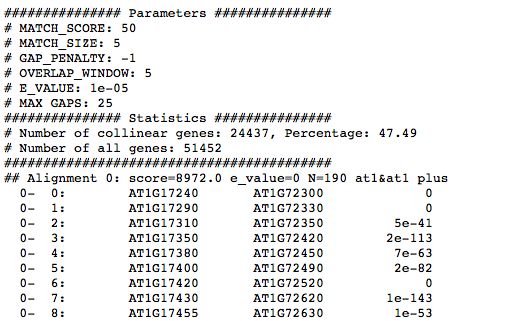
运行成功后得到at_vv.html,at_vv.collinearity,at_vv.tandem输出文件
at_vv.collinearity里记录了共线性信息
可以看到collinear gene的数目和占比以及具体的比对信息
第三步 下游分析及可视化
1、常用的下游分析有:
duplicate_gene_classifier XX (0:singleton(非重复基因)1:dispersed(不是2,3,4的其它重复)2:proximal(染色体附近的重复,但是不相邻)3:tandem(串联重复)4:WGD/segmental(在共线性区域的共线性基因))
dissect_multiple_alignment -g XX.gff -c XX.collinearity -o XX.dis
group_collinear_genes.pl -i XX.collinearity -o XX.collinear.groups
2、可视化:软件自带的分析包不能调颜色,所以推荐用python版的MCscan(JCVI 包),具体参考我的另一篇文章其实MCScanX画图也可以很好看
绘图瞬间高大上
这里直接使用下游dot_plotter, dual_synteny_plotter, circle_plotter和bar_plotter 4个java包更便捷
gff和collinearity是上一步的输出,还需要编辑一个control文件,设置需要展示的染色体信息(和gff的第一列一致)
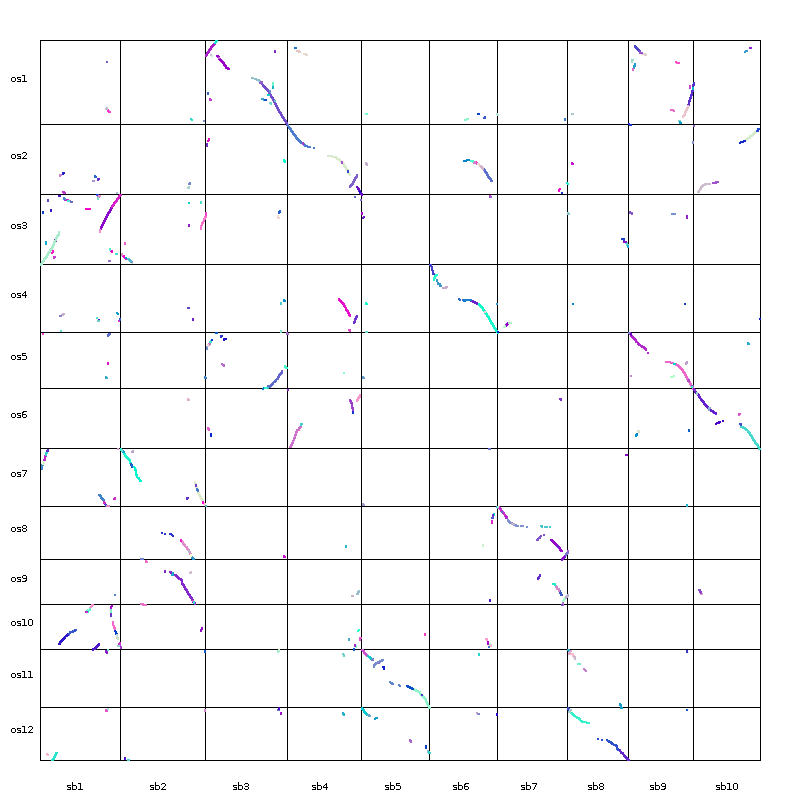
1.dot_plotter
java dot_plotter -g at_vv.gff -s at_vv.collinearity -c dot.ctl -o dot.PNG
dot.ctl
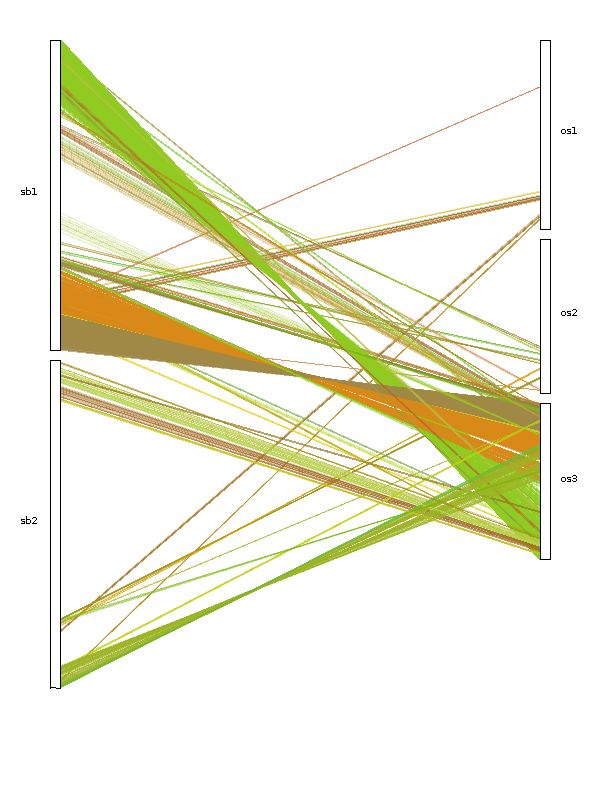
2.dual_synteny_plotter
java dual_synteny_plotter -g at_vv.gff -s at_vv.collinearity -c dual_synteny.ctl -o dual_synteny.PNG
dual_synteny.ctl
3.circle_plotter
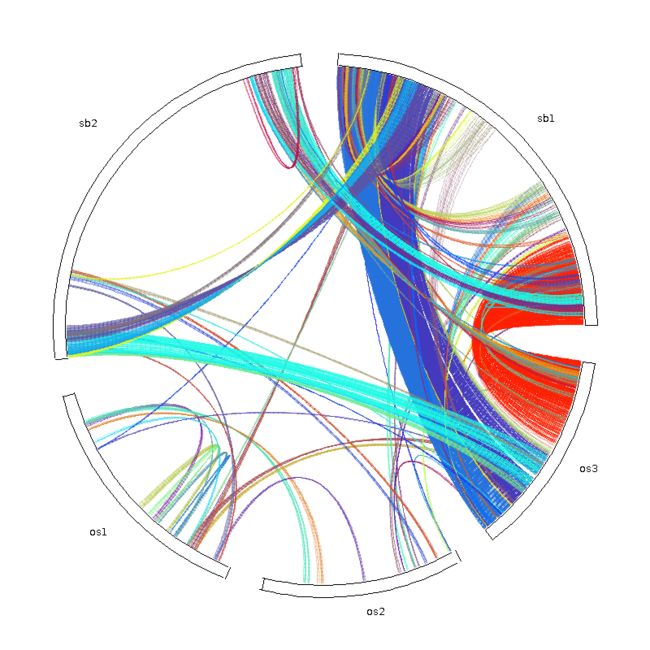
java circle_plotter -g at_vv.gff -s at_vv.collinearity -c circle.ctl -o circle.PNG
circle.ctl
4.bar_plotter
java bar_plotter -g at_vv.gff -s at_vv.collinearity -c bar.ctl -o bar.PNG
bar.ctl
第四步 其他分析
duplicate_gene_classifier、detect_collinear_tandem_arrays、dissect_multiple_alignments对结果做进一步分析,借助其他相关信息,还可以做
origin_enrichment_analysis,family_tree_plotter,add_ka_and_ks_to_collinearity等
小结
MCScanX官网界面友好,亲测好评,后面画图如果想自己调试改颜色啥的,还是学一下circos的使用吧~
有问题可以联系作者
Please be sure to direct your questions to [email protected]