本文不得转载。
摘要:对中国科学技术大学(USTC)2016年波谱解析题进行分析与解答,旨在帮助备考者。同时简要介绍了qcc-NMR的应用。
题目:
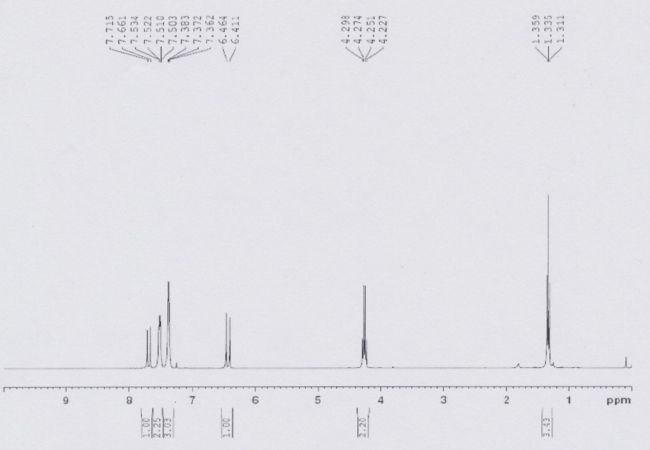
1.某化合物的分子式为C10H12O2,下面是其核磁共振氢谱(所用核磁共振仪的频率为300 MHz,溶剂为CDCl3;谱图中上面数据为化学位移,下面数据为峰面积)
(1) 给出该化合物的不饱和度。
(2)给出四重峰δ4.26的偶合常数(J)。
(3)推测该化合物的结构(如有立体化学,请注明)。
解答:
(1)不饱和度的计算十分容易,这里不加赘述,答案为6。
(2)耦合常数的计算=化学位移的差值*核磁的兆数。题目里告诉我们是四重峰,每个小峰之间的距离都是相等的,即它们只有一个耦合常数(不考虑仪器测量误差的情况下)。取任意相邻的两个小峰计算得到的耦合常数都是相等的。但是我们这里稍微验证下:
第一个小峰与第二个小峰之间的耦合常数:
第二个小峰与第三个小峰之间的耦合常数:
第三个小峰与第四个小峰之间的耦合常数:
计算完发现有一个是6.9HZ,这里我们认为是仪器测量的误差,可以不管。那么最终结果是J=7.2HZ,不要忘掉了单位。
(3)观察图谱,在化学位移为4.2和1.3处分别有(q,2H)和(t,3H),十分符合乙基的特征。大胆猜测该物质有乙基。此外,化学位移向低场移动,说明和吸电子计算相连,考虑到体系中有氧原子,可以认为是一个乙氧基。体系中共有两个氧原子,可以认为这里是一个酯基结构。
接着看到芳香区有不少氢原子,结合高达6的不饱和度,该体系必然含有苯环。但是化学位移6~9共有7个氢原子,这里的苯环最多只有5个氢原子,那这是怎么回事呢。首先排除化学位移6~7的一个氢原子不在苯环上,因为苯环氢的化学位移一般不会这么低,但是如此之高的化学位移总要有个归属,除了苯环,烯基氢的化学位移也相对较高,可以认为化学位移6~7的氢原子属于烯烃。不过我们知道,乙烯的氢的化学位移5左右,说明该双键上连接有吸电子基团——刚才的酯基结构。
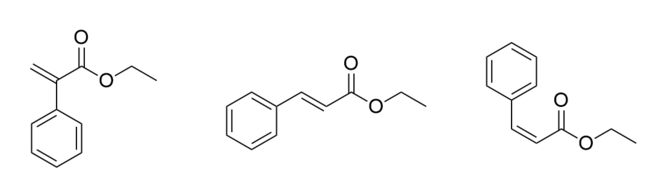
至此,问题基本解决:一个双键上连接有苯环和酯基,可能有如下几种结构:
第一个应当首先排除,因为从图中看,烯基氢的化学位移一个在6~7,一个在7.8~8,相差很大。按照第一个结构,二者的化学位移应当很接近。
那么问题就在于剩下的两个结构的区别了,烯烃顺反异构可以通过计算双键氢原子的耦合常数来判断。考察6.464~6.411的两个峰,其耦合常数:。相当之大,我们知道顺式烯烃的耦合常数一般在7~11HZ,反式烯烃的耦合常数一般在12~18HZ,所以这里应该是反式烯烃,即中间一个结构。这是由于反式烯氢的两轨道电子云交盖程度比顺式烯氢的大导致的。
事实上,第(2)小问计算得到的耦合常数7.2HZ也很常见,大家可以记住它。
大家还可以思考一下,化学位移7.8~8的那个氢原子是靠近苯环的还是远离苯环的,以及原因(靠近苯环的)。
我们可以使用量子化学计算NMR 参数(qcc-NMR)。例如计算化学位移、耦合常数等。这里简要介绍一下计算耦合常数J计算时的方法。一般来说需要有较紧的高斯函数来加强描述,大家可以在Basis Set Exchange (BSE)网站下载IGLO-III或pcJ基组,这两个基组的表现较好,不过需要学习自定义基组的知识。在关键词中加上NMR=spinspin即可计算耦合常数(spin即自旋的意思)。这里以乙酸乙酯的计算结果为例。
完成计算后,查看输出文件,翻到“Total nuclear spin-spin coupling J (Hz)”一栏(见附录),直接查看以HZ为单位的总耦合常数。这里相当于一个n*n的矩阵,计算了任意两个核之间的耦合常数。以酯基上的亚甲基氢和甲基氢的耦合为进行分析。分别查看6H-9H,6H-10H,6H-11H的耦合常数,依次为14.4370,1.96294,5.09961,由于甲基时可以旋转的,应当对这三个数值取平均,结果为7.16HZ,大约为7.2HZ。与之前题目里的实验数据完全一致,说明计算结果是很可靠的。
为何这个数值不直接是7.2HZ,而是3个不同的数值呢?实际上由Karplus公式(见任何一本波谱解析书籍)可以计算耦合常数与二面角的关系,甲基上的三个氢与6号氢有完全不同的三个二面角,所以耦合常数相差巨大。不过最终宏观上体现出来的是平均值7.2HZ。
附录:
```
Total nuclear spin-spin coupling J (Hz):
1 2 3 4 5
1 0.000000D+00
2 0.124472D+03 0.000000D+00
3 0.132934D+03 -0.117515D+02 0.000000D+00
4 0.124477D+03 -0.168693D+02 -0.117480D+02 0.000000D+00
5 0.200221D+01 0.562956D+00 0.265548D+01 0.563680D+00 0.000000D+00
6 0.855502D-01 0.412612D+00 0.160212D+00 -0.486707D+00 0.140714D+03
7 0.854848D-01 -0.486922D+00 0.160451D+00 0.414829D+00 0.140710D+03
8 -0.129839D+00 0.149726D+00 0.284824D-03 0.149969D+00 0.392041D+02
9 0.196045D+00 -0.351586D-01 0.722496D-01 0.325557D-01 -0.584264D+01
10 0.315463D+00 -0.267275D-01 0.692497D-01 -0.267284D-01 0.342190D+01
11 0.196255D+00 0.328098D-01 0.726703D-01 -0.351391D-01 -0.584183D+01
12 -0.307064D+01 -0.205062D+01 -0.699984D+00 -0.205117D+01 0.112028D+00
13 0.553711D+02 -0.519051D+01 -0.437854D+01 -0.518984D+01 -0.155003D+01
14 -0.423786D+01 0.853568D+00 -0.714745D+00 0.853533D+00 0.241942D+02
6 7 8 9 10
6 0.000000D+00
7 -0.882273D+01 0.000000D+00
8 -0.205187D+01 -0.205267D+01 0.000000D+00
9 0.144370D+02 0.509730D+01 0.126098D+03 0.000000D+00
10 0.196294D+01 0.196502D+01 0.122368D+03 -0.123455D+02 0.000000D+00
11 0.509961D+01 0.144370D+02 0.126100D+03 -0.105430D+02 -0.123448D+02
12 -0.276409D-01 -0.270016D-01 0.564641D-01 0.578701D-01 -0.145799D+00
13 0.964477D+00 0.966106D+00 0.435141D+01 -0.308230D+00 0.169684D+01
14 -0.215744D+01 -0.215560D+01 -0.945946D+01 -0.186961D+01 -0.151932D+02
11 12 13 14
11 0.000000D+00
12 0.578795D-01 0.000000D+00
13 -0.308331D+00 0.250752D+02 0.000000D+00
14 -0.186710D+01 0.792811D+00 0.346717D+02 0.000000D+00
```