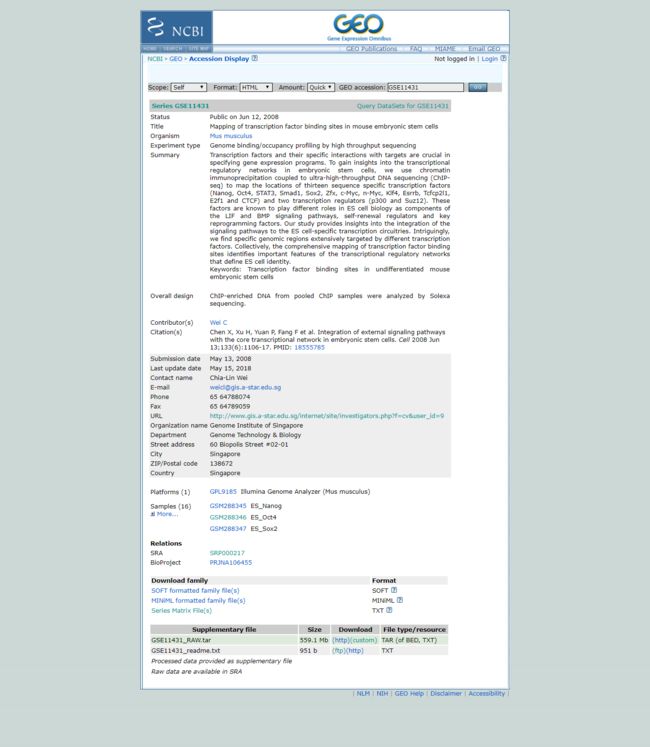
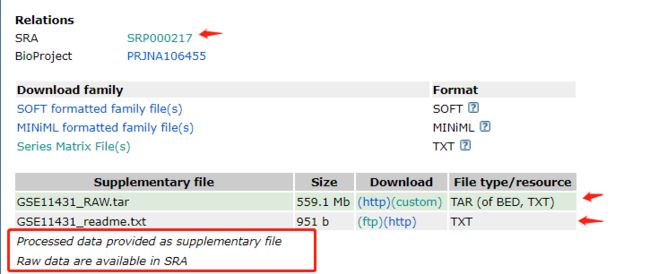
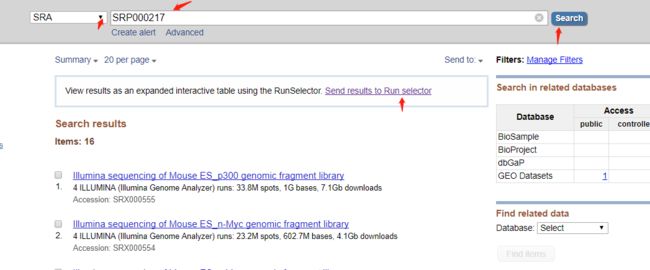
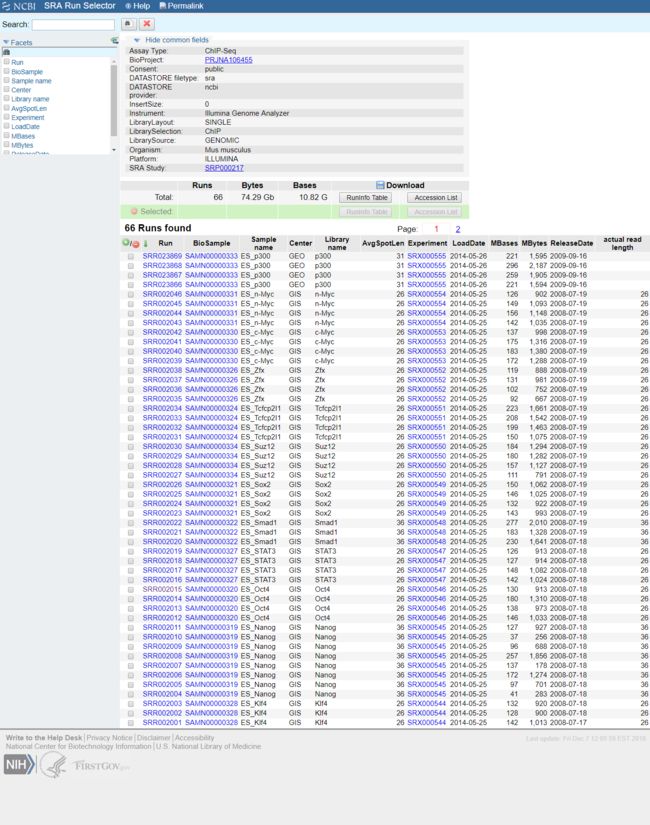
- Halcon 3月License 持续更新
lkasi
haclon计算机视觉c++
大家新年快乐啊!3月License:仅仅支持以下版本24.05和24.1111月:呜呜呜呜,才装的23,又要装24了12月:yeah!我是24.05!!!1月:新的一年,24还是可以用大家春节快乐呀2月:新的一年,24依然可以用3月:24.0524.11依然可以用!!!链接:https://pan.baidu.com/s/1GmymNfYVFlokESK2r1HnmA?pwd=agey提取码:ag
- 零基础必看!CCF-GESP Python一级考点全解析:运算符这样学就对了
奕澄羽邦
python开发语言
第一章编程世界的基础工具:运算符三剑客在Python编程语言中,运算符如同魔法咒语般神奇。对于CCF-GESPPython一级考生而言,正确掌握比较运算符、算术运算符和逻辑运算符这三大基础工具,就相当于打开了数字世界的大门。这三个运算符家族共同构成了程序逻辑的核心骨架,其灵活组合能实现从简单计算到复杂判断的多样功能。1.1运算符分类图谱算术运算符:负责数字间的数学运算(+-*/%)比较运算符:用于
- 机器学习(Machine Learning)
七指琴魔御清绝
大数据学习
原文链接:http://blog.csdn.net/zhoubl668/article/details/42921187希望转载的朋友,你可以不用联系我.但是一定要保留原文链接,因为这个项目还在继续也在不定期更新.希望看到文章的朋友能够学到更多.《BriefHistoryofMachineLearning》介绍:这是一篇介绍机器学习历史的文章,介绍很全面,从感知机、神经网络、决策树、SVM、Ada
- Python 字符串操作
iteye_13776
PythonPythonCC++C#
Python截取字符串使用变量[头下标:尾下标],就可以截取相应的字符串,其中下标是从0开始算起,可以是正数或负数,下标可以为空表示取到头或尾。#例1:字符串截取str='12345678'printstr[0:1]>>1#输出str位置0开始到位置1以前的字符printstr[1:6]>>23456#输出str位置1开始到位置6以前的字符num=18str='0000'+str(num)#合并字
- ES6解构赋值详解
漫天转悠
ES6es6前端ecmascript
ES6解构赋值详解ES6解构赋值是JavaScript语言的一项强大特性,它允许从数组或对象中提取数据,并将其赋值给变量。这一特性不仅简化了代码,提高了可读性,还增强了代码的灵活性。本文将详细介绍ES6解构赋值的基本概念、语法、应用场景以及一些高级用法。1.基本概念解构赋值是对赋值运算符的扩展。它允许按照一定的模式,从数组或对象中提取值,并赋值给变量。这种语法使得从复杂数据结构中提取数据变得更加简
- 【PX4】Ubuntu20.04安装PX4教程
davidson1471
PX4git无人机linuxubuntu
*建议早上安装*1.下载以往版本从github上clone源码gitclonehttps://github.com/PX4/PX4-Autopilot.git进入PX4-Autopilot文件夹cdPX4-Autopilot查看当前分支,位于origin/maingitstatus查看所有远程分支,带release的gitbranch-r|grep"release"切换到发行分支v1.12gitc
- ES6解构语法详解
勇敢小陈
javascriptvue.js前端es6
ES6的解构语法简化了我们开发过程中复杂的取值过程,可能你在a.b.c.d的时候别人早就把值一开始就取出来了,使代码更加简洁。下面开始进行语法的详解。一、单层解构单层解构constearth={people:'人类',animal:'动物'}const{people,animal}=earthconsole.log(people,animal);//人类动物单层解构并更改变量名称consteart
- Go 语言使用Protobuf 进行序列化详解
尘鹄
Go语言学习之路golang开发语言后端rpcgo
文章目录Go语言使用Protobuf进行序列化详解1.Protobuf是什么?2.安装Protobuf及Go依赖3.编写.proto文件4.实现序列化和反序列化Go语言使用Protobuf进行序列化详解1.Protobuf是什么?以下是Protobuf官方中文文档的概述:Protobuf(ProtocolBuffers)是一种语言中立、平台中立的可扩展机制,用于序列化结构化数据。它类似于JSON,
- Qt 串口类QSerialPort 使用笔记
一对一答疑的编程作家朱文伟
qtqt笔记开发语言
Qt串口类QSerialPort使用笔记虽然现在大多数的家用PC机上已经不提供RS232接口了。但是由于RS232串口操作简单、通讯可靠,在工业领域中仍然有大量的应用。Qt以前的版本中,没有提供官方的对RS232串口的支持,编写串口程序很不方便。现在好了,在Qt5.1中提供了QtSerialPort模块,方便编程人员快速的开发应用串口的应用程序。本文就简单的讲讲QtSerialPort模块的使用。
- Towards Multimodal Large-Language Models for Parent-Child Interaction: A Focus on Joint Attention
UnknownBody
LLMDailyMultimodal语言模型人工智能大数据
摘要共同注意是儿童早期语言发展的关键组成部分,也是亲子互动有效性的重要指标。然而,目前对共同注意的检测和分析研究仍然有限,尤其是在多模态大语言模型(MLLMs)方面。本研究通过分析由两位语言病理学家标注的26段亲子互动视频,评估了多模态大语言模型理解共同注意的能力。这些标注识别出了共同注意程度高和低的片段,作为评估模型解释能力的基准。我们的研究结果显示,由于当前的多模态大语言模型对儿童发起的眼神交
- web前端期末大作业:婚纱网页主题网站设计——唯一旅拍婚纱公司网站HTML+CSS+JavaScript
IT-司马青衫
前端课程设计html
静态网站的编写主要是用HTMLDⅣV+CSSJS等来完成页面的排版设计,一般的网页作业需要融入以下知识点:div布局、浮动定位、高级css、表格、表单及验证、js轮播图、音频视频Fash的应用、uli、下拉导航栏、鼠标划过效果等知识点,学生网页作业源码,制作水平和原创度都适合学习或交作业用,记得点赞。精彩专栏推荐【作者主页——获取更多优质源码】【web前端期末大作业——毕设项目精品实战案例(1
- 在 ASP.NET Core WebAPI 中使用 JWT 验证
.NET跨平台
ASP.NETCoreWebAPIIdentityServer4OAuth2.0协议.NETCOREWEBAPIJWT
为了保护WebAPI仅提供合法的使用者存取,有很多机制可以做,透过JWT(JSONWebToken)便是其中一种方式,这篇示范如何使用官方所提供的System.IdentityModel.Tokens.Jwt扩充套件,处理呼叫API的来源是否为合法的使用者身分。顺道一提,要产生JWTToken有很多套件可以帮助开发者快速建立,JWT这个NuGet套件就是其中一个,但这裡我使用官方所提供的Syste
- API身份验证使用JWT的.NET实现
雨夜思绪~静谧思考
.net
API身份验证使用JWT的.NET实现在现代的应用程序开发中,API身份验证是一项至关重要的任务。JWT(JSONWebToken)是一种常用的身份验证机制,它使用JSON格式表示身份验证信息,并使用签名进行验证。在.NET平台上,我们可以使用一些库来实现JWT身份验证。本文将介绍如何使用.NET来进行API身份验证并使用JWT作为身份验证机制。引入依赖项首先,我们需要在我们的项目中引入相关的依赖
- 关联规则算法:揭秘数据中的隐藏关系,从理论到实战
秋声studio
机器学习算法详解关联规则算法数据挖掘Apriori算法FP-Growth算法大数据优化数据预处理增量式更新
引言在当今数据驱动的时代,如何从海量数据中挖掘出有价值的信息成为了各行各业的核心挑战。关联规则算法作为数据挖掘领域的重要工具,能够帮助我们发现数据中隐藏的关联关系,从而为决策提供支持。无论是电商平台的商品推荐,还是医疗领域的疾病诊断,关联规则算法都展现出了强大的应用潜力。本文将从基础概念出发,逐步深入探讨关联规则算法的核心原理、经典算法及其优化策略。无论你是数据挖掘的初学者,还是希望进一步了解关联
- 【Go语言圣经1.1】
Pyroyster
golang开发语言后端
目标学习Go的编译方式、包的组织方式以及工具链的统一调用方式概念与定义packageGo语言通过包来组织代码。包类似于其它语言的库librarries或模块modules,每个包通常对应一个目录,目录中的所有.go文件都属于同一个包。特殊的main包:当代码使用packagemain声明时,表示这是一个可独立执行的程序而非一个库。程序的执行入口就是main函数import通过import语句,编译
- Spike Neural Network Introduction and Research Directions
Debug_Snail
SNNNeuralnetwork人工智能AIGC
1.SNNs是一类神经网络,其中的神经元通过脉冲(spikes)来传递信息,而不是像传统的人工神经网络中那样使用实数值激活。SNNs更接近生物学上的神经系统,因为生物神经元也是通过电信号脉冲来传递信息的。与传统神经网络相比,SNNs具有以下几个特点:更低的功耗-因为只在发生脉冲时才激活神经元,所以整体功耗会比传统神经网络低很多。这使得SNNs很适合应用在对功耗要求非常严格的场景,如边缘计算。时序编
- 一文理清:阿里系数据中台-数据治理工具集(傻傻也能分清楚)
Debug_Snail
HadoopBigData技术工具人工智能hadoop数据仓库
阿里云提供的大数据与数据分析产品种类较多,各产品的定位和核心功能有所不同。以下是对DataWorks、MaxCompute、Dataphin、AnalyticDBforMySQL(ADB)、QuickBI、EMR的详细梳理。一、核心产品定位与功能DataWorks定位:一站式大数据开发治理平台,提供数据集成、开发、调度、治理、服务等全链路能力。核心功能:数据集成:支持异构数据源(如数据库、OSS、
- 灵犀X2:人形机器人的新篇章
Anima.AI
机器人
简介灵犀X2是智元机器人推出的最新款人形机器人,很可能是其前代产品灵犀X1的升级版本。灵犀X1作为一款开源的模块化机器人,其机械设计和软件代码完全公开,全球开发者都可以参与优化和创新。这款机器人身高130厘米,体重33公斤,具备34到44个自由度(DegreesofFreedom,DoF,即关节活动范围),能够执行轻型任务,如端茶送水、整理房间等。灵犀X2在继承这些特性的基础上,可能进一步提升了动
- 如何注册下载币安
FAC306
区块链web3
安卓注册下载链接如下,苹果下载需要更换IDwww.marketwebb.click/zh-CN/join?ref=TLMVPKLP
- 哪个AI论文生成助手好用?5 款AI论文工具深度评测
AI论文图鉴
人工智能
2025年,AI论文写作工具如雨后春笋般涌现,迅速在学术圈走红。身为一个常被论文写作困扰的“懒人”,我对这些工具的实际表现充满好奇。于是,我亲测了五款当下国内外最热门的AI论文写作助手,从功能、交互、写作水平、写作效率等维度进行全面评测,结果令人惊喜。相信这篇文章能为仍在观望的你带来新的启发与认识。这次测评,我挑选了五款极具代表性的AI工具,以“基于大语言模型的医疗诊断研究”为主题,看它们如何大显
- 大语言模型(LLM)入门学习路线图_llm教程,从零基础到精通,理论与实践结合的最佳路径!
AGI学习社
语言模型学习人工智能LLM大模型大数据自然语言处理
Github项目上有一个大语言模型学习路线笔记,它全面涵盖了大语言模型的所需的基础知识学习,LLM前沿算法和架构,以及如何将大语言模型进行工程化实践。这份资料是初学者或有一定基础的开发/算法人员入门活深入大型语言模型学习的优秀参考。这份资料重点介绍了我们应该掌握哪些核心知识,并推荐了一系列优质的学习视频和博客,旨在帮助大家系统性地掌握大型语言模型的相关技术。大语言模型(LargeLanguageM
- Webpack打包构建流程
码上跑步
webpack前端node.js
webpack的打包构建流程为什么需要打包?在前端有非常多的资源,如css、js、vue、vue、图片、字体等。有些资源需要加工处理1.ts->jsts-loader2.css->css-loader+style-loader3.图片->file-loader+url-loader4.html->html-webpack-plugin需要对产物进行优化optimization(webpack优化配
- webpack
码上跑步
webpack前端node.js
webpack介绍webpack是一个构建工具,实现了模块化管理项目.他的工作方式是用各种loader将各种资源转化为js文件或者对js文件进行压缩编译亦或对静态资源进行处理.官网:webpack由来模块化存在一些问题1.ESM的兼容性问题2.模块文件过多,网络请求频繁3.前端的所有资源包括html和css都需要模块化构建工具应运而生,需要一个集编译,模块打包,支持不同的资源的模块打包工具.Web
- Vue初体验
码上跑步
vue.js前端
Vue基础Vue是什么?Vue是javascript的渐进式框架。Vue初识Vue工作时必须要创建一个Vue的实例,并且传入一个配置对象。root容器里的代码是符合html的语法但是新添加了一些Vue语法,在这些地方Vue会自动进行解析。root容器里的代码称为Vue模版。Vue实例和容器是一一对应的。在实际开发中只有一个Vue,配合组件使用。在vue里的插值{{}}内部只要写js表达式就能正常解
- vue脚手架
码上跑步
vue.js前端javascript
Vue脚手架脚手架是官方提供的标准化开发工具。下载配置//全局安装vue的脚手架npminstall@vue/cli-g//在项目目录下开启一个脚手架vuecreate‘项目名’//进入项目目录,直接运行npmrunserve1.vue.js与vue.runtime.xxx.js的区别:(1).vue.js是完整版的Vue,包含:核心功能+模板解析器。(2).vue.runtime.xxx.js是
- AI-大模型中的流式输出与非流式输出
岂不闻
AI人工智能开发语言AI编程
1.前言在大模型API开发中,流式与非流式输出对应着两种不同的数据交互,在代码中stream中通过参数true与false来进行设定。2.流式输出与非流式输出的原理2.1.非流式输出-请求一次响应返回完整数据非流式输出,传统的请求-响应模式,发起请求-等待完整内容生成后一次性返回给客户端。完整性:返回经过处理和验证的完整响应。单次传输:采用标准HTTP请求与响应模式,一次传输所有数据。等待时间:用
- ES6 解构详解
yqcoder
es6前端javascript
一、数组解构1.基本用法可以按照数组元素的顺序将数组中的值提取到变量中。constarr=[1,2,3];const[a,b,c]=arr;console.log(a);//1console.log(b);//2console.log(c);//32.忽略某些元素如果不想提取数组中的某些元素,可以使用逗号占位。const[x,,z]=[1,2,3];console.log(x);//1consol
- JWT在.NET8 Webapi中的使用
Evan.Pei
.net
JWT身份验证在现代Web应用中广泛使用,主要用于安全地传输用户身份信息.1.身份验证(用户登录后,服务器生成一个JWT并返回给客户端。客户端在后续请求中携带该JWT,服务器通过验证JWT来确认用户身份)2.授权(JWT中可以包含用户的角色或权限信息(如role:“admin”)。服务器根据JWT中的信息决定用户是否有权访问特定资源)3.信息交换(JWT可以包含一些非敏感的用户信息(如用户ID、用
- Bug:QT不能生成可执行文件
lkasi
bug
问题描述:为了生成可执行文件,将项目以release方式进行构建,并且在.pro文件中加入TEMPLATE=app#这生成一个exeQMAKE_LFLAGS+=-no-pie并且执行runqmake,生成的仍是sharedlibrary!!!解决方法:将下面代码放在.pro靠下的位置,并且再次执行runqmake,再构建就成功了TEMPLATE=app#这生成一个exeQMAKE_LFLAGS+=
- 机器学习实战——音乐流派分类(主页有源码)
喵了个AI
机器学习实战机器学习分类人工智能
✨个人主页欢迎您的访问✨期待您的三连✨✨个人主页欢迎您的访问✨期待您的三连✨✨个人主页欢迎您的访问✨期待您的三连✨1.简介音乐流派分类是音乐信息检索(MusicInformationRetrieval,MIR)中的一个重要任务,旨在通过分析音频信号的特征,将音乐自动分类到不同的流派(如古典、摇滚、爵士、流行等)。随着数字音乐平台的普及,音乐流派分类技术被广泛应用于音乐推荐、自动标签生成和音乐库管理
- java类加载顺序
3213213333332132
java
package com.demo;
/**
* @Description 类加载顺序
* @author FuJianyong
* 2015-2-6上午11:21:37
*/
public class ClassLoaderSequence {
String s1 = "成员属性";
static String s2 = "
- Hibernate与mybitas的比较
BlueSkator
sqlHibernate框架ibatisorm
第一章 Hibernate与MyBatis
Hibernate 是当前最流行的O/R mapping框架,它出身于sf.net,现在已经成为Jboss的一部分。 Mybatis 是另外一种优秀的O/R mapping框架。目前属于apache的一个子项目。
MyBatis 参考资料官网:http:
- php多维数组排序以及实际工作中的应用
dcj3sjt126com
PHPusortuasort
自定义排序函数返回false或负数意味着第一个参数应该排在第二个参数的前面, 正数或true反之, 0相等usort不保存键名uasort 键名会保存下来uksort 排序是对键名进行的
<!doctype html>
<html lang="en">
<head>
<meta charset="utf-8&q
- DOM改变字体大小
周华华
前端
<!DOCTYPE html PUBLIC "-//W3C//DTD XHTML 1.0 Transitional//EN" "http://www.w3.org/TR/xhtml1/DTD/xhtml1-transitional.dtd">
<html xmlns="http://www.w3.org/1999/xhtml&q
- c3p0的配置
g21121
c3p0
c3p0是一个开源的JDBC连接池,它实现了数据源和JNDI绑定,支持JDBC3规范和JDBC2的标准扩展。c3p0的下载地址是:http://sourceforge.net/projects/c3p0/这里可以下载到c3p0最新版本。
以在spring中配置dataSource为例:
<!-- spring加载资源文件 -->
<bean name="prope
- Java获取工程路径的几种方法
510888780
java
第一种:
File f = new File(this.getClass().getResource("/").getPath());
System.out.println(f);
结果:
C:\Documents%20and%20Settings\Administrator\workspace\projectName\bin
获取当前类的所在工程路径;
如果不加“
- 在类Unix系统下实现SSH免密码登录服务器
Harry642
免密ssh
1.客户机
(1)执行ssh-keygen -t rsa -C "xxxxx@xxxxx.com"生成公钥,xxx为自定义大email地址
(2)执行scp ~/.ssh/id_rsa.pub root@xxxxxxxxx:/tmp将公钥拷贝到服务器上,xxx为服务器地址
(3)执行cat
- Java新手入门的30个基本概念一
aijuans
javajava 入门新手
在我们学习Java的过程中,掌握其中的基本概念对我们的学习无论是J2SE,J2EE,J2ME都是很重要的,J2SE是Java的基础,所以有必要对其中的基本概念做以归纳,以便大家在以后的学习过程中更好的理解java的精髓,在此我总结了30条基本的概念。 Java概述: 目前Java主要应用于中间件的开发(middleware)---处理客户机于服务器之间的通信技术,早期的实践证明,Java不适合
- Memcached for windows 简单介绍
antlove
javaWebwindowscachememcached
1. 安装memcached server
a. 下载memcached-1.2.6-win32-bin.zip
b. 解压缩,dos 窗口切换到 memcached.exe所在目录,运行memcached.exe -d install
c.启动memcached Server,直接在dos窗口键入 net start "memcached Server&quo
- 数据库对象的视图和索引
百合不是茶
索引oeacle数据库视图
视图
视图是从一个表或视图导出的表,也可以是从多个表或视图导出的表。视图是一个虚表,数据库不对视图所对应的数据进行实际存储,只存储视图的定义,对视图的数据进行操作时,只能将字段定义为视图,不能将具体的数据定义为视图
为什么oracle需要视图;
&
- Mockito(一) --入门篇
bijian1013
持续集成mockito单元测试
Mockito是一个针对Java的mocking框架,它与EasyMock和jMock很相似,但是通过在执行后校验什么已经被调用,它消除了对期望 行为(expectations)的需要。其它的mocking库需要你在执行前记录期望行为(expectations),而这导致了丑陋的初始化代码。
&nb
- 精通Oracle10编程SQL(5)SQL函数
bijian1013
oracle数据库plsql
/*
* SQL函数
*/
--数字函数
--ABS(n):返回数字n的绝对值
declare
v_abs number(6,2);
begin
v_abs:=abs(&no);
dbms_output.put_line('绝对值:'||v_abs);
end;
--ACOS(n):返回数字n的反余弦值,输入值的范围是-1~1,输出值的单位为弧度
- 【Log4j一】Log4j总体介绍
bit1129
log4j
Log4j组件:Logger、Appender、Layout
Log4j核心包含三个组件:logger、appender和layout。这三个组件协作提供日志功能:
日志的输出目标
日志的输出格式
日志的输出级别(是否抑制日志的输出)
logger继承特性
A logger is said to be an ancestor of anothe
- Java IO笔记
白糖_
java
public static void main(String[] args) throws IOException {
//输入流
InputStream in = Test.class.getResourceAsStream("/test");
InputStreamReader isr = new InputStreamReader(in);
Bu
- Docker 监控
ronin47
docker监控
目前项目内部署了docker,于是涉及到关于监控的事情,参考一些经典实例以及一些自己的想法,总结一下思路。 1、关于监控的内容 监控宿主机本身
监控宿主机本身还是比较简单的,同其他服务器监控类似,对cpu、network、io、disk等做通用的检查,这里不再细说。
额外的,因为是docker的
- java-顺时针打印图形
bylijinnan
java
一个画图程序 要求打印出:
1.int i=5;
2.1 2 3 4 5
3.16 17 18 19 6
4.15 24 25 20 7
5.14 23 22 21 8
6.13 12 11 10 9
7.
8.int i=6
9.1 2 3 4 5 6
10.20 21 22 23 24 7
11.19
- 关于iReport汉化版强制使用英文的配置方法
Kai_Ge
iReport汉化英文版
对于那些具有强迫症的工程师来说,软件汉化固然好用,但是汉化不完整却极为头疼,本方法针对iReport汉化不完整的情况,强制使用英文版,方法如下:
在 iReport 安装路径下的 etc/ireport.conf 里增加红色部分启动参数,即可变为英文版。
# ${HOME} will be replaced by user home directory accordin
- [并行计算]论宇宙的可计算性
comsci
并行计算
现在我们知道,一个涡旋系统具有并行计算能力.按照自然运动理论,这个系统也同时具有存储能力,同时具备计算和存储能力的系统,在某种条件下一般都会产生意识......
那么,这种概念让我们推论出一个结论
&nb
- 用OpenGL实现无限循环的coverflow
dai_lm
androidcoverflow
网上找了很久,都是用Gallery实现的,效果不是很满意,结果发现这个用OpenGL实现的,稍微修改了一下源码,实现了无限循环功能
源码地址:
https://github.com/jackfengji/glcoverflow
public class CoverFlowOpenGL extends GLSurfaceView implements
GLSurfaceV
- JAVA数据计算的几个解决方案1
datamachine
javaHibernate计算
老大丢过来的软件跑了10天,摸到点门道,正好跟以前攒的私房有关联,整理存档。
-----------------------------华丽的分割线-------------------------------------
数据计算层是指介于数据存储和应用程序之间,负责计算数据存储层的数据,并将计算结果返回应用程序的层次。J
&nbs
- 简单的用户授权系统,利用给user表添加一个字段标识管理员的方式
dcj3sjt126com
yii
怎么创建一个简单的(非 RBAC)用户授权系统
通过查看论坛,我发现这是一个常见的问题,所以我决定写这篇文章。
本文只包括授权系统.假设你已经知道怎么创建身份验证系统(登录)。 数据库
首先在 user 表创建一个新的字段(integer 类型),字段名 'accessLevel',它定义了用户的访问权限 扩展 CWebUser 类
在配置文件(一般为 protecte
- 未选之路
dcj3sjt126com
诗
作者:罗伯特*费罗斯特
黄色的树林里分出两条路,
可惜我不能同时去涉足,
我在那路口久久伫立,
我向着一条路极目望去,
直到它消失在丛林深处.
但我却选了另外一条路,
它荒草萋萋,十分幽寂;
显得更诱人,更美丽,
虽然在这两条小路上,
都很少留下旅人的足迹.
那天清晨落叶满地,
两条路都未见脚印痕迹.
呵,留下一条路等改日再
- Java处理15位身份证变18位
蕃薯耀
18位身份证变15位15位身份证变18位身份证转换
15位身份证变18位,18位身份证变15位
>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>
蕃薯耀 201
- SpringMVC4零配置--应用上下文配置【AppConfig】
hanqunfeng
springmvc4
从spring3.0开始,Spring将JavaConfig整合到核心模块,普通的POJO只需要标注@Configuration注解,就可以成为spring配置类,并通过在方法上标注@Bean注解的方式注入bean。
Xml配置和Java类配置对比如下:
applicationContext-AppConfig.xml
<!-- 激活自动代理功能 参看:
- Android中webview跟JAVASCRIPT中的交互
jackyrong
JavaScripthtmlandroid脚本
在android的应用程序中,可以直接调用webview中的javascript代码,而webview中的javascript代码,也可以去调用ANDROID应用程序(也就是JAVA部分的代码).下面举例说明之:
1 JAVASCRIPT脚本调用android程序
要在webview中,调用addJavascriptInterface(OBJ,int
- 8个最佳Web开发资源推荐
lampcy
编程Web程序员
Web开发对程序员来说是一项较为复杂的工作,程序员需要快速地满足用户需求。如今很多的在线资源可以给程序员提供帮助,比如指导手册、在线课程和一些参考资料,而且这些资源基本都是免费和适合初学者的。无论你是需要选择一门新的编程语言,或是了解最新的标准,还是需要从其他地方找到一些灵感,我们这里为你整理了一些很好的Web开发资源,帮助你更成功地进行Web开发。
这里列出10个最佳Web开发资源,它们都是受
- 架构师之面试------jdk的hashMap实现
nannan408
HashMap
1.前言。
如题。
2.详述。
(1)hashMap算法就是数组链表。数组存放的元素是键值对。jdk通过移位算法(其实也就是简单的加乘算法),如下代码来生成数组下标(生成后indexFor一下就成下标了)。
static int hash(int h)
{
h ^= (h >>> 20) ^ (h >>>
- html禁止清除input文本输入缓存
Rainbow702
html缓存input输入框change
多数浏览器默认会缓存input的值,只有使用ctl+F5强制刷新的才可以清除缓存记录。
如果不想让浏览器缓存input的值,有2种方法:
方法一: 在不想使用缓存的input中添加 autocomplete="off";
<input type="text" autocomplete="off" n
- POJO和JavaBean的区别和联系
tjmljw
POJOjava beans
POJO 和JavaBean是我们常见的两个关键字,一般容易混淆,POJO全称是Plain Ordinary Java Object / Pure Old Java Object,中文可以翻译成:普通Java类,具有一部分getter/setter方法的那种类就可以称作POJO,但是JavaBean则比 POJO复杂很多, Java Bean 是可复用的组件,对 Java Bean 并没有严格的规
- java中单例的五种写法
liuxiaoling
java单例
/**
* 单例模式的五种写法:
* 1、懒汉
* 2、恶汉
* 3、静态内部类
* 4、枚举
* 5、双重校验锁
*/
/**
* 五、 双重校验锁,在当前的内存模型中无效
*/
class LockSingleton
{
private volatile static LockSingleton singleton;
pri