1.GEPIA2
GEPIA服务器已经运行了两年,为来自42个国家的约11万名用户处理了约28万份分析请求。GEPIA2是GEPIA的更新版本,用于分析来自TCGA和GTEx项目的9736个肿瘤和8587个正常样本的==RNA测序表达数据==。
GEPIA2保留了GEPIA的所有功能,除此之外,GEPIA2还增加了新的数据集和功能。
重点增加的功能增加了基因亚型的分析、肿瘤亚型的分类、用户可以上传自己的数据与TCGA和GTEx上的数据进行对比,糅合等。
网页说明原文如下:
Highlight:
· Isoform analysis: Users can perform all expression analyses such as survival analysis and differential analysis at the isoform level. Usage of different transcripts across all cancer types can also be profiled.
· Cancer subtypes:Users can select their subtype of interest for further analysis, or compare different subtypes for expression and survival.
· Signature score:This function analyzes the prevalence of a gene signature in TCGA and GTEx samples, and provides tools such as correlation analysis and survival analysis to investigate the signature scores.
· Custom data:Users can upload their own cancer RNA-Seq data to identify its molecular subtype, TCGA immune subtype, and pan-cancer subtype. The gene and isoform expression can also be compared with the TCGA and GTEx data.
· Local package:GEPIA2 provides a python package for fast analysis and retrieval of the results from programs.
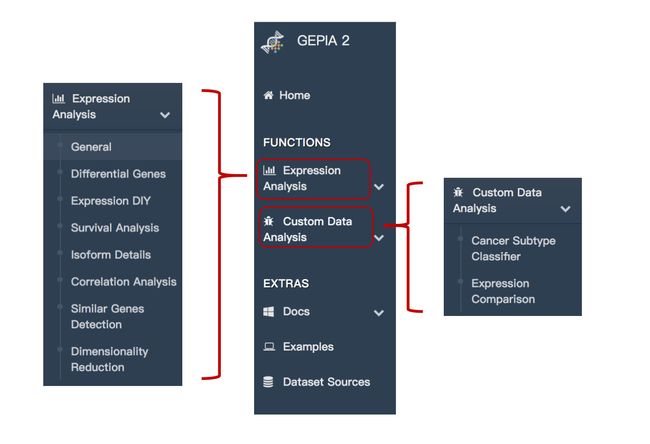
网页左边工具栏,主要是Expressin Analysis和Custom Data Analysis这两个板块。
Expressin Analysis
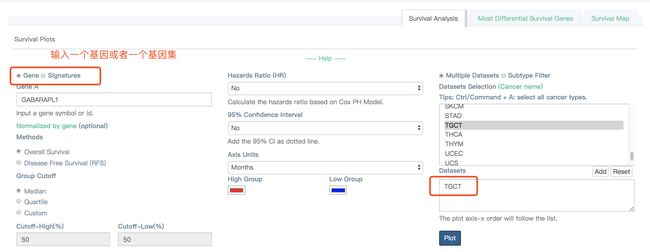
在输入基因栏处输入感兴趣的基因名,可以是基因symbol或者是gene Ensembl ID
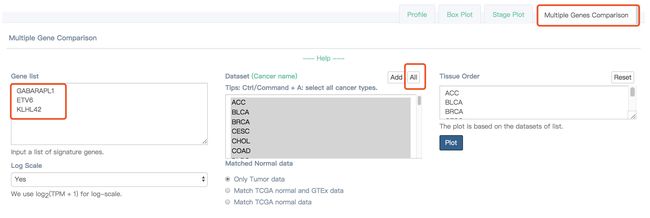
恰好看到老大让复现一张图的文章https://www.ncbi.nlm.nih.gov/pubmed/31213465中涉及到了染色体12p上的3个基因:GABARAPL1、ETV6、KLHL42,这三个基因与睾丸癌、卵巢癌、和肺癌中相关,所以想借着要学习GEPIA2这个网页神奇,用这三个基因在网页工具中学习。
General
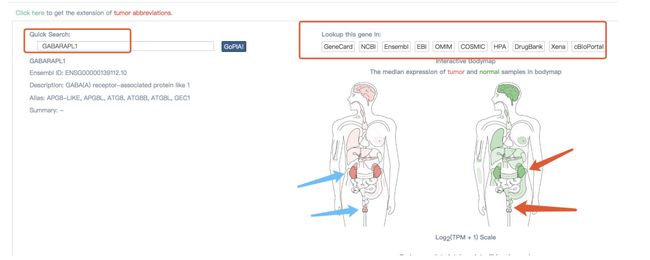
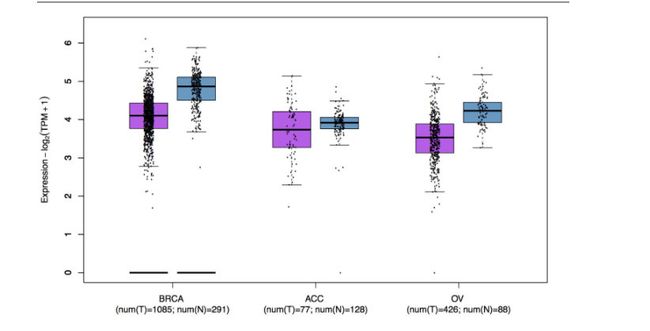
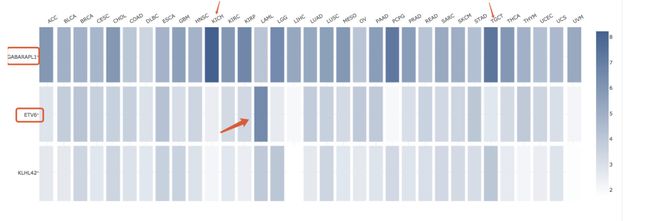
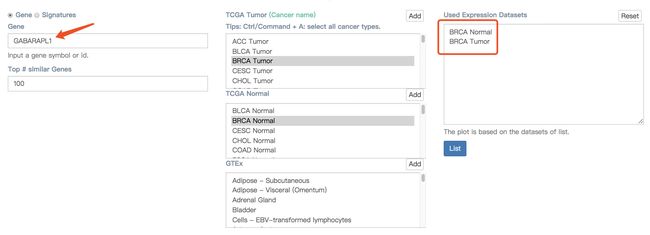
输入基因symbol或者是gene Ensembl ID,这里面输入了GABARAPL1这个基因。右边的两个示例人左边tumor,右边normal,颜色深浅代表高低表达,可以看到这个基因在肾中的表达,不论是在tumor还是mormal中,颜色都较其他组织深,不过肉眼看也分不清楚在肾中的表达到底是tumor高还是normal高。而在睾丸中,可以肉眼看到这个基因在睾丸中的表达高于其他组织,而在normal中,这个基因在睾丸中的表达与其他器官的表达是咩什么差别的。通过上面的粗略观察,我想这个正好和上面文章中表述的就是GABARAPL1、ETV6、KLHL42这三个基因有在睾丸中高表达是一致的。同时这个基因也会其他数据库想关联,可以跳转到其他数据库。
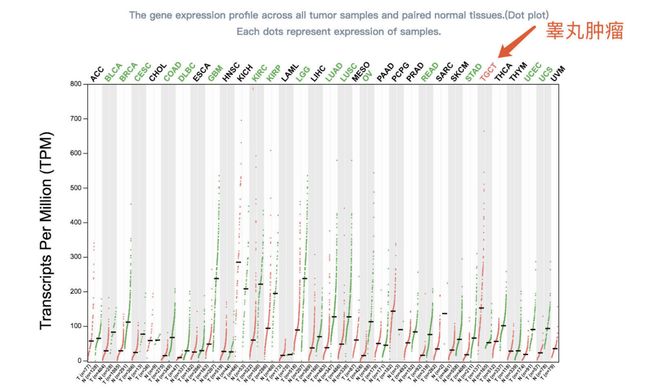
下面还有Dot plot图展示
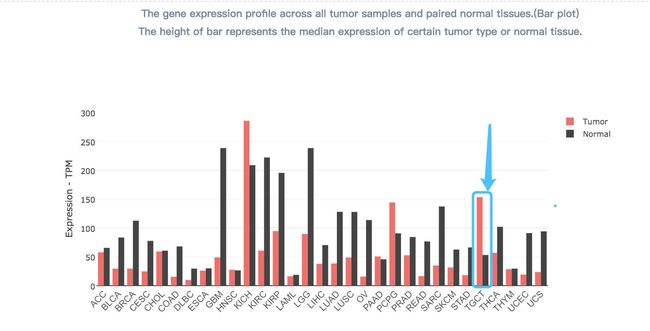
Bar plot图展示
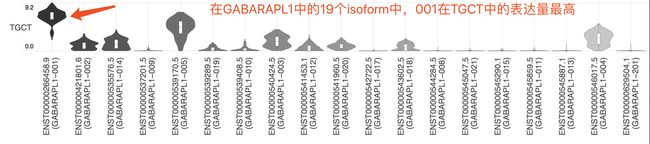
GABARAPL1-001的表达量最高。
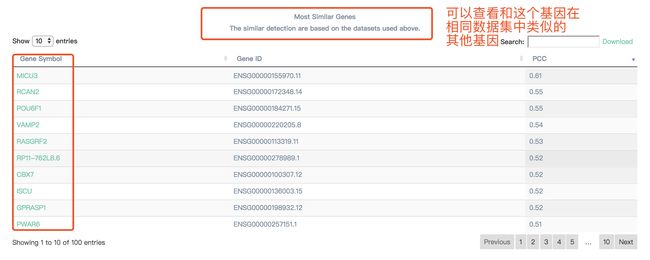
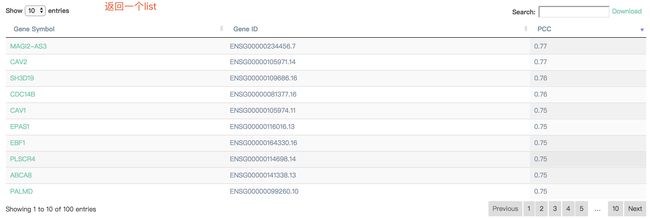
往下滑就是可以查看与这基因在同一数据集中的其他类似的基因
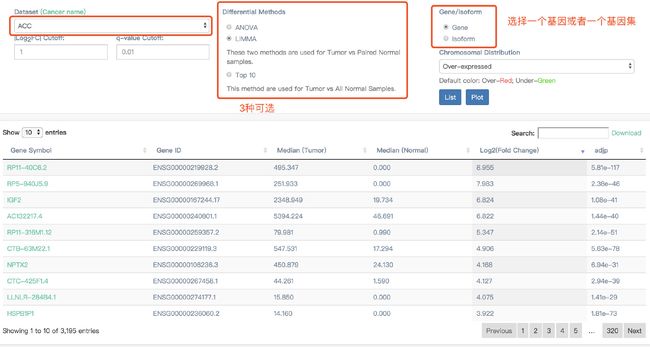
Differential Genes
列出肿瘤/正常组织中差异表达的基因或癌症类型的亚型,并绘制这些基因的染色体分布。
基因在染色体上的分布
Expression DIY
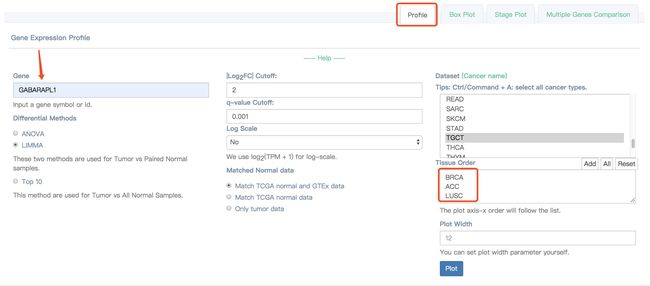
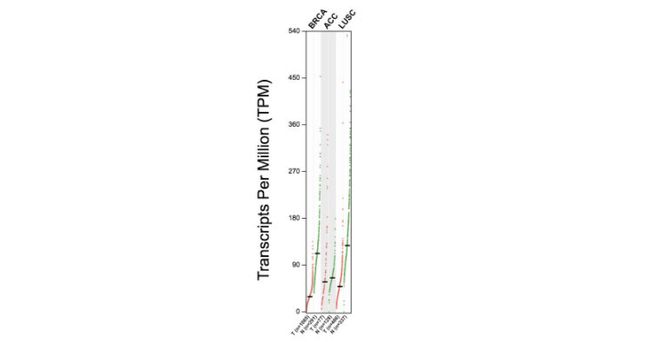
1.这个profile可以自己定义查看某个基因在某些癌症中的表达量,画出的图如下
2.查看某个基因或者某个基因集在某个癌症类型或某些癌症中的基因表达量
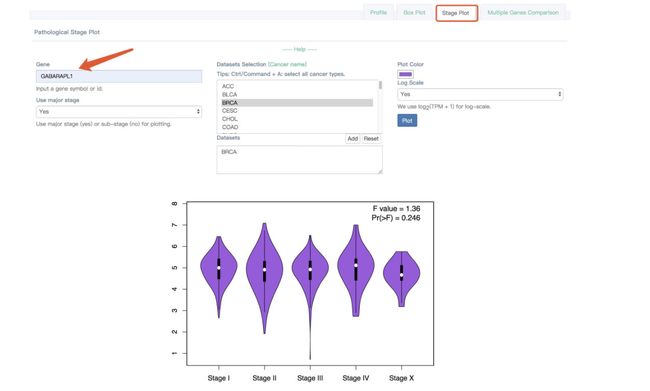
3.方框图来分析一个基因在不同癌症阶段的表达情况。
4.交互式的heatmap来分析不同癌症类型中一个基因的组织特异性表达。
Survial Analysis
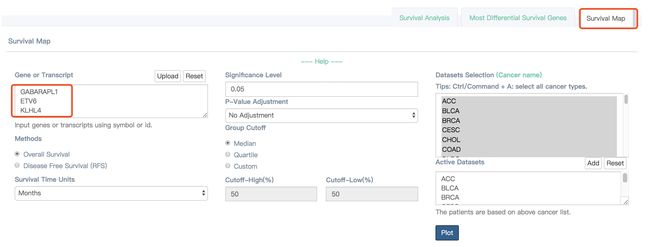
Sunvival Map
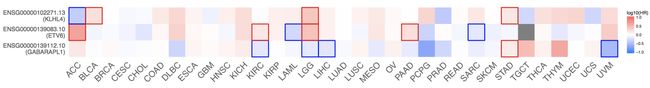
可根据生存热图查看基因或亚型表达水平对预后的影响。热图显示了不同基因的对数比例(log10)。红色和蓝色方块分别表示风险较高和较低。带有框架的矩形在预后分析中意味着显著的不利和有利结果。
Survival Map 结果图
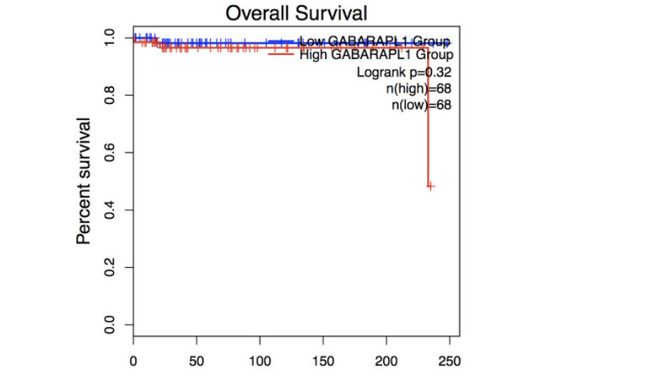
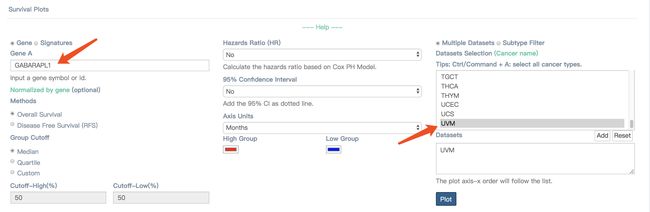
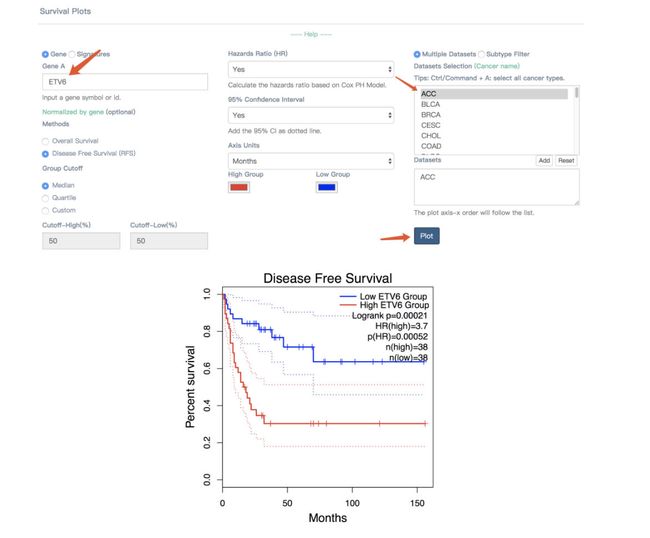
根据上面的生存分析热图中的结果,选择两个画框的基因对应的癌症,看看生存分析结果,分别选择GABARAPL1在UVM和ETV6在ACC中,做生存分析
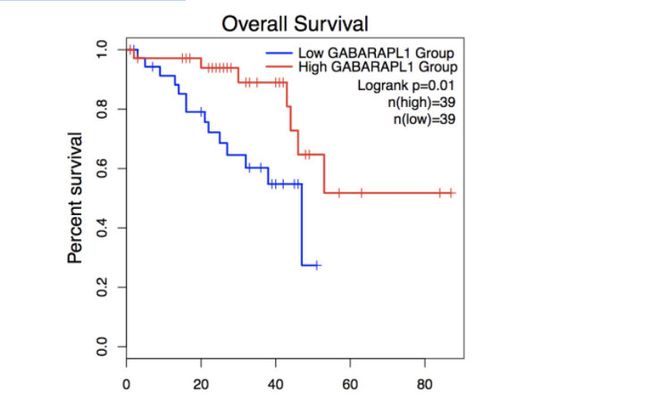
GABARAPL1这个基因对UVM(眼癌)的生存分析结果如下图,可以从上面热图看到,GABARAPL1这个基因在UVM中是低风险,虽然GABARAPL1高表达,但是对预后并不是危险因素
ETV6对ACC(肾上腺皮质癌)的生存影响,同样从上面的Survival map中可以看到,ETV6对ACC是红色画框,表示ETV6这个基因在表达量高的组的预后显著低于表达量低的组。
插播一些关于生存分析的概念性问题
- OS(overall survival):指从开始到任意原因死亡的时间,我们一般见到的5年生存率、10年生存率都是基于OS的
- progression-free survival(PFS,无进展生存期):指从开始到肿瘤发生任意进展或者发生死亡的时间;PFS相比OS包含了恶化这个概念,可用于评估一些治疗的临床效益
- time to progress(TTP,疾病进展时间):从开始到肿瘤发生任意进展或者进展前死亡的时间;TTP相比PFS只包含了肿瘤的恶化,不包含死亡
- disease-free survival(DFS,无病生存期):指从开始到肿瘤复发或者任何原因死亡的时间;常用于根治性手术治疗或放疗后的辅助治疗,如乳腺癌术后内分泌疗法等:
- event free survival(EFS,无事件生存期):指从开始到发生任何事件的时间,这里的事件包括肿瘤进展,死亡,治疗方案的改变,致死副作用等(主要用于病程较长的恶性肿瘤、或该实验方案危险性高等情况下)
COX比例风险模型和HR
COX模型是针对生存资料的一种回归分析方法,它可以估计HR及其可信区间。HR是治疗组风险率与对照组风险率之比的估计值。COX回归分析的一个假定条件是HR不随时间变化。因此,在一个临床试验研究中,假如“治愈”是终点事件,那么,HR表示在任何时间点,治疗组病人与对照组病人相比,疾病治愈的相对可能性大小。HR相当于高风险组中的个体首先出现终点事件的优势(假如终点事件是负性事件)。HR指治疗组病人治愈时间比对照组短的优势。有文献给出了一定条件下治疗组先被治愈的优势HR和治疗组先被治愈的概率P之间的关系公式:HR=odds=P/(1一P);P=HR/(I+HR)。所以HR=2相当于治疗组个体先被治愈的概率P为0.667。而对照组中个体先被治愈的概率为0.333。
Isoform Details
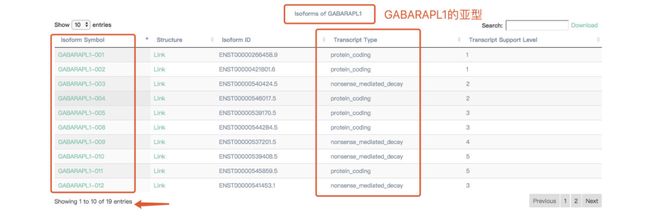
1.Isoform用法
应用GEPIA2生成小提琴图和条形图,可以查看给定的基因中每种亚型在不同肿瘤类型中的表达分布
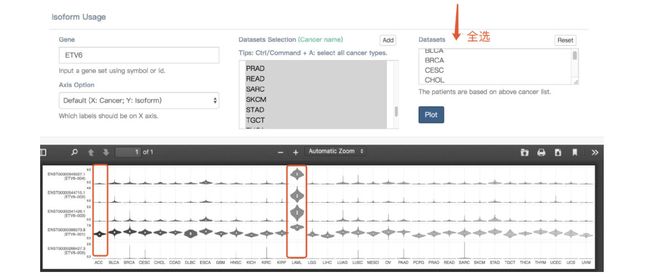
再输入一个基因ETV6,并add所有癌症类型,可以通过小提琴图看到ETV6的所有基因亚型在所有癌症中的表达情况。其中ETV6的好几个基因亚型都在LAML(急性髓细胞样白血病)中高表达(我查阅文献也找到很多关于这个基因与白血病的相关文章)。
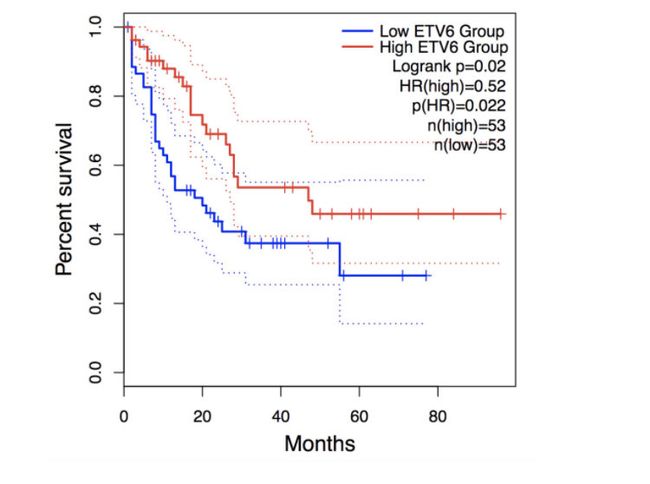
既然在表达量上相比其他基因如此显著,我就想看看生存分析结果了,正好前面的Survival heatmap显示的结果是这个基因高表达在LAML中是低风险,所以我去画个生存分析图看看。结果如下图,果真高表达,
2.查询基因亚型结构
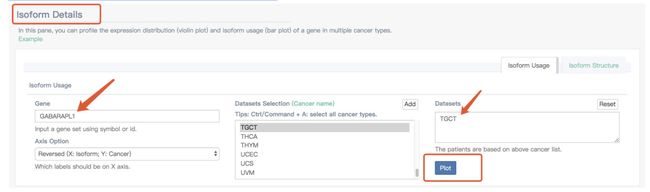
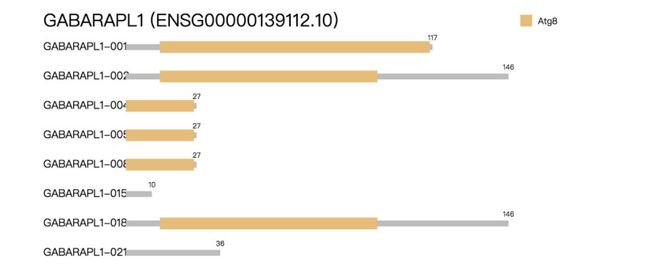
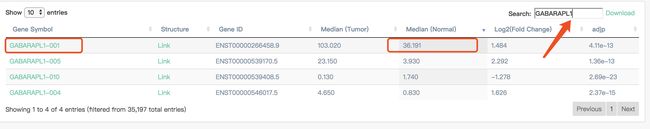
上面看到GABARAPL1这个基因是GABARAPL1-001在TGCT中的表达最高,因此在回到前面基因表达的栏目里验证一下他在这个TGCT里的表达量。
可以看到这个GABARAPL1-001亚型在TGCT中的表达量是最高的
Correlation Analysis
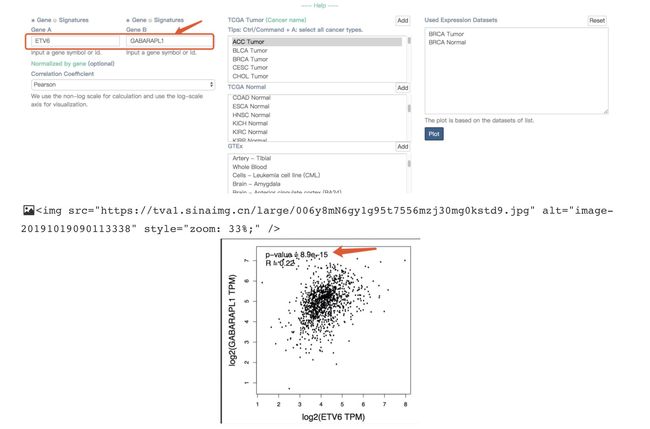
选择文章中ch12p的3个基因中的两个基因看相关性,ETV6和GABARAPL1,可以看到这两个基因在BRCA中的相关性很高。
选择的ETV6和一个它的表达量被列在与BRCA相关排位18线的一个基因做相关性,二者在乳腺癌中就没什么相关性。
Similar Genes Detection
这个模块是可以在不同的癌症类型和组织中寻找与基因或特征表达模式相似的基因。
Dimensionality Reduction
你可以根据一组基因的表达对来自不同癌症类型和组织的样本进行PCA降维。
Custom Data Analysis
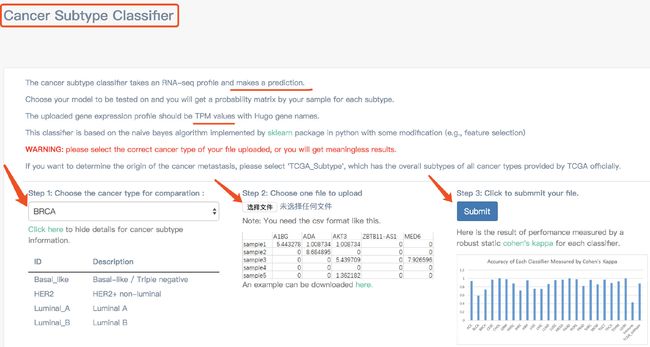
Cancer-subtype Classifier
可以根据基因表达准确预测上传样本的癌症类型或癌症亚型。
Uploaded expression data comparison
GEPIA2填补了用户自定义数据和公共癌症基因组数据之间的空白。该功能允许用户上传自己的数据,与肿瘤或来自TCGA或GTEx项目的正常数据进行差异分析。
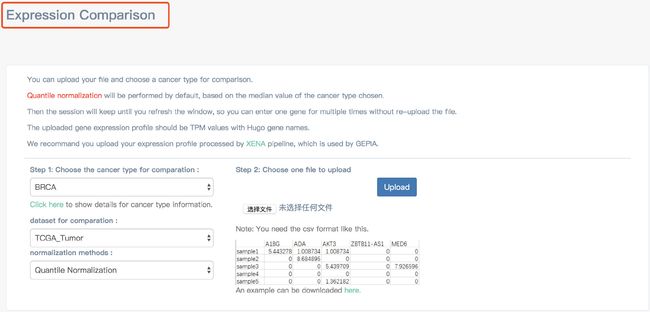
Expression Comparison
可以上传用户自己的文件并选择一个癌症类型进行比较。默认情况下,分位数归一化将根据所选癌症类型的中位数来执行。然后该会话将一直持续到刷新窗口,这样就可以多次输入一个基因而无需重新上传文件。上传的基因表达谱应该是带有Hugo基因名称的TPM值。
2.cBioPortal
- cBioPortal网站:http://cbioportal.org/
cBioPortal的数据来源和分析选项来源于多个网站,如TCGA data portal (https://tcga-data.nci.nih.gov/tcga/), the ICGC data portal(http://dcc.icgc.org/), the Broad Institute's Genome Data Analysis Center (GDAC) Firehose (http://gdac.broadinstitute.org)/), the IGV, the University of California, Santa Cruz (UCSC) Cancer Genomics Browser , IntOGen, Regulome Explorer (http://explorer.cancerregulome.org), 以及Oncomine (Reserach Edition) .
cBioPortal整合的基因数据类型包括体细胞突变,DNA拷贝数改变,mRNA和microRNA表达,DNA甲基化,蛋白丰度以及磷蛋白丰度。
- cBioPortal也收录了来自METABRIC数据库中的数据
METABRIC数据库
网址:http://molonc.bccrc.ca/aparicio-lab/research/metabric/
国际乳腺癌协会的分子分类数据库(Molecular Taxonomy of Breast Cancer International Consortium, METABRIC) 是一个加拿大-英国联合项目,旨在根据有助于确定最佳治疗过程的分子特征将乳腺肿瘤进一步分类。我们迄今为止已经根据肿瘤的基因指纹将乳腺癌重新分类为10个全新的类别。这些基因可以对乳腺癌生物学提供迫切需要的洞察力,使医生能够预测肿瘤是否会对某种特定的治疗产生反应。肿瘤是否有可能扩散到身体的其他部位,或者治疗后是否有可能复发。
- 关于CNA和CNV的理解
Copy number alterations/aberrations (CNAs) are changes in copy number that have arisen in somatic tissue (for example, just in a tumor).
Copy number variations (CNVs) originated from changes in copy number in germline cells (and are thus in all cells of the organism).
https://www.biostars.org/p/158956/
- 前面通过对GEPIA网页工具的学习已经可以画生存分析图了,但是关于文章最开始提到的这三个基因做生存分析

这三个基因在BRCA中并不显著影响生存分析结果,是因为GEPIA2这个数据库是针对TCGA和GTEx项目中的==RNA测序表达数据==做分析,而文章中的是CNA上的change,所以去cBioPortal这个网站做生存分析。
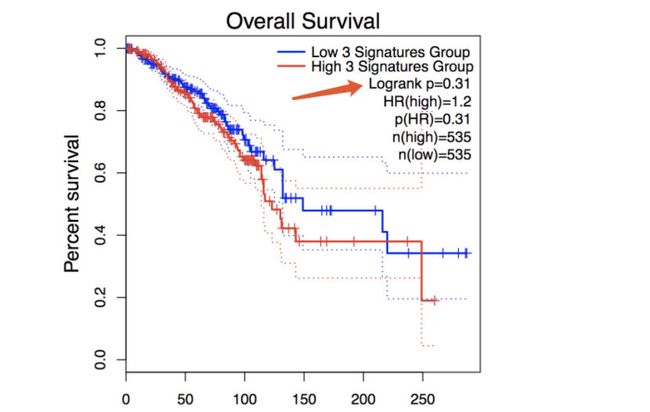
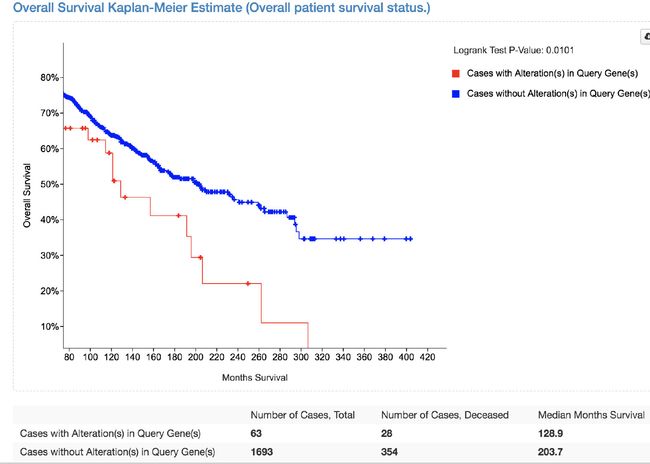
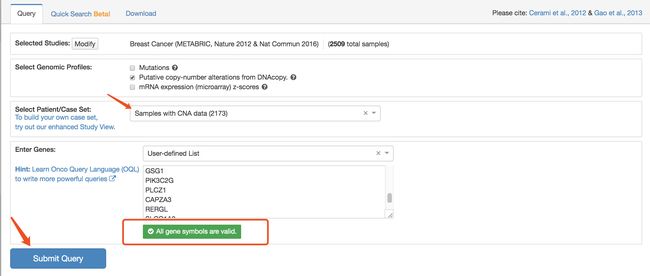
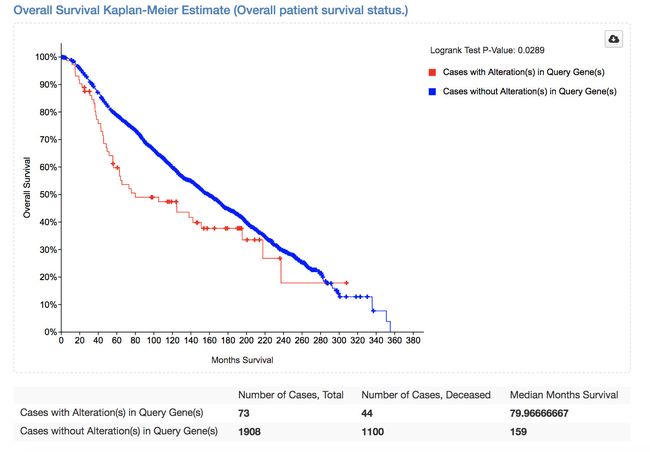
去cBioPortal主页,选择框中的数据集,然乎基因是一开始就提到的文章中的ch12p上的前150个基因和3个GABARAPL1、ETV6、KLHL42基因,选择copy-number alterations,最后作出生存分析图如下
如下图所示,可以看到当那些含有chr12p amplification 的patients的生存还是相比较对照组差的。
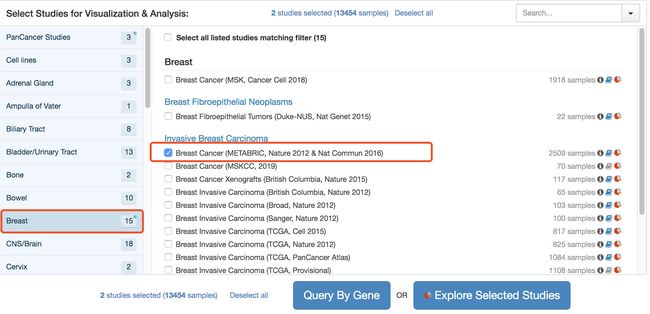
- 选择下面红框圈中的这个来自METABRIC这个数据库中的数据集,文章中也是选择的这个数据集。
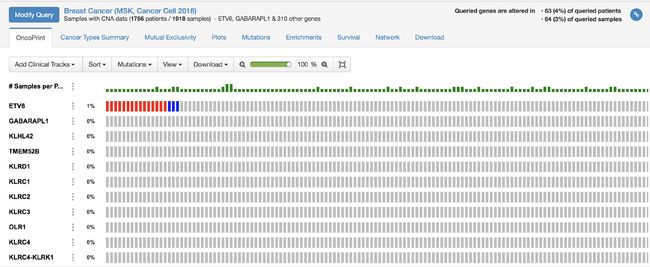
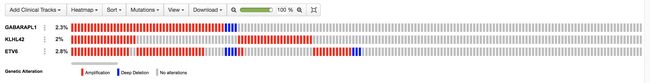
选择GABARAPL1、ETV6、KLHL42这3个基因,可以看到基因改变类型的比例的情况,这三个基因均发生了的Amplification
三个基因GABARAPL1、ETV6、KLHL42在BRCA中的生存分析图如下
最后友情宣传生信技能树
生物信息学“义诊”
生物信息学"拍卖会"
全国巡讲:R基础,Linux基础和RNA-seq实战演练 : 预告:12月28-30长沙站
广州珠江新城GEO数据挖掘滚动开班
DNA及RNA甲基化数据分析与课题设计