文章来源:科研圈 2018-05-12
北京大学生命科学学院贾璐萌,李亭亭(李程研究组)
上回说到Hi-C更适用于研究大尺Hi度上的染色质结构,而不适用于研究转录调控元件之间的相互作用。
转录调控元件主要包括启动子,增强子,绝缘子和抑制子。在转录的起始阶段,基因的启动子与其他远距离调控元件(增强子,绝缘子和抑制子)空间靠近(图4)。距离调控元件通过帮助启动子招募具有抑制或者激活功能的转录相关蛋白,调控基因的转录(Zhang et al。,2013)。为了结合转录相关蛋白,这些转录调控元件通常缺少核小体的结合,这使用活跃调控区域的染色质相对开放(开放染色质)。针对转录调控元件的这些特点,可通过捕获转录相关蛋白结合区域[ChIP-seq(Johnson et al。,2007)]和捕获染色质开放区域[DNase-seq(Boyle等人,2008),FAIRE-seq(Giresi等人,2007),MNase-seq(Schlesinger等人,2013),ATAC-seq(Buenrostro等人,2013) -seq(Ponnaluri等,2017)]的技术来检测转录调控元件。
为了更好的捕获基因组转录调控元件之间的远距离相互作用,结合转录调控元件检测技术的其他染色质构象捕获方法被提出。这些方法主要包括三类。
第一类,是转录相关蛋白介导的染色质相互作用的捕获技术(Chia-PET(Fullwood等人,2009),HiChIP(Mumbach等人,2016),PLAC-seq(Fang等人, 2016))。在这类技术中,最早被提出的是阮一骏等开发的嘉PET技术(图5)。在上回说到喜-C更适用于研究大尺度上的染色质结构,而不适用于研究转录调控元件之间的相互作用。
转录调控元件主要包括启动子,增强子,绝缘子和抑制子。在转录的起始阶段,基因的启动子与其他远距离调控元件(增强子,绝缘子和抑制子)空间靠近(图4)。距离调控元件通过帮助启动子招募具有抑制或者激活功能的转录相关蛋白,调控基因的转录(Zhang et al。,2013)。为了结合转录相关蛋白,这些转录调控元件通常缺少核小体的结合,这使用活跃调控区域的染色质相对开放(开放染色质)。针对转录调控元件的这些特点,可通过捕获转录相关蛋白结合区域[ChIP-seq(Johnson et al。,2007)]和捕获染色质开放区域[DNase-seq(Boyle等人,2008),FAIRE-seq(Giresi等人,2007),MNase-seq(Schlesinger等人,2013),ATAC-seq(Buenrostro等人,2013) -seq(Ponnaluri等,2017)]的技术来检测转录调控元件。
为了更好的捕获基因组转录调控元件之间的远距离相互作用,结合转录调控元件检测技术的其他染色质构象捕获方法被提出。这些方法主要包括三类。
第一类,是转录相关蛋白介导的染色质相互作用的捕获技术(Chia-PET(Fullwood等人,2009),HiChIP(Mumbach等人,2016),PLAC-seq(Fang等人, 2016))。在这类技术中,最早被提出的是阮一骏等开发的嘉PET技术(图5)。在嘉PET技术中,交联好的染色质首先被超声打碎成独立的相互作用复合体;结合有目的蛋白的复合体通过免疫沉淀技术被富集;目的蛋白结合的复合体的DNA末端被连接包含MMEI位点的生物素化寡核苷酸接头;通过连接体间的连接,目的蛋白介导的存在相互作用的DNA被连接至同一片段; MmeI片段化DNA后,DNA被纯化;随后PET片段被建立库测序.Chia-PET作为一种可以有效富集蛋白介导的染色质间相互作用的技术,自开发以来,得到了广泛的应用.RNAPII ChIA-PET及CTCF ChIA-PET技术不仅帮助解析了转录相关的染色质拓扑结构(Li等,2012; Tang等,2015 ),还成功帮助解 了解非编码区SNP位点对远距离调控基因表达的影响(Tang et al。,2015)。2017年,同样是用于捕获基因组转换调控元件之间的远距离相互作用的技术Hi-ChIP(Mumbach这两种技术都在Hi-C技术的基础上结合了免疫沉淀技术,从实验目的上与ChIA-PET一致,目前来看,与嘉PET技术相比,喜芯片与PLAC-SEQ能得到更高比例的有效读取。相信喜芯片与PLAC-seq的也将被广泛应用于蛋白介导的染色质相互作用的研究。
第二类,是目的探针所在区域的染色质相互作用的捕获(Capture Hi-C)。这类技术以启动子捕获Hi-C(Mifsud等,2015)技术为代表,根据启动子区域设计RNA探针,在高 - C实验的基础上加入了一步探针的富集,检测启动子探针所在区域的染色质相互作用。
第三类,是基于开放染色质间相互作用的捕获[DNase Hi-C(Ma等人,2015),Micro-C(Hsieh等人,2015),BL-Hi-C(Liang等人, ,DNase Hi-C技术使用DNase酶取代Hi-C实验中的限制性内切酶,消化染色质,可特异性地捕获DNase I敏感的开放染色质间相互作用(Ma et al。 ,2015).Micro-C技术使用MNase取代Hi-C实验中的限制性内切酶消化染色质,可以得到核小体尺度分辨率的DNA相互作用图谱(Hsieh等,2015).BL- Hi-C技术使用识别序列为“GGCC”的限制性内切酶HaeIII进行染色质消化。由于结合有转录相关蛋白及结构蛋白的活跃染色质区域DNA的GC碱基组成比例更高,故BL-喜-C可富集高GC区域的活跃染色质间的相互作用。
这些方法各有优劣及侧重,从不同层面上解析染色质空间结构,回答重要生物学问题.Chia-PET(Fullwood等人,2009),HiChIP(Mumbach等人,2016),PLAC-seq (Fang et al。,2016)仅能捕获某一种蛋白介导的染色质间的相互作用; Capture Hi-C技术通过特异性探针,捕获探针所在区域(如启动子区)染色质间相互作用(Mifsud et al。,2015); DNase Hi-C虽然可以富集开放染色质之间的相互作用,但是由于DNase I整合效率非常高,高度片段化的染色质之间自连或随机连接,减少了真实相互作用的捕获比例(Li et al。,2018).BL-Hi-C捕获了10000多个高GC区域的染色质相互作用,这些相互作用大多是CTCF HiChIP及RNAPII HiChIP的子集。这些技术虽然各有优势,然而大都依赖目的蛋白,探针序列,酶切偏好。综上所述,一种不依赖于探针序列及蛋白抗体,用染色质开放程度为富集条件,直接高效富集全基 组活跃转录调控元件间相互作用的技术需要被开发。
OCEAN-C(Open Chromatin Enrichment And Network Hi-C)技术(图 6)(Li et al., 2018)是结合了 FAIRE-seq 技术及 Hi-C 技术的关键步骤而开发的一种可以不依赖蛋白抗体及靶序列的新技术。在OCEAN-C技术中,染色质首先被甲醛交联;随后,染色质被限制性内切酶(MboI)消化;DNA末端修复时引入带有生物素标记的脱氧核糖核苷酸;随后空间存在相互作用的DNA被连接;超声打断染色质后,开放染色质被酚-氯仿抽提;最后,存在相互作用的开放染色质因为有生物素信号,可以被富集用于后续文库构建。
need-to-insert-img
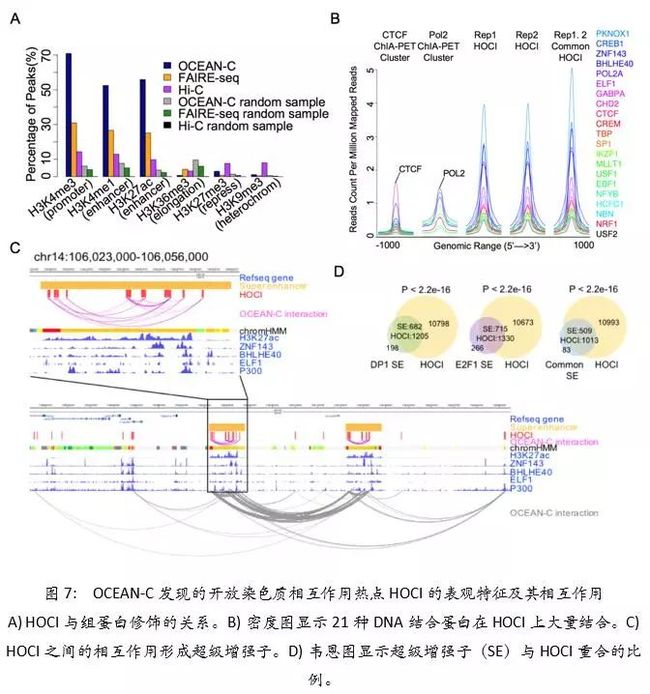
与其他技术相比,第一:OCEAN- C 技术不仅能检测到TAD、compartments等三维基因组结构,而且同时富集开放染色质区域及这些开放染色质之间的相互作用。第二:我们在三种 B 细胞相关细胞(U266,RPMI8226,GM12878)中应用了 OCEAN-C 技术,发现三种细胞中均有近 10000 个开放染色质相互作用中心(HOCI)。HOCI长度在 1 kb 左右、以活跃启动子及增强子为主、并结合有大量转录相关蛋白。许多HOCI也与Super-enhancer、broad H3K4me3区域有重合,帮助理解这些新发现的重要调控区域内部和之间的互作(图7)。第三:HOCI作为相互作用网络的重要节点,与其他开放染色质互作,形成全基因组开放染色质相互作用网络,调控基因表达(图8)。
need-to-insert-img
随着二代测序技术的发展,越来越多的染色质构象捕获技术被开发。三维基因组实验技术的应用越来越广泛,在细胞周期、细胞分化、疾病、指导基因组组装等各方面的研究中都有应用。例如,Hi-C技术用于研究细胞有丝分裂过程中的染色质结构变化:在有丝分裂中期,染色质会失去分区和 TAD结构,坍缩为一个密集的染色质状态 (Naumova et al., 2013);5C技术用于研究干细胞分化过程中核染色质的变化:在分化过程中,由于染色质三维结构的变化,OCT4的启动子和远程增强子的相互作用减弱,导致OCT4表达量下降,促进细胞分化 (Beagan et al., 2016)。Hi-C技术同样成功揭示了TAD边界的变化对疾病产生的影响:TAD边界遭到破坏造成原本被隔离的增强子与启动子形成异常的相互作用,导致基因时空表达的紊乱而产生疾病,如手指畸形疾病 (Lupianez et al., 2015)和大脑胶质瘤 (Flavahan et al., 2016);
如今,随着三维基因组研究方法的更新和研究范围的扩展,越来越多的新的染色质构象捕获技术被开发。最近,曹罡课题组和李国亮课题组又合作开发了更加高效经济的DLO Hi-C技术(Lin et al., 2018)。不仅如此,单细胞Hi-C技术也逐渐被开发 (Flyamer et al., 2017; Nagano et al., 2013; Ramani et al., 2017)。2014年美国国立卫生研究院(NIH)为此专门制定了4DN计划(4D Nucleome)(Dekker et al., 2017),斥资1.2亿美元资助了24个研究项目。4DN计划结合三维基因组学、单细胞测序、单细胞实时成像等前沿技术,开展标准化、高通量、单细胞层面、可动态观测的三维基因组学技术和应用研究。这些技术和项目融合了多学科的技术,将极大地促进后基因组学时代从基因组的一维列研究向四维的基因组空间结构和随时间变化的研究转变。我们将继续开发和应用OCEAN-C技术,研究以富集超级增强子、broad H3K4me3等结合有大量转录因子的基因调控元件间的相互作用,并与基因表达变化关联,帮助进一步阐明基因组结构、功能与疾病的关系。
技术中,交联好的染色质首先被超声打碎成独立的相互作用复合体;结合有目的蛋白的复合体通过免疫沉淀技术被富集;目的蛋白结合的复合体的DNA末端被连接包含MMEI位点的生物素化寡核苷酸接头;通过连接体间的连接,目的蛋白介导的存在相互作用的DNA被连接至同一片段; MmeI片段化DNA后,DNA被纯化;随后PET片段被建立库测序.Chia-PET作为一种可以有效富集蛋白介导的染色质间相互作用的技术,自开发以来,得到了广泛的应用.RNAPII ChIA-PET及CTCF ChIA-PET技术不仅帮助解析了转录相关的染色质拓扑结构(Li等,2012; Tang等,2015 ),还成功帮助解 了解非编码区SNP位点对远距离调控基因表达的影响(Tang et al。,2015)。2017年,同样是用于捕获基因组转换调控元件之间的远距离相互作用的技术Hi-ChIP(Mumbach这两种技术都在Hi-C技术的基础上结合了免疫沉淀技术,从实验目的上与ChIA-PET一致,目前来看,与嘉PET技术相比,喜芯片与PLAC-SEQ能得到更高比例的有效读取。相信喜芯片与PLAC-seq的也将被广泛应用于蛋白介导的染色质相互作用的研究。
第二类,是目的探针所在区域的染色质相互作用的捕获(Capture Hi-C)。这类技术以启动子捕获Hi-C(Mifsud等,2015)技术为代表,根据启动子区域设计RNA探针,在高 - C实验的基础上加入了一步探针的富集,检测启动子探针所在区域的染色质相互作用。
第三类,是基于开放染色质间相互作用的捕获[DNase Hi-C(Ma等人,2015),Micro-C(Hsieh等人,2015),BL-Hi-C(Liang等人, ,DNase Hi-C技术使用DNase酶取代Hi-C实验中的限制性内切酶,消化染色质,可特异性地捕获DNase I敏感的开放染色质间相互作用(Ma et al。 ,2015).Micro-C技术使用MNase取代Hi-C实验中的限制性内切酶消化染色质,可以得到核小体尺度分辨率的DNA相互作用图谱(Hsieh等,2015).BL- Hi-C技术使用识别序列为“GGCC”的限制性内切酶HaeIII进行染色质消化。由于结合有转录相关蛋白及结构蛋白的活跃染色质区域DNA的GC碱基组成比例更高,故BL-喜-C可富集高GC区域的活跃染色质间的相互作用。
这些方法各有优劣及侧重,从不同层面上解析染色质空间结构,回答重要生物学问题.Chia-PET(Fullwood等人,2009),HiChIP(Mumbach等人,2016),PLAC-seq (Fang et al。,2016)仅能捕获某一种蛋白介导的染色质间的相互作用; Capture Hi-C技术通过特异性探针,捕获探针所在区域(如启动子区)染色质间相互作用(Mifsud et al。,2015); DNase Hi-C虽然可以富集开放染色质之间的相互作用,但是由于DNase I整合效率非常高,高度片段化的染色质之间自连或随机连接,减少了真实相互作用的捕获比例(Li et al。,2018).BL-Hi-C捕获了10000多个高GC区域的染色质相互作用,这些相互作用大多是CTCF HiChIP及RNAPII HiChIP的子集。这些技术虽然各有优势,然而大都依赖目的蛋白,探针序列,酶切偏好。综上所述,一种不依赖于探针序列及蛋白抗体,用染色质开放程度为富集条件,直接高效富集全基 组活跃转录调控元件间相互作用的技术需要被开发。
OCEAN-C(Open Chromatin Enrichment And Network Hi-C)技术(图 6)(Li et al., 2018)是结合了 FAIRE-seq 技术及 Hi-C 技术的关键步骤而开发的一种可以不依赖蛋白抗体及靶序列的新技术。在OCEAN-C技术中,染色质首先被甲醛交联;随后,染色质被限制性内切酶(MboI)消化;DNA末端修复时引入带有生物素标记的脱氧核糖核苷酸;随后空间存在相互作用的DNA被连接;超声打断染色质后,开放染色质被酚-氯仿抽提;最后,存在相互作用的开放染色质因为有生物素信号,可以被富集用于后续文库构建。
与其他技术相比,第一:OCEAN- C 技术不仅能检测到TAD、compartments等三维基因组结构,而且同时富集开放染色质区域及这些开放染色质之间的相互作用。第二:我们在三种 B 细胞相关细胞(U266,RPMI8226,GM12878)中应用了 OCEAN-C 技术,发现三种细胞中均有近 10000 个开放染色质相互作用中心(HOCI)。HOCI长度在 1 kb 左右、以活跃启动子及增强子为主、并结合有大量转录相关蛋白。许多HOCI也与Super-enhancer、broad H3K4me3区域有重合,帮助理解这些新发现的重要调控区域内部和之间的互作(图7)。第三:HOCI作为相互作用网络的重要节点,与其他开放染色质互作,形成全基因组开放染色质相互作用网络,调控基因表达(图8)。
随着二代测序技术的发展,越来越多的染色质构象捕获技术被开发。三维基因组实验技术的应用越来越广泛,在细胞周期、细胞分化、疾病、指导基因组组装等各方面的研究中都有应用。例如,Hi-C技术用于研究细胞有丝分裂过程中的染色质结构变化:在有丝分裂中期,染色质会失去分区和 TAD结构,坍缩为一个密集的染色质状态 (Naumova et al., 2013);5C技术用于研究干细胞分化过程中核染色质的变化:在分化过程中,由于染色质三维结构的变化,OCT4的启动子和远程增强子的相互作用减弱,导致OCT4表达量下降,促进细胞分化 (Beagan et al., 2016)。Hi-C技术同样成功揭示了TAD边界的变化对疾病产生的影响:TAD边界遭到破坏造成原本被隔离的增强子与启动子形成异常的相互作用,导致基因时空表达的紊乱而产生疾病,如手指畸形疾病 (Lupianez et al., 2015)和大脑胶质瘤 (Flavahan et al., 2016);
如今,随着三维基因组研究方法的更新和研究范围的扩展,越来越多的新的染色质构象捕获技术被开发。最近,曹罡课题组和李国亮课题组又合作开发了更加高效经济的DLO Hi-C技术(Lin et al., 2018)。不仅如此,单细胞Hi-C技术也逐渐被开发 (Flyamer et al., 2017; Nagano et al., 2013; Ramani et al., 2017)。2014年美国国立卫生研究院(NIH)为此专门制定了4DN计划(4D Nucleome)(Dekker et al., 2017),斥资1.2亿美元资助了24个研究项目。4DN计划结合三维基因组学、单细胞测序、单细胞实时成像等前沿技术,开展标准化、高通量、单细胞层面、可动态观测的三维基因组学技术和应用研究。这些技术和项目融合了多学科的技术,将极大地促进后基因组学时代从基因组的一维列研究向四维的基因组空间结构和随时间变化的研究转变。我们将继续开发和应用OCEAN-C技术,研究以富集超级增强子、broad H3K4me3等结合有大量转录因子的基因调控元件间的相互作用,并与基因表达变化关联,帮助进一步阐明基因组结构、功能与疾病的关系。