把之前写在CSDN的博文搬运了过来,集中记录自己的学习过程。
第一次写博客,必须mark一下:2018.07.27
sra toolkit是ncbi上将 .sra文件转换为 .fastq.gz文件的工具。
1.下载/调用 SRA Toolkit
可以直接在linux里在线下载,要根据自己的系统选择合适的安装版本。我查看了一下主机的linux为redhat类型,没找到这个类型的版本,又去服务器看了一下其他人的sratoolkit版本,都是centos_linux,因而选择这个版本应该是合适的。
wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/2.9.2/sratoolkit.2.9.2-centos_linux64.tar.gz
不清楚版本可将 2.9.2 替换成 current 。
关于调用,直接调用其他用户路径下的工具(locate sratoolkit)会显示permission denied,显然是设置了权限,无法查看和调用其他用户home目录下的东西。
但是问题来了,按理说这种常用的软件公用系统(不知道这个说法对不对,计算机小白轻喷)里肯定会有,但是我不知道怎么用命令直接调用。
下载好之后解压:
tar xzvf sratoolkit.2.9.2-centos_linux64.tar.gz
解压缩之后就可以通过绝对路径使用,但为了以后使用方便,可以直接用程序名调用,最好配置一下环境变量。
mv sratoolkit.2.9.2-centos_linux64 ~/local/app/ #移动到指定文件夹
cd ~/local/app/ #进入本地程序安装路径
mv sratoolkit.2.9.2-centos_linux64 sratoolkit #去掉版本号是为了避免因升级而需要修改配置文件
此处参考> http://blog.shenwei.me/local-blast-installation/
接下来是添加环境变量:
vi ~/.bashrc #用vi/vim编辑器修改bashrc文件
i #由command line进入insertion line
export PATH=$PATH:/home/urname/local/app/sratoolkit/bin
ESC, :wq #退出vi编辑器并保存文件
source ~/.bashrc #让配置生效
上面是配置永久生效的办法,如果只是临时使用,直接在终端中输入:
export PATH=$PATH:/home/urname/local/app/sratoolkit/bin
2.从NCBI的SRA库里下载数据
关于SRA库的基础知识,可以看这里:https://shengxin.ren/article/16
我是跟着 生信技能树 学习他们的 HiC数据分析实战 https://vip.biotrainee.com/d/779-hic ,所以和里面的教程一样,我也只下载了 SRR824846一个测序结果集。
下载数据有两种方法:
wget https://trace.ncbi.nlm.nih.gov/Traces/sra/?run=SRR824846
prefetch SRR824846
本来以为高枕无忧了,结果客户端显示连接超时了,只能重登重新下载,请教师兄,学习到了一个让程序后台运行的命令nohup,只要服务器主机没掉线就行,即使关闭了本地终端窗口或者断网了也可以继续执行命令。让进程后台运行的命令有好几种,如nohup/&/disown等,disown是事后补救用的,我没有多了解。一般nohup和&一起用。
nohup 命令可以使命令永久的执行下去,和终端没有关系,退出终端也不会影响程序的运行; & 是后台运行的意思,但当用户退出的时候,命令自动也跟着退出。 那么,把两个结合起来nohup 命令 &这样就能使命令永久的在后台执行。
nohup prefetch SRR824846 &
如何查看进程?如果命令正在当前终端运行:
jobs -l #可以查看当前终端生效的进程
如果已经退出,又打开新的终端:
top #查看所有用户的运行进程,也可以看到自己的进程号
ps -ef|grep urname ##只查看自己的进程记录
如果要终止后台运行命令:
kill -9 进程号
又遇到了一个坑。。在上面修改环境变量的过程中,我是非常马虎地把bashrc文件清空后(因为之前只添加了blast的环境变量,我也没仔细看是否还有其他内容),再添加了新的环境变量。结果用户名的位置变成了-bash-4.1$。google了一下原因, 表明是
没有执行 .bash_profile .bashrc 导致的结果。
解决办法:查了一下网上的教程,据说在 .bash_profile里修改PS1变量就可以了,但我的问题出在 .bashrc上,不敢再动 .bash_profile了。我肯定问题出在bashrc文件里,现在要做的就是把bashrc文件恢复过来,然而我没有备份…这里要敲黑板,.bashrc .bash_profile等环境变量设置文件是很重要的文件,修改不当甚至会无法进入linux系统,所以要谨慎修改并在修改前做好备份!
用系统中存储的.bashrc备份文件恢复到~/目录下
cp /etc/skel/.bashrc ~/
之后退出系统再登录,用户名路径就恢复正常了。
3.文件拆分及格式转换
文件下载完成后放在了home目录下的ncbi/public/sra 文件夹里,可以看到新建了一个SRR824846.sra 文件。在转换文件格式前要清楚sra文件的数据类型,在下载时就能看到其为“paired-end”测序结果。二代测序主要有单端测序和双端测序两种方式,具体内容见:https://vip.biotrainee.com/d/127-paired-end-reads ,以及http://www.bio-info-trainee.com/298.html 。
使用fastq-dump拆分PE文件时有两种常用的参数,--split-files 和--split-3 ,很奇怪,查到以下解释:
--split-spot: 将双端测序分为两份,但是都放在同一个文件中
--split-files: 将双端测序分为两份,放在不同的文件,但是对于一方有而一方没有的reads直接丢弃
--split-3 : 将双端测序分为两份,放在不同的文件,但是对于一方有而一方没有的reads会单独放在一个文件夹里
作者:hoptop
链接:https://www.jianshu.com/p/a8d70b66794c
cd ~/ncbi/public/sra/ #将拆分文件放在sra文件夹里
fastq-dump --split-3 SRR824846 #将双端测序文件拆分为两个reads
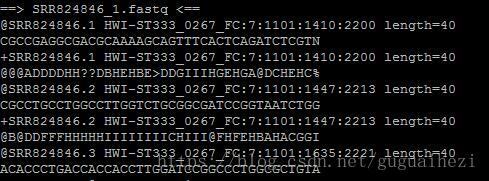
打开1文件,得到如图结果:
不过在上一步可以加上
-gzip 命令,输出gz的压缩格式,好处是可以节省空间,而且比对软件一般都支持。(参考hoptop的文章: https://www.jianshu.com/p/a8d70b66794c)