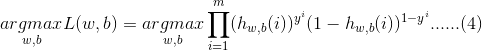《Python机器学习及实践:从零开始通往Kaggle竞赛之路》第2章 基础篇 学习笔记(一)2.1.1.1线性分类器总结
目录
2.1.1.1线性分类器
0、引言
(1)监督学习任务的基本架构和流程
(2)分类学习
1、模型介绍
(1)线性分类器的定义
(2)逻辑斯蒂函数
(3)逻辑斯蒂回归模型
2、数据描述
(1)数据下载
(2)数据描述
(3)数据预处理
(4)训练和测试数据
3、编程实践
4、性能测评
5、特点分析
2.1.1.1线性分类器
0、引言
对于每一类经典模型,都将从模型简介、数据描述、编程实践、性能测评以及特点分析5个角度分别进行阐述。
机器学习中监督学习模型的任务重点在于,根据已有经验知识对未知样本的目标/标记进行预测。根据目标预测变量的类型不同,把监督学习任务大体分为分类学习与回归预测两类。
(1)监督学习任务的基本架构和流程
首先准备训练数据,可以是文本、图像、音频等;然后抽取所需要的特征,形成特征向量;接着,把这些特征向量连同对应的标记/目标一并送入学习算法中,训练出一个预测模型;然后,采用同样的特征抽取方法作用于新测试数据,得到用于测试的特征向量;最后,使用预测模型对这些待测试的特征向量进行预测并得到结果。
(2)分类学习
分类学习是最为常见的监督学习问题,其中,最基础的便是二分类问题,即判断是非,从两个类别中选择一个作为预测结果;除此之外还有多类分类的问题,即在多于两个类别中选择一个;甚至还有多标签分类问题,与上述二分类以及多类分类问题不同,多标签分类问题判断一个样本是否同时属于多个不同类别。
比如,医生对肿瘤性质的判定;邮政系统对手写体邮编数字进行识别;互联网资讯公司对新闻进行分类;生物学家对物种类型的鉴定;甚至,还能够对某些大灾难的经历者是否生还进行预测等。
1、模型介绍
(1)线性分类器的定义
线性分类器是一种假设特征与分类结果存在线性关系的模型,这个模型通过累加计算每个维度的特征与各自权重的乘积来帮助类别决策。
(2)逻辑斯蒂函数
如果定义![]() 来代表n维特征列向量,同时用n维列向量
来代表n维特征列向量,同时用n维列向量![]() 来代表对应的权重,或者叫做系数;同时为了避免其过坐标原点这种硬性假设,增加一个截距b。由此这种线性关系便可以表达为:
来代表对应的权重,或者叫做系数;同时为了避免其过坐标原点这种硬性假设,增加一个截距b。由此这种线性关系便可以表达为:
![]()
这里的![]() ,取值范围分布在整个实数域中。
,取值范围分布在整个实数域中。
然而,所要处理的最简单的二分类问题希望![]() ;因此需要一个函数把原先的
;因此需要一个函数把原先的![]() 映射到
映射到![]() 。于是想到了逻辑斯蒂函数:
。于是想到了逻辑斯蒂函数:
这里的![]() 并且
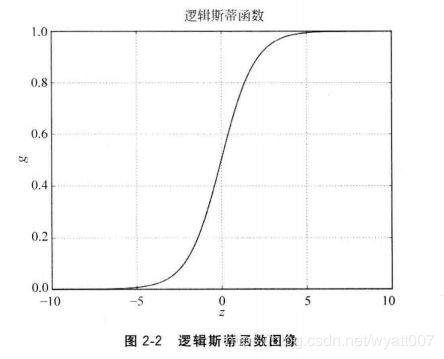
并且![]() ,其函数图像为:
,其函数图像为:
(3)逻辑斯蒂回归模型
综上,如果将z替换为f,整合方程式(1)和方程式(2),就获得了一个经典的线性分类器,逻辑斯蒂回归模型(Logistic Regression):
备注:虽然它叫做“回归”模型,但是事实上这只是一种约定俗成的称谓,事实上它仍然是一种经典的分类模型。
可以观察到该模型如何处理一个待分类的特征向量:如果![]() ,那么
,那么![]() ;若
;若![]() ,则
,则![]() ,这个特征向量被判别为一类;反之,若
,这个特征向量被判别为一类;反之,若![]() ,则
,则![]() ,其被归为另外一类。
,其被归为另外一类。
当使用一组m个用于训练的特征向量![]() 和其所对应的分类目标
和其所对应的分类目标![]() ,希望逻辑斯蒂模型可以在这组训练集上取得最大似然估计的概率
,希望逻辑斯蒂模型可以在这组训练集上取得最大似然估计的概率![]() 。或者说,至少要在训练集上表现如此:
。或者说,至少要在训练集上表现如此:
备注:事实上,任何模型在训练集上的表现都不一定能够代表其最终在未知待测数据集上的性能;但是,至少要先保证模型可以被训练集优化。
为了学习到决定模型的参数,即参数w和截距b。普遍使用一种精确计算的解析算法和一种快速估计的随机梯度上升算法。
备注:事实上,不管是随机梯度上升(SGA)还是随机梯度下降(SGD),都隶属于用梯度法迭代渐进估计参数的过程。梯度上升用于目标最大化,梯度下降用于目标最小化。
2、数据描述
使用“良/恶性乳腺癌肿瘤预测”数据为例子,完整地使用该数据所有的特征作为训练分类器参数的依据,同时采用更为精细的测评指标对模型性能进行评价。
(1)数据下载
原始数据的下载地址为:https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/。
备注:下载文件为:breast-cancer-wisconsin.names。
(2)数据描述
5. Number of Instances: 699 (as of 15 July 1992)
6. Number of Attributes: 10 plus the class attribute
7. Attribute Information: (class attribute has been moved to last column)
# Attribute Domain
-- -----------------------------------------
1. Sample code number id number
2. Clump Thickness 1 - 10
3. Uniformity of Cell Size 1 - 10
4. Uniformity of Cell Shape 1 - 10
5. Marginal Adhesion 1 - 10
6. Single Epithelial Cell Size 1 - 10
7. Bare Nuclei 1 - 10
8. Bland Chromatin 1 - 10
9. Normal Nucleoli 1 - 10
10. Mitoses 1 - 10
11. Class: (2 for benign, 4 for malignant)
8. Missing attribute values: 16
There are 16 instances in Groups 1 to 6 that contain a single missing
(i.e., unavailable) attribute value, now denoted by "?".
9. Class distribution:
Benign: 458 (65.5%)
Malignant: 241 (34.5%)
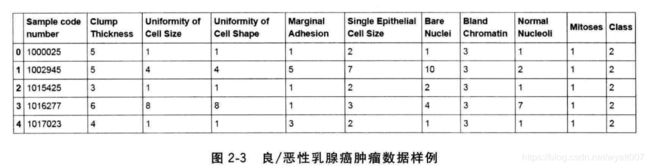
得知该原始数据共有699条样本,每条样本有11列不同的数值:1列用于检索的id,9列与肿瘤相关的医学特征,以及一列表征肿瘤类型的数值。所有9列用于表示肿瘤医学特质的数值均被量化为1~10之间的数字,而肿瘤的类型也借由数字2和数字4分别指代良性与恶性。不过,这份数据也声明其中包含16个缺失值,并且用“?”标出。
(3)数据预处理
# 代码13:良/恶性乳腺癌肿瘤数据预处理
# 导入pandas与numpy工具包。
import pandas as pd
import numpy as np
# 创建特征列表。
column_names = ['Sample code number', 'Clump Thickness', 'Uniformity of Ceil Size', 'Uniformity of Ceil Shape', 'Marginal Adhesion', 'Single Epithelial Ceil Size', 'Bare Nuclei', 'Bland Chromatin', 'Normal Nucleoli', 'Mitoses', 'Class']
# 使用pandas.read_csv函数从互联网读取指定数据。
data = pd.read_csv('https://archive.ics.uci.edu/ml/machine-learning-databases/breast-cancer-wisconsin/breast-cancer-wisconsin.data', names=column_names)
# 将?替换为标准缺失值表示。
data = data.replace(to_replace='?', value=np.nan)
# 丢弃带有缺失值的数据(只要有一个维度有缺失)。
data = data.dropna(how='any')
# 输出data的数据量和维度。
data.shape
print(data.shape)本地输出:
(683, 11)结论:经过简单的处理之后,无缺失值的数据样本共有683条,特征包括细胞厚度、细胞大小、形状等9个维度,并且每个维度的特征均量化为1~10之间的数值进行表示:
(4)训练和测试数据
由于原始数据没有提供对应的测试样本用于评估模型性能,因此需要对带有标记的数据进行分割。通常情况下,25%的数据会作为测试集,其余75%的数据用于训练。
# 代码14:准备良/恶性乳腺癌肿瘤训练、测试数据
# 使用sklearn.model_selection里的train_test_split模块用于分割数据。
from sklearn.model_selection import train_test_split
# 随机采样 25%的数据用于测试,剩下的75%用于构建训练集合。
X_train, X_test, y_train, y_test = train_test_split(data[column_names[1:10]], data[column_names[10]], test_size=0.25, random_state=33)
# 查验训练样本的数量和类别分布。
y_train.value_counts()
print(y_train.value_counts())
# 查验测试样本的数量和类别分布。
y_test.value_counts()
print(y_test.value_counts())备注:原来的导入模型from sklearn.cross_validation import train_test_split的时候,提示错误:
from sklearn.cross_validation import train_test_split
ModuleNotFoundError: No module named 'sklearn.cross_validation'需要替换cross_validation:
from sklearn.model_selection import train_test_split本地输出:
2 344
4 168
Name: Class, dtype: int64
2 100
4 71
Name: Class, dtype: int64结论:用于训练样本共有512条(344条良性肿瘤数据、168条恶性肿瘤数据),测试样本有171条(100条良性肿瘤数据、71条恶性肿瘤数据)。
3、编程实践
使用逻辑斯蒂回归与随机梯度参数估计两种方法对上述处理后的训练数据进行学习,并且根据测试样本特征进行预测。
# 代码15:使用线性分类模型从事良/恶性肿瘤预测任务
# 从sklearn.preprocessing里导入StandardScaler。
from sklearn.preprocessing import StandardScaler
# 从sklearn.linear_model里导入LogisticRegression与SGDClassifier。
from sklearn.linear_model import LogisticRegression
from sklearn.linear_model import SGDClassifier
# 标准化数据,保证每个维度的特征数据方差为1,均值为0。使得预测结果不会被某些维度过大的特征值而主导。
ss = StandardScaler()
X_train = ss.fit_transform(X_train)
X_test = ss.transform(X_test)
# 初始化LogisticRegression与SGDClassifier。
lr = LogisticRegression()
sgdc = SGDClassifier()
# 调用LogisticRegression中的fit函数/模块用来训练模型参数。
lr.fit(X_train, y_train)
# 使用训练好的模型lr对X_test进行预测,结果储存在变量lr_y_predict中。
lr_y_predict = lr.predict(X_test)
# 调用SGDClassifier中的fit函数/模块用来训练模型参数。
sgdc.fit(X_train, y_train)
# 使用训练好的模型sgdc对X_test进行预测,结果储存在变量sgdc_y_predict中。
sgdc_y_predict = sgdc.predict(X_test)4、性能测评
在编程实践中,分别利用LogisticRegression与SGDClassifier针对171条测试样本进行预测工作。由于这171条测试样本拥有正确标记,并记录正在变量y_test中,因此非常直观的做法是比对预测结果和原本正确标记,计算171条测试样本中,预测正确的百分比。把这个百分比称作准确性(Accuracy),并且将其作为评估分类模型的一个重要性能指标。
然而,在许多实际问题中,往往更加关注模型对某一特定类别的预测能力。比如,在“良/恶性肿瘤预测任务”里,医生和患者往往更加关心有多少恶性肿瘤被正确地诊断出来,因为这种肿瘤更加致命。也就是说,在二分类任务下,预测结果和正确标记之间存在4中不同的组合,构成混淆矩阵(Confusion Matrix)。
如果恶性肿瘤为阳性,良性肿瘤为阴性,那么,预测正确的恶性肿瘤即为真阳性,预测正确的良性肿瘤为真阴性;原本是良性肿瘤,误判为恶性肿瘤的为假阳性;而实际是恶性肿瘤,但是预测模型没有检测出来,则为假阴性。
因此,除了准确性之外,还引入了两个评价指标,分别是召回率(Recall)和准确率(Precision),它们的定义分别是:
其中,#(True positive)代表真阳性样本的数量,其余以此类推。此外,为了综合考量召回率与精确率,计算这两个指标的调和平均数,得到F1指标:
对于乳腺癌肿瘤预测的问题,显然更加关注召回率,也就是应该被正确识别的恶性肿瘤的百分比。
# 代码16:使用线性分类模型从事良/恶性肿瘤预测任务的性能分析
# 从sklearn.metrics里导入classification_report模块。
from sklearn.metrics import classification_report
# 使用逻辑斯蒂回归模型自带的评分函数score获得模型在测试集上的准确性结果。
print('Accuracy of LR Classifier:', lr.score(X_test, y_test))
# 利用classification_report模块获得LogisticRegression其他三个指标的结果。
print(classification_report(y_test, lr_y_predict, target_names=['Benign', 'Malignant']))
# 使用随机梯度下降模型自带的评分函数score获得模型在测试集上的准确性结果。
print('Accuracy of SGD Classifier:', lr.score(X_test, y_test))
# 利用classification_report模块获得SGDClassifier其他三个指标的结果。
print(classification_report(y_test, sgdc_y_predict, target_names=['Benign', 'Malignant']))本地输出:
Accuracy of LR Classifier: 0.9883040935672515
precision recall f1-score support
Benign 0.99 0.99 0.99 100
Malignant 0.99 0.99 0.99 71
accuracy 0.99 171
macro avg 0.99 0.99 0.99 171
weighted avg 0.99 0.99 0.99 171
Accuracy of SGD Classifier: 0.9707602339181286
precision recall f1-score support
Benign 0.95 1.00 0.98 100
Malignant 1.00 0.93 0.96 71
accuracy 0.97 171
macro avg 0.98 0.96 0.97 171
weighted avg 0.97 0.97 0.97 171结论:LogisticRegression比起SGDClassifier在测试集上表现有更高的准确性。这是因为Scikit-learn中采用解析的方式精确计算LogisticRegression的参数,而使用梯度法估计SGDClassifier的参数。
5、特点分析
所使用的模型包括LogisticRegression与SGDClassifier。相比之下,前者对参数的计算采用精确解析的方式,计算时间长但是模型性能略高;后者采用随机梯度上升算法估计模型参数,计算时间短但是产出的模型性能略低。一般而言,对于训练数据规模在10万量级以上的数据,考虑到时间的耗用,更加推荐使用随机梯度算法对模型参数进行估计。