QIIME 2用户文档. 4人体各部位微生物组分析实战Moving Pictures(2018.11)
文章目录
- 前情提要
- QIIME 2用户文档. 4人体各部位微生物组
- 启动QIIME2运行环境
- 样本元数据
- 下载和导入数据
- 拆分样品
- 序列质控和生成特征表
- 方法1. DADA2
- 方法2. Deblur
- 特征表和特征序列汇总
- 构建进化树用于多样性分析
- Alpha和beta多样性分析
- Alpha稀释取线
- 物种组成分析
- 使用ANCOM差异丰度分析
- 译者简介
- Reference
- 猜你喜欢
- 写在后面
前情提要
- 文章导读:QIIME 2可重复、交互和扩展的微生物组数据分析流程
- 1简介和安装
- 2插件工作流程概述
- 3老司机上路指南
QIIME 2用户文档. 4人体各部位微生物组
“Moving Pictures” tutorial
https://docs.qiime2.org/2018.11/tutorials/moving-pictures/
注意:本文学习需要安装好QIIME 2,请务必完成《1简介和安装》
在本教程中,你将使用QIIME 2在五个时间点对来自四个身体部位的两个人的微生物组样本进行分析,第一个时间点紧接着是抗生素的使用。基于这些样本的研究文章《Moving pictures of the human microbiome》在2011年发表于Genome Biology。本教程中使用的数据基于Illumina HiSeq产出,使用地球微生物组项目扩增16S rRNA基因高变区4(V4)测序的方法。
对于熟悉QIIME 1的用户,本数据也出现在QIIME的教程中。
在开始本教程前,我们需要进入工作环境创建新目录并进入
启动QIIME2运行环境
对于上文提到了两种常用安装方法,我们每次在分析数据前,需要打开工作环境,根据情况选择对应的打开方式。
# 创建qiime2学习目录并进入
mkdir -p qiime2
cd qiime2
# Miniconda安装的请运行如下命令加载工作环境
source activate qiime2-2018.11
# 如果是docker安装的请运行如下命令,默认加载当前目录至/data目录
docker run --rm -v $(pwd):/data --name=qiime -it qiime2/core:2018.11
# 创建本节学习目录
mkdir qiime2-moving-pictures-tutorial
cd qiime2-moving-pictures-tutorial
样本元数据
Sample metadata
在开始分析之前,探索样本元数据,以熟悉本研究中使用的样本。示例元数据作为Google 表格提供。你可以通过选择 File > Download as > Tab-separated values,以制表符分隔的文本格式下载该文件。或者,以下命令将作为制表符分隔的文本下载示例元数据,并将其保存在文件sample-metadata.tsv,这个sample-metadata.tsv文件在本教程中一直被用到。
wget \
-O "sample-metadata.tsv" \
"https://data.qiime2.org/2018.11/tutorials/moving-pictures/sample_metadata.tsv"
注意:QIIME 2 官方测试数据均保存在Google服务器上,国内下载比较困难。可使用代理服务器(如蓝灯)下载此链接 https://data.qiime2.org/2018.11/tutorials/moving-pictures/sample_metadata.tsv ,或公众号后台回复"qiime2"获取测试数据批量下载链接,你还可以跳过以后的wget步骤。
提示:Keemei是一个用于验证示例元数据的Google Sheets插件。在开始任何分析之前,样本元数据的验证非常重要。尝试按照Keemei网站上的说明安装Keemei,然后验证上面链接的示例元数据电子表格。该电子表格还包括一个带有一些无效数据的表格,以便使用Keemei进行测试。
提示:要了解关于元数据的更多信息,包括如何格式化元数据以便与QIIME 2一起使用,请参阅元数据教程。
下载和导入数据
Obtaining and importing data
下载在本次分析中使用的序列。在本教程中,我们将处理完整的序列数据的一小部分,以便命令能够快速运行(减少等待时间)。
创建子目录并下载实验测序数据,无法下载,请公众号后台回复"qiime2"获取测试数据备用下载链接。
mkdir -p emp-single-end-sequences
wget -O "emp-single-end-sequences/barcodes.fastq.gz" "https://data.qiime2.org/2018.11/tutorials/moving-pictures/emp-single-end-sequences/barcodes.fastq.gz"
wget -O "emp-single-end-sequences/sequences.fastq.gz" "https://data.qiime2.org/2018.11/tutorials/moving-pictures/emp-single-end-sequences/sequences.fastq.gz"
用于输入到QIIME 2的所有数据都以QIIME 2对象的形式出现,其中包含有关数据类型和数据源的信息。因此,我们需要做的第一件事是将这些序列数据文件导入到QIIME 2对象中。
这个QIIME 2对象的语义类型是EMPSingleEndSequences。 QIIME 对象EMPSingleEndSequences是包含多样本混合的序列文件,这意味着序列尚未分配给样本(因此包括sequences.fastq.gz和barcode.fastq.gz文件,其中barcode.fastq.gz包含与sequences.fastq.gz中的每个序列相关联的条形码)。要导入其他格式的序列数据,请参阅导入数据教程。
导入数据:生成qiime2要求的对象格式
qiime tools import \
--type EMPSingleEndSequences \
--input-path emp-single-end-sequences \
--output-path emp-single-end-sequences.qza
输出对象:
emp-single-end-sequences.qza: 查看 | 下载
注意:公众号无法打开外部链接,想要直接访问查看、下载链接,可访问QIIME 2论坛、CSDN或科学网阅读同名文档,可用百度搜索本节标题直达。
提示:
上面的查看和下载由文档中的命令创建的QIIME 2对象和可视化链接。例如,上面的命令创建了单个emp-single-end-sequences.qza文件,上面链接了相应的预计算文件(输出结果)。你可以查看预计算的QIIME 2对象和可视化而不需要安装额外的软件(例如,QIIME 2)。
QIIME 1用户:
在QIIME 1中,我们一般建议通过QIIME执行样本拆分(例如,使用split_libraries.py或split_libraries_fastq.py),因为这个步骤还执行序列的质量控制。现在我们将样本拆分和质量控制步骤分开,因此你可以使用混合多样本序列(如我们在此所做的)或拆分后的序列开始QIIME 2分析。
拆分样品
Demultiplexing sequences
为了混合序列进行样本拆分,我们需要知道哪个条形码序列与每个样本相关联。此信息包含在示例元数据文件中。你可以运行以下命令来对序列进行样本拆分(demux emp-single命令指的是这些序列是根据地球微生物学方法添加的条形码,并且是单端序列)。QIIME 2对象demux.qza包含样本拆分后的序列。
qiime demux emp-single \
--i-seqs emp-single-end-sequences.qza \
--m-barcodes-file sample-metadata.tsv \
--m-barcodes-column BarcodeSequence \
--o-per-sample-sequences demux.qza
输出结果:demux.qza
在样本拆分之后,生成拆分结果的摘要是很有用的。这允许你确定每个样本获得多少序列,并且还可以获得序列数据中每个位置处序列质量分布的摘要。
结果统计
qiime demux summarize \
--i-data demux.qza \
--o-visualization demux.qzv
输出可视化结果demux.qzv: 查看 | 下载
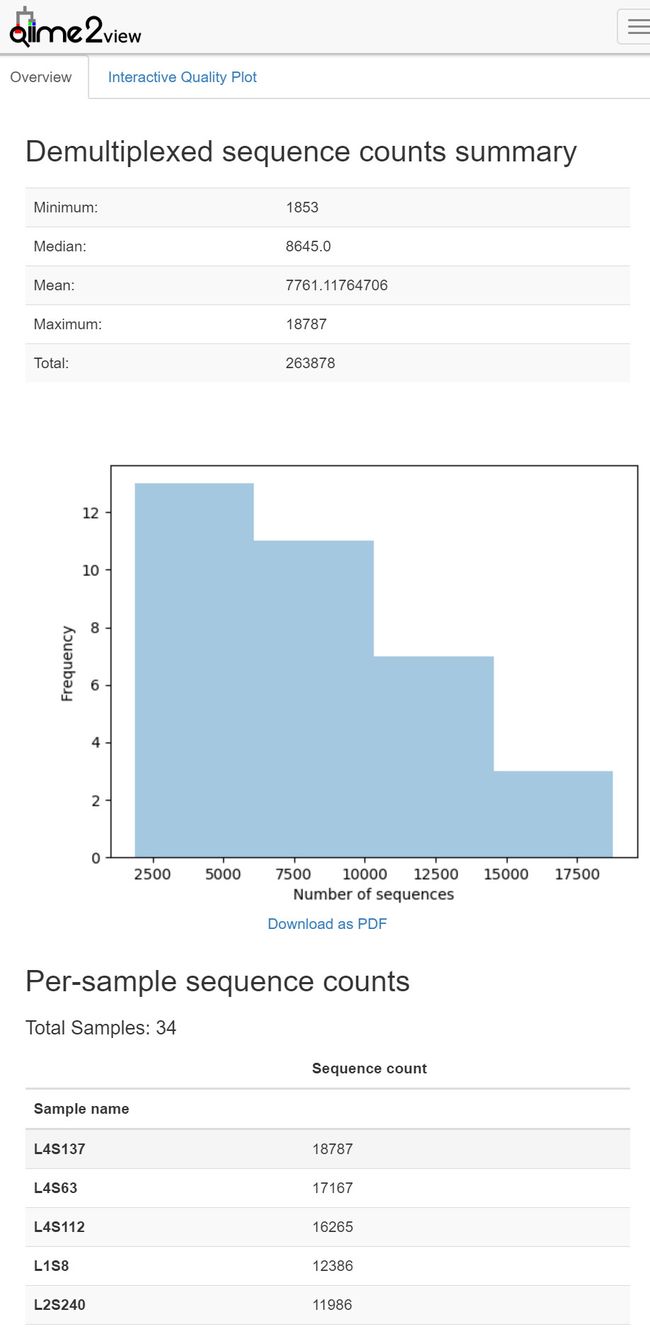
图1. 样本拆分结果统计结果——样本数据量可视化图表。主要分为三部分:上部为摘要;中部为样本不同数据量分布频率柱状图,可下载PDF,下部为每个样本的测序量。上方面板还可切换至交互式质量图Interactive Qaulity Plot
图2. 交互式质量图Interactive Qaulity Plot查看页面。同样为三部分:上部为每个位置碱基的质量分布交互式箱线图,鼠标点击在方面(中部)文字和表格中显示鼠标所在位置碱基的详细信息;下部为拆分样本的长度摘要(一般无差别,略)。
所有QIIME 2可视化对象(即,使用--o-visualization参数指定的文件)将生成一个.qzv文件。你可以使用qiime tools view查看这些文件。我们提供了用于查看可视化的第一个命令,但是对于本教程的其余部分,我们将告诉你在运行可视化程序之后查看结果可视化,这意味着你应该在生成的.qzv文件上运qiime tools view。
qiime tools view demux.qzv
这条命令的显示需要图形界面的支持,如在有图型界面的Linux上,但仅使用SSH登陆方式无法显示图形。
推荐使用 https://view.qiime2.org 网址显示结果
可选使用[XShell+XManager]支持SSH方式的图型界面(https://mp.weixin.qq.com/s/rbh3jynnq8E9tuhyCLtRIA)、虚拟机或服务器远程桌面方式支持上面命令的图形结果。
目前命令行方式想要查看结果可能很多使用服务器人员无法实现 (即依赖服务器安装了桌面,本地依赖XShell+XManager或其它ssh终端和图形界面软件)
本地查看可解压.qzv,目录中的data目录包括详细的图表文件,主要关注 pdf 和 html 文件,目录结构如下。
── demux
└── 8743ab13-72ca-4adf-9b6c-d97e2dbe8ee3
├── checksums.md5
├── data
│ ├── data.jsonp
│ ├── demultiplex-summary.pdf
│ ├── demultiplex-summary.png
│ ├── dist
│ │ ├── bundle.js
│ │ ├── d3-license.txt
│ │ └── vendor.bundle.js
│ ├── forward-seven-number-summaries.csv
│ ├── index.html
│ ├── overview.html
│ ├── per-sample-fastq-counts.csv
│ ├── q2templateassets
│ │ ├── css
│ │ │ ├── bootstrap.min.css
│ │ │ ├── normalize.css
│ │ │ └── tab-parent.css
│ │ ├── fonts
│ │ │ ├── glyphicons-halflings-regular.eot
│ │ │ ├── glyphicons-halflings-regular.svg
│ │ │ ├── glyphicons-halflings-regular.ttf
│ │ │ ├── glyphicons-halflings-regular.woff
│ │ │ └── glyphicons-halflings-regular.woff2
│ │ ├── img
│ │ │ └── qiime2-rect-200.png
│ │ └── js
│ │ ├── bootstrap.min.js
│ │ ├── child.js
│ │ ├── jquery-3.2.0.min.js
│ │ └── parent.js
│ └── quality-plot.html
├── metadata.yaml
├── provenance
│ ├── action
│ │ └── action.yaml
│ ├── artifacts
│ │ ├── 9594ef07-c414-4658-9345-c726de100d8d
│ │ │ ├── action
│ │ │ │ └── action.yaml
│ │ │ ├── citations.bib
│ │ │ ├── metadata.yaml
│ │ │ └── VERSION
│ │ └── a7a882f3-5e4f-4b5e-8a35-6a1098d21608
│ │ ├── action
│ │ │ ├── action.yaml
│ │ │ └── barcodes.tsv
│ │ ├── citations.bib
│ │ ├── metadata.yaml
│ │ └── VERSION
│ ├── citations.bib
│ ├── metadata.yaml
│ └── VERSION
└── VERSION
qzv文件解压后文件详细
序列质控和生成特征表
Sequence quality control and feature table construction
QIIME 2插件多种质量控制方法可选,包括DADA2、Deblur和基于基本质量分数的过滤。在本教程中,我们使用DADA2和Deblur介绍这个步骤。这些步骤是可互相替换的,因此你可以使用自己喜欢的方法。这两种方法的结果将是一个QIIME 2对象FeatureTable[Frequency]和一个FeatureData[Sequence],Frequency对象包含数据集中每个样本中每个唯一序列的计数(频率),Sequence对象将FeatureTable中的特征ID与序列对应。
译者注:此步主要有DADA2和Deblur两种方法可选,推荐使用DADA2,去年发表在Nature Method上,比较同类方法优于其它OTU聚类结果;相较QIIME的UPARSE聚类方法,目前DADA2方法仅去噪去嵌合,不再按相似度聚类。比上一代分析结果更精确。
注意:本节中此次存在两种可选方法时,你将创建具有特定方法名称的对象(例如,使用dada2去噪生成的特性表将被称为
table-dada2.qza)。在创建这些对象之后,你将把两个选项之一的对象重命名为更通用的文件名(例如,table.qza)。为对象创建特定名称,然后对其进行重命名的过程仅允许你选择在本步骤中使用的两个选项中之一完成教程,而不必再次关注该选项。需要注意的是,在这个步骤或QIIME 2中的任何步骤中,你给对象或可视化的文件命名并不重要。
QIIME1 用户注意:
QIIME 2对象FeatureTable[Frequency]等价于QIIME 1 OTU或BIOM表,QIIME 2对象FeatureData[Sequence]等价于QIIME 1代表序列文件。由于DADA2和Deblur产生的“OTU”是通过对唯一序列进行分组而创建的,因此这些OTU相当于来自QIIME 1的100%相似度的OTU,通常称为序列变体。在QIIME 2中,这些OTU比QIIME 1默认的97%相似度聚类的OTU具有更高的分辨率,并且它们具有更高的质量,因为这些质量控制步骤比QIIME 1中实现更好。因此,与QIIME 1相比,可以对样本的多样性和分类组成进行更准确的估计。
方法1. DADA2
DADA2是用于检测和校正(如果有可能的话)Illumina扩增序列数据的工作流程。正如在q2-dada2插件中实现的,这个质量控制过程将过滤掉在测序数据中识别的任何phiX序列(通常存在于标记基因Illumina测序数据中,用于提高扩增子测序质量),并同时过滤嵌合序列。
译者注:DADA2是Susan P. Holmes于2016年发表于Nature Methods的文章,截止18年12月22号Google学术统计引用483次,关于教授的工作介绍,详见《斯坦福大学统计系教授带你玩转微生物组分析》;关于dada2的介绍,详见《16S测序,不知道OTU你就out了!》;引文: Callahan, Benjamin J., Paul J. McMurdie, Michael J. Rosen, Andrew W. Han, Amy Jo A. Johnson, and Susan P. Holmes. “DADA2: high-resolution sample inference from Illumina amplicon data.” Nature methods 13, no. 7 (2016): 581.
dada2 denoise-single方法需要两个用于质量过滤的参数:--p-trim-left m,它去除每个序列的前m个碱基(如引物、barcode);--p-trunc-len n,它在位置n截断每个序列。这允许用户去除序列的低质量区域、引物等。为了确定要为这两个参数传递什么值,你应该查看上面由qiime demux summarize生成的demux.qzv文件中的交互质量图选项卡。
读者思考时间:基于上图对拆分样品的统计结果,如何设置
--p-trunc-len和--p-trim-left的参数值。
- –p-trim-left 截取左端低质量序列,我们看上图中箱线图,左端质量都很高,无低质量区,设置为0;
- –p-trunc-len 序列截取长度,也是为了去除右端低质量序列,我们看到大于120以后,质量下降极大,甚至中位数都下降至20以下,需要全部去除,综合考虑决定设置为120。
单端序列去噪, 输入样本拆分后结果;去除左端 0 bp (–p-trim-left,有时用于切除低质量序列、barocde或引物),序列切成 120 bp 长(–p-trunc-len);生成代表序列、特征表和去噪过程统计。
qiime dada2 denoise-single \
--i-demultiplexed-seqs demux.qza \
--p-trim-left 0 \
--p-trunc-len 120 \
--o-representative-sequences rep-seqs-dada2.qza \
--o-table table-dada2.qza \
--o-denoising-stats stats-dada2.qza
生成三个输出文件:
- stats-dada2.qza: dada2计算统计结果。查看 | 下载
- table-dada2.qza: 特征表。查看 | 下载
- rep-seqs-dada2.qza: 代表序列。 查看 | 下载
对特征表统计进行进行可视化
qiime metadata tabulate \
--m-input-file stats-dada2.qza \
--o-visualization stats-dada2.qzv
输出可视化结果:stats-dada2.qzv,特征表统计结果,查看 | 下载
内容为每个样本,输入、过滤、去噪和非嵌合的统计
sample-id input filtered denoised non-chimeric
L6S93 11270 7483 7483 7025
L6S68 9554 6169 6169 6022
我们的下游分析,将继续使用dada2的结果,需要将它们改名方便继续分析:
mv rep-seqs-dada2.qza rep-seqs.qza
mv table-dada2.qza table.qza
方法2. Deblur
Deblur使用序列错误配置文件将错误的序列与从其来源的真实生物序列相关联,从而得到高质量的序列变异数据,主要为两个步骤。首先,应用基于质量分数的初始质量过滤过程,是Bokulich等人2013年发表的质量过滤方法。
详者注:Deblur是本软件作者作为通讯作者2013发表于Nature Methods的重要扩增子代表序列鉴定方法,截止18年12月22号Google学术统计引用940次,
引文:Bokulich, Nicholas A., et al. “Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing.” Nature methods 10.1 (2013): 57. 作者只将自己的方法作为了备选,分析教程中首选了dada2方法。
按测序碱基质量过滤序列
qiime quality-filter q-score \
--i-demux demux.qza \
--o-filtered-sequences demux-filtered.qza \
--o-filter-stats demux-filter-stats.qza
输出对象:
- demux-filtered.qza: 序列质量过滤后结果。 查看 | 下载
- demux-filter-stats.qza: 序列质量过滤后结果统计。 查看 | 下载
注意:在Deblur的论文中,作者使用了当时推荐的过滤参数。而这里使用的参数基于最新的经验,效果更好。
接下来,使qiime deblur denoise-16S方法应用于Deblur工作流程。此方法需要一个用于质量过滤的参数,即截断位置n长度的序列的--p-trim-length n。通常,Deblur开发人员建议将该值设置为质量分数中位数开始下降至低质量区时的长度。在本次数据上,质量图(在质量过滤之前)表明合理的选择是在115至130序列位置范围内。这是一个主观的评估。你可能不采用该建议的一种原因是存在多个批次测序的元分析时。在这种情况的元分析中,比较所有批次的序列长度是否相同,以避免人为引入特定的偏差,全局考虑这些是非常重要的。由于我们已经使用修剪长度为120 bp用于qiime dada2 denoise-single分析,并且由于120 bp是基于质量图的结果,这里我们将使用--p-trim-length 120参数。下一个命令可能需要10分钟才能运行完成。
详者注:deblur最大缺点就是慢,本次只分析了33个样品,共177,092条序列。而实际研究中大项目会有成千上万的样本,1亿-10亿条序列,此步分析可能需要几个月甚至根本无法完成,不推荐。
deblur去噪16S过程,输入文件为质控后的序列,设置截取长度参数,生成结果文件有代表序列、特征表、样本统计。
time qiime deblur denoise-16S \
--i-demultiplexed-seqs demux-filtered.qza \
--p-trim-length 120 \
--o-representative-sequences rep-seqs-deblur.qza \
--o-table table-deblur.qza \
--p-sample-stats \
--o-stats deblur-stats.qza
注:在我的服务器上单线程运行了3m24s完成。
输出结果:
- deblur-stats.qza: 过程统计
- table-deblur.qza: 特征表
- rep-seqs-deblur.qza: 代表序列
注意: 本节中使用的两个命令生成QIIME 2对象包含汇总统计信息。为了查看这些汇总统计数据,你可以分别使用qiime元数据表和
qiime deblur visualize-stats命令来可视化它们。
qiime metadata tabulate \
--m-input-file demux-filter-stats.qza \
--o-visualization demux-filter-stats.qzv
qiime deblur visualize-stats \
--i-deblur-stats deblur-stats.qza \
--o-visualization deblur-stats.qzv
输出结果:
- demux-filter-stats.qzv: 质量过程统计表,同上面统计表类似 查看 | 下载
- deblur-stats.qzv: deblur分析统计表,有分析中每个步骤的统计表 查看 | 下载
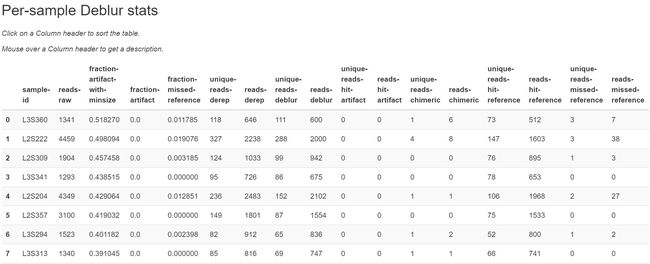
图3. deblur去噪和鉴定ASV处理过程统计结果
如果你想用此处结果下游分析,可以改名为下游分析的起始名称:
mv rep-seqs-deblur.qza rep-seqs.qza
mv table-deblur.qza table.qza
记住,以上两种方法只选择一种即可。推荐dada2速度更快一些。有精力的条件下,可以比较一下两种方法哪个结果更适合自己。其实每种方法都有存在的意义,而且也有适用的范围,要在具体的项目中,结合背景知识分析哪种方法结果更好时才知道。
特征表和特征序列汇总
FeatureTable and FeatureData summaries
在质量筛选步骤完成之后,你将希望探索数据结果。可以使用以下两个命令进行此操作,这两个命令将创建数据的可视化摘要。特性表汇总命令(feature-table summarize)将向你提供关于与每个样品和每个特性相关联的序列数量、这些分布的直方图以及一些相关的汇总统计数据的信息。特征表序列表格feature-table tabulate-seqs命令将提供特征ID到序列的映射,并提供链接以针对NCBI nt数据库轻松BLAST每个序列。当你想要了解关于数据集中重要的特定特性的更多信息时,可视化将在本教程的后续分析中非常有用。
qiime feature-table summarize \
--i-table table.qza \
--o-visualization table.qzv \
--m-sample-metadata-file sample-metadata.tsv
qiime feature-table tabulate-seqs \
--i-data rep-seqs.qza \
--o-visualization rep-seqs.qzv
输出结果:
- table.qzv: 特征表统计。查看 | 下载
- rep-seqs.qzv: 代表序列统计。查看 | 下载
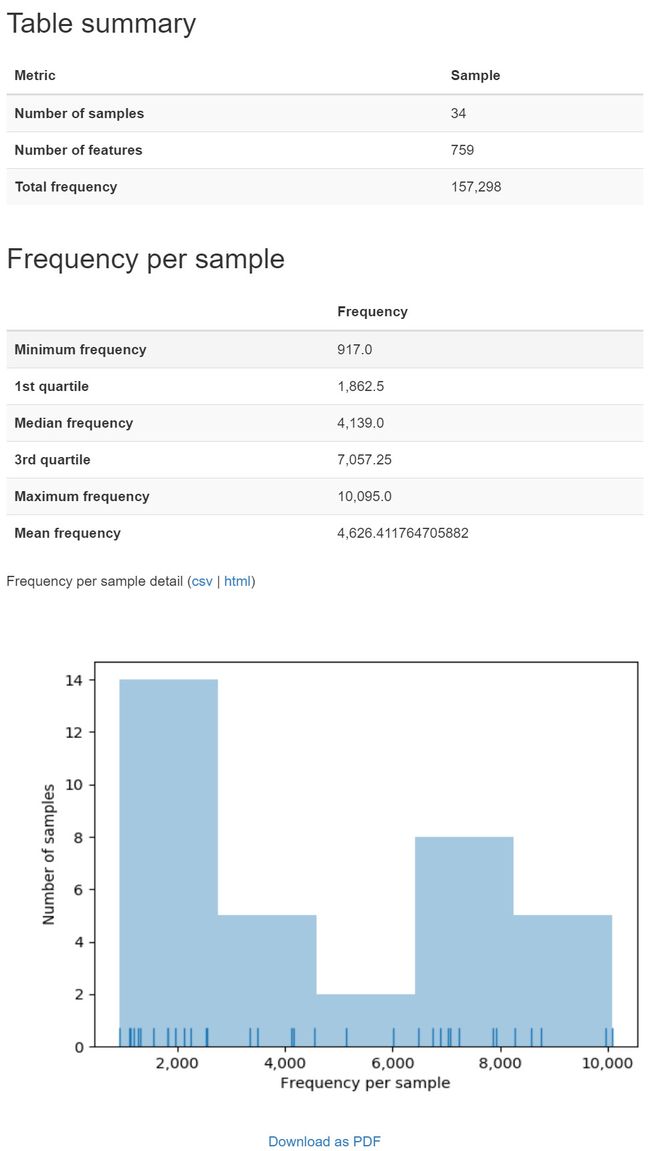
图4. 图中展示了特征表的统计结果
上为摘要、中间为样本数据量分布和图,下方为特征出现频率的统计表和图。
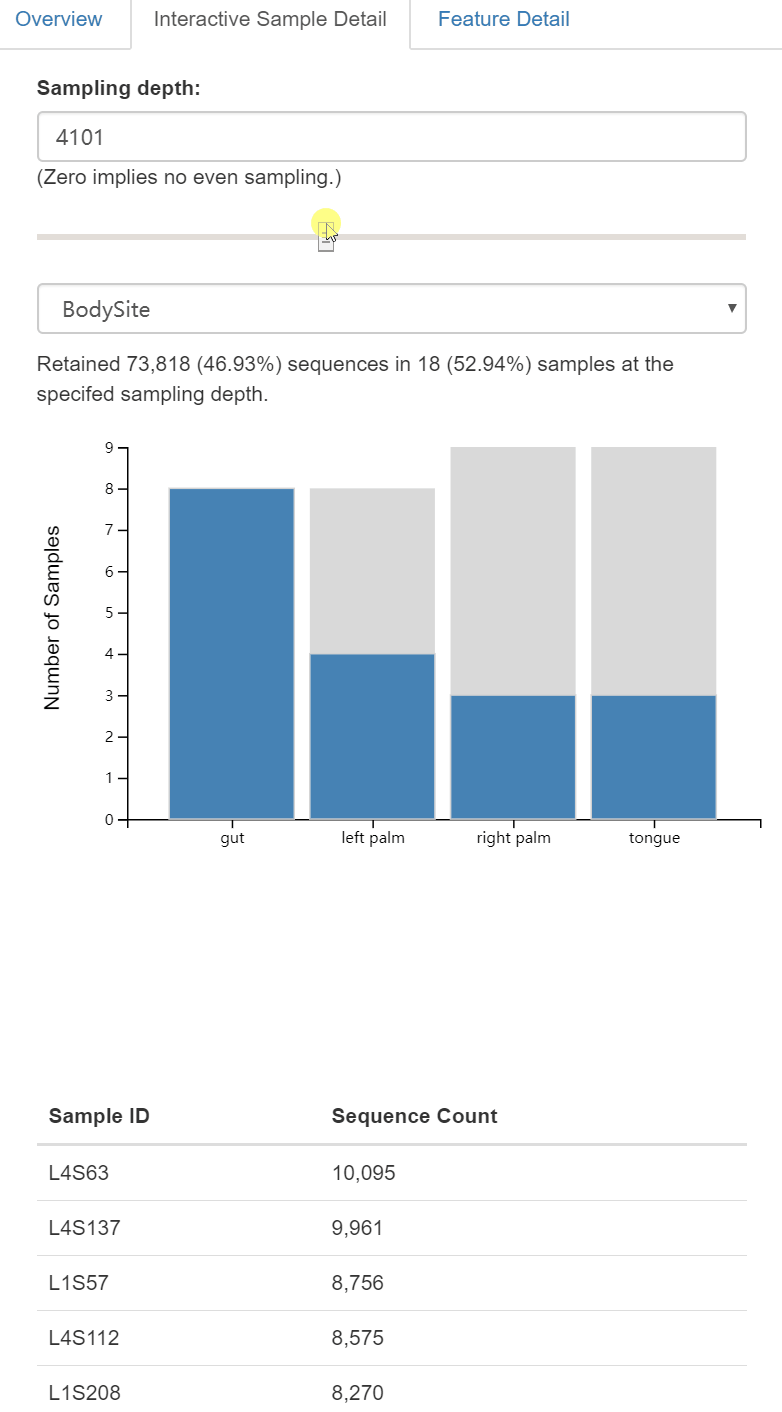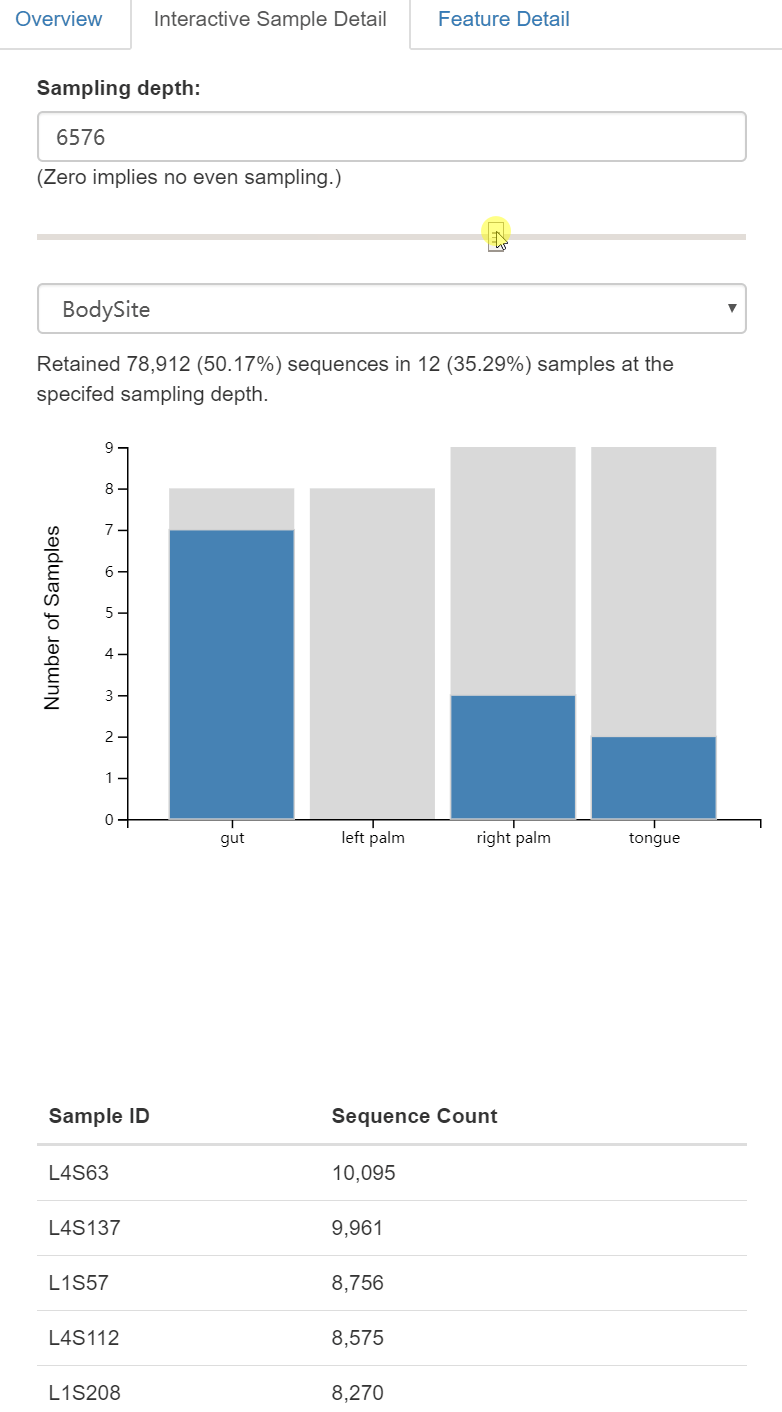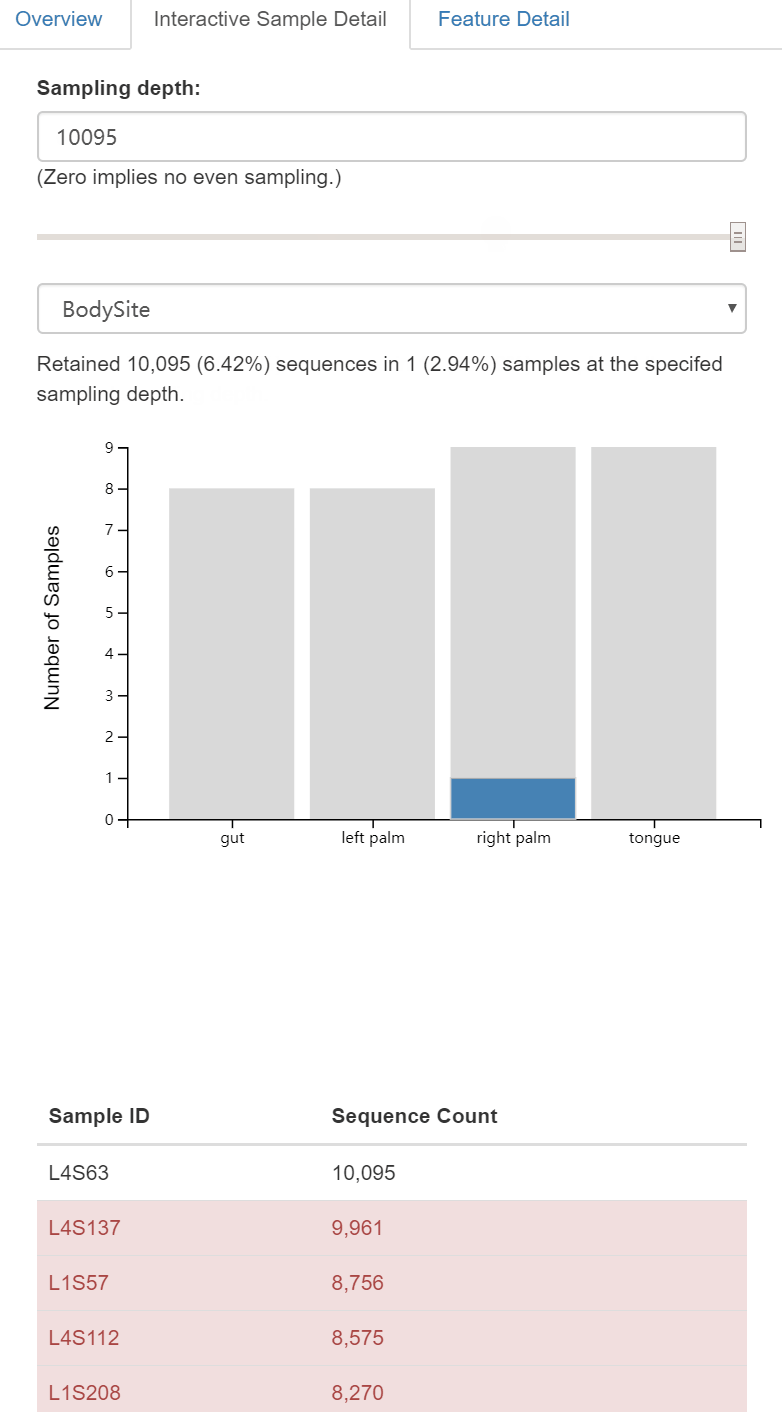
图5. 交互式查看每组剩余样本量
右侧还有Feature Detail进一步查看每个特征的频率和在样本中出现的次数
构建进化树用于多样性分析
Generate a tree for phylogenetic diversity analyses
QIIME 2支持几种系统发育多样性度量方法,包括Faith’s Phylogenetic Diversity、weighted和unweighted UniFrac。除了每个样本的特征计数(即QIIME2对象FeatureTable[Frequency])之外,这些度量还需要将特征彼此关联结合有根进化树。此信息将存储在一个QIIME 2对象的有根系统发育对象Phylogeny[Rooted]中。为了生成系统发育树,我们将使用q2-phylogeny插件中的align-to-tree-mafft-fasttree工作流程。
首先,工作流程使用mafft程序执行对FeatureData[Sequence]中的序列进行多序列比对,以创建QIIME 2对象FeatureData[AlignedSequence]。接下来,流程屏蔽(mask或过滤)对齐的的高度可变区(高变区)。这些位置通常被认为会增加系统发育树的噪声。随后,流程应用FastTree基于过滤后的比对结果生成系统发育树。FastTree程序创建的是一个无根树,因此在本节的最后一步中,应用根中点法将树的根放置在无根树中最长端到端距离的中点,从而形成有根树。
qiime phylogeny align-to-tree-mafft-fasttree \
--i-sequences rep-seqs.qza \
--o-alignment aligned-rep-seqs.qza \
--o-masked-alignment masked-aligned-rep-seqs.qza \
--o-tree unrooted-tree.qza \
--o-rooted-tree rooted-tree.qza
输出结果文件:
- aligned-rep-seqs.qza: 多序列比对结果
- masked-aligned-rep-seqs.qza: 过滤去除高变区后的多序列比对结果
- rooted-tree.qza: 有根树,用于多样性分析
- unrooted-tree.qza: 无根树
Alpha和beta多样性分析
Alpha and beta diversity analysis
QIIME 2的多样性分析可以通过q2-diversity插件实现,该插件支持计算α和β多样性指数、并应用相关的统计检验以及生成交互式可视化图表。我们将首先应用core-metrics-phylogenetic方法,该方法将FeatureTable[Frequency](特征表[频率])抽平到用户指定的测序深度,计算常用几种α和β多样性指数,并使用Emperor为每个β多样性指数生成主坐标分析(PCoA)图。默认情况下计算的方法有:
划重点:理解下面4种alpha和beta多样性指数的所代表的生物学意义至关重要。
- α多样性
- 香农(Shannon’s)多样性指数(群落丰富度的定量度量,即包括丰富度
richness和均匀度evenness两个层面) - Observed OTUs(群落丰富度的定性度量,只包括丰富度)
- Faith’s系统发育多样性(包含特征之间的系统发育关系的群落丰富度的定性度量)
- 均匀度(或 Pielou’s均匀度;群落均匀度的度量)
- 香农(Shannon’s)多样性指数(群落丰富度的定量度量,即包括丰富度
- β多样性
- Jaccard距离(群落差异的定性度量,即只考虑种类,不考虑丰度)
- Bray-Curtis距离(群落差异的定量度量)
- 非加权UniFrac距离(包含特征之间的系统发育关系的群落差异定性度量)
- 加权UniFrac距离(包含特征之间的系统发育关系的群落差异定量度量)
需要提供给这个脚本的一个重要参数是--p-sampling-depth,它是指定重采样(即稀疏/稀释)深度。因为大多数多样指数对不同样本的不同测序深度敏感,所以这个脚本将随机地将每个样本的测序量重新采样至该参数提供的值。例如,如果提供--p-sampling-depth 500,则此步骤将对每个样本中的计数进行无放回抽样,从而使得结果表中的每个样本的总计数为500。如果任何样本的总计数小于该值,那么这些样本将从多样性分析中删除。选择这个值很棘手。我们建议你通过查看上面创建的表table.qzv文件中呈现的信息并选择一个尽可能高的值(因此每个样本保留更多的序列)同时尽可能少地排除样本来进行选择。
读者思考时间:
查看QIIME 2的table.qzv对象,尤其是交互式可视化表格。对于采样深度p,应该选择什么值呢?根据这个选择,分析中多少个样本将被排除?在core-metrics-phylogenetic命令中,你将分析的总序列是多少条呢?
译者注:下面多样性分析,需要基于标准化的特征表,标准化采用无放回重抽样至序列一致,如何设计样品重抽样深度参数
--p-sampling-depth呢?
如是数据量都很大,选最小的即可。如果有个别数据量非常小,去除最小值再选最小值。比如此分析最小值为917,我们选择1109深度重抽样,即保留了大部分样品用于分析,又去除了数据量过低的异常值。
注:本示例为近10年前测序技术的通量水平,现在看来数据量很小。目录一般采用HiSeq PE250测序,数据量都非常大,通常可以采用3万或5万的标准筛选,仍可保留90%以上样本。过低或过高一般结果也会波动较大,不建议放在一起分析。
计算核心多样性
qiime diversity core-metrics-phylogenetic \
--i-phylogeny rooted-tree.qza \
--i-table table.qza \
--p-sampling-depth 1109 \
--m-metadata-file sample-metadata.tsv \
--output-dir core-metrics-results
输出对象(数据文件):
- core-metrics-results/faith_pd_vector.qza: Alpha多样性考虑进化的faith指数
- core-metrics-results/unweighted_unifrac_distance_matrix.qza: 无权重unifrac距离矩阵
- core-metrics-results/bray_curtis_pcoa_results.qza: 基于Bray-Curtis距离PCoA的结果
- core-metrics-results/shannon_vector.qza: Alpha多样性香农指数
- core-metrics-results/rarefied_table.qza: 抽样后的特征表
- core-metrics-results/weighted_unifrac_distance_matrix.qza: 有权重的unifrac距离矩阵
- core-metrics-results/jaccard_pcoa_results.qza: jaccard距离PCoA结果
- core-metrics-results/observed_otus_vector.qza: Alpha多样性observed otus指数
- core-metrics-results/weighted_unifrac_pcoa_results.qza: 基于有权重的unifrac距离的PCoA结果
- core-metrics-results/jaccard_distance_matrix.qza: jaccard距离矩阵
- core-metrics-results/evenness_vector.qza: Alpha多样性均匀度指数
- core-metrics-results/bray_curtis_distance_matrix.qza: Bray-Curtis距离矩阵
- core-metrics-results/unweighted_unifrac_pcoa_results.qza: 无权重的unifrac距离的PCoA结果
输出对象(可视化结果):
- core-metrics-results/unweighted_unifrac_emperor.qzv: 无权重的unifrac距离PCoA结果采用emperor可视化。 查看 | 下载
- core-metrics-results/jaccard_emperor.qzv: jaccard距离PCoA结果采用emperor可视化。查看 | 下载
- core-metrics-results/bray_curtis_emperor.qzv: Bray-Curtis距离PCoA结果采用emperor可视化。查看 | 下载
- core-metrics-results/weighted_unifrac_emperor.qzv: 有权重的unifrac距离PCoA结果采用emperor可视化。查看 | 下载
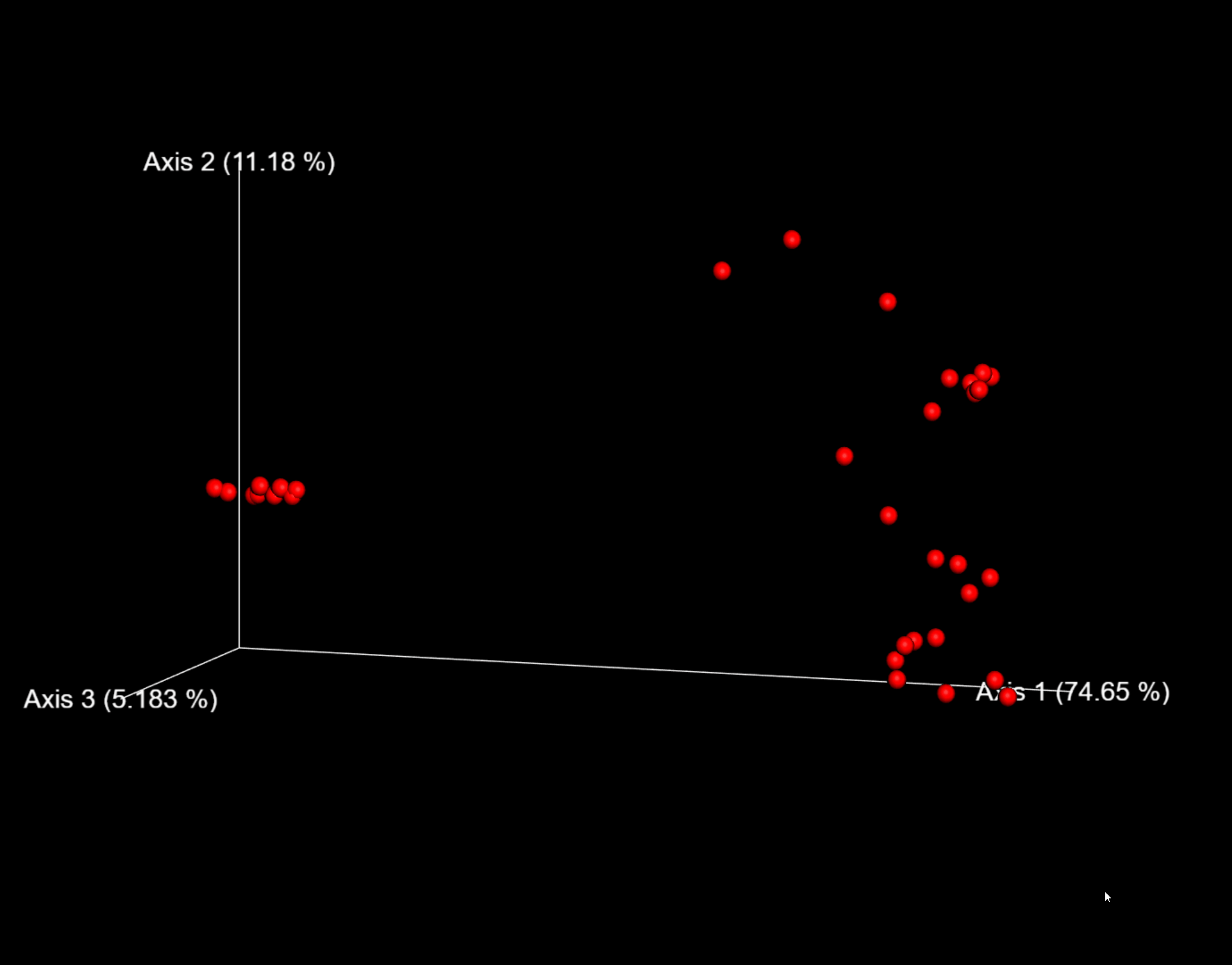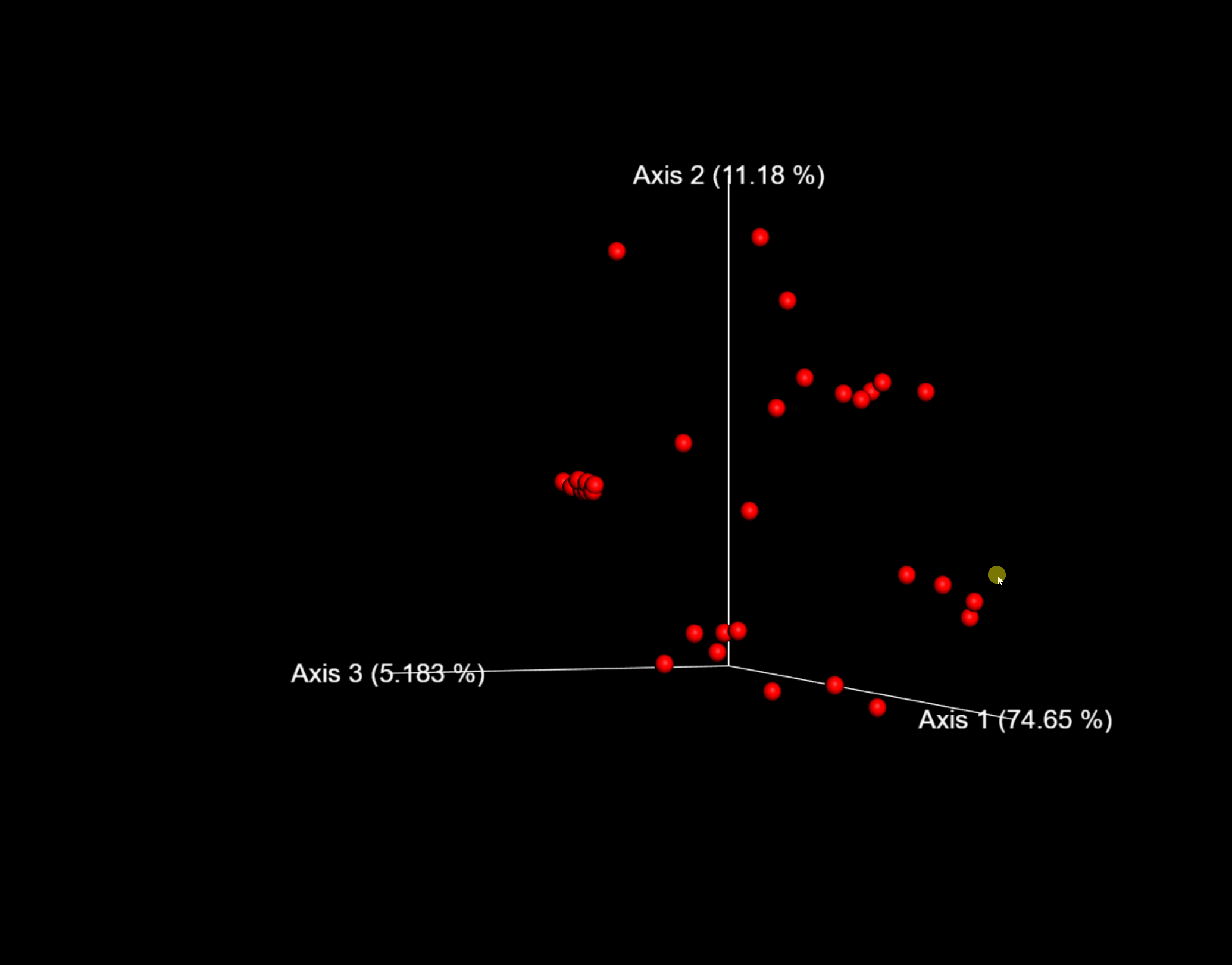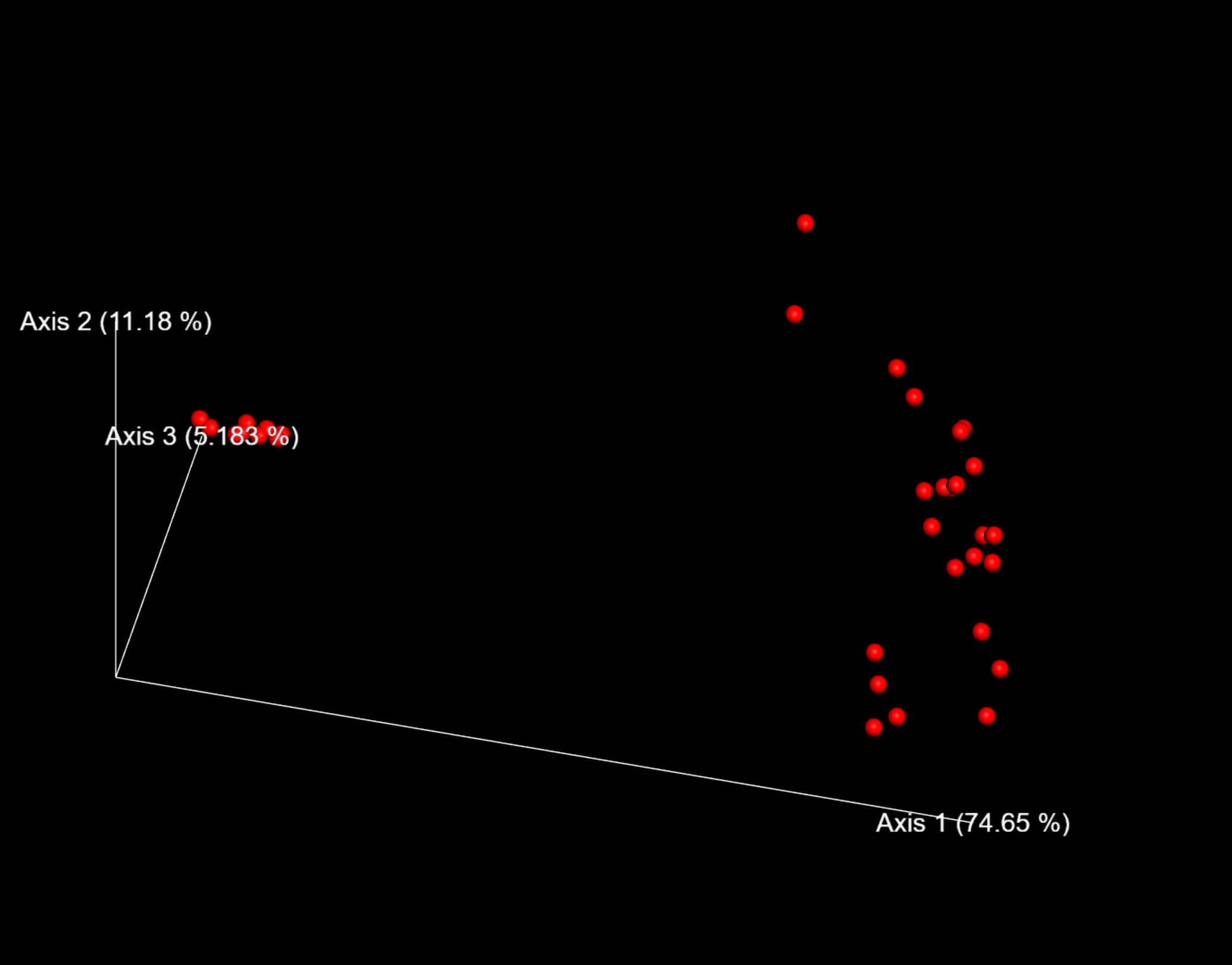
图6. 以weighted_unifrac距离的PCoA结果交互式可视化为例,可用鼠标托动空间查看每个样本的分布位置。
这里,我们将--p-sampling-depth参数设置为1109。这个值是根据L3S341样本中的序列数量来选择的,因为它与接下来几个序列计数较高的样本中的序列数量接近,并且因为它仅比序列较少的一个样本中的序列数量高。这将允许我们保留大部分样品。具有较少序列的一个样本将从core-metrics-phylogenetic分析和任何使用这些结果的任何下游分析中删除。
注意:根据DADA2特征表汇总选择1109的采样深度。如果使用的是Deblur特性表而不是DADA2特性表,则可能需要选择不同的采样深度。应用上一段的逻辑来帮助你选择合理的采样深度。
注意:在许多Illumina测序结果中,你将观察到一些序列计数非常低的例子。你通常希望通过在此阶段采样深度选择更大的值来从分析中剔除它们。
在计算多样性度量之后,我们可以开始在样本元数据的分组信息或属性值背景下探索样本的微生物组成差异。此信息存在于先前下载的示例元数据文件中。
我们将首先测试分类元数据列和alpha多样性数据之间的关系。我们将在这里为Faith系统发育多样性(群体丰富度的度量)和均匀度进行可视化操作。
Alpha多样性组间显著性分析和可视化
qiime diversity alpha-group-significance \
--i-alpha-diversity core-metrics-results/faith_pd_vector.qza \
--m-metadata-file sample-metadata.tsv \
--o-visualization core-metrics-results/faith-pd-group-significance.qzv
qiime diversity alpha-group-significance \
--i-alpha-diversity core-metrics-results/evenness_vector.qza \
--m-metadata-file sample-metadata.tsv \
--o-visualization core-metrics-results/evenness-group-significance.qzv
输出可视化结果:
- core-metrics-results/faith-pd-group-significance.qzv。查看 | 下载
- core-metrics-results/evenness-group-significance.qzv。查看 | 下载
图7. 以faith-pd为例将互探索不同元数据条件下组间差异,可用鼠标选择不同元数据的列名,切换分组方式,探索对应的生物学意义。
问题:哪些分类样本元数据列与微生物群落丰富度的差异密切相关?这些差异在统计学上有显著性吗?
问题:哪些分类样本元数据列与微生物群落均匀性的差异密切相关?这些差异在统计学上有显著性吗?
读者思考时间:实验设计中的那一种分组方法,与微生物群体的丰富度差异相关,这些差异显著吗?
解答:图中可按Catalogy选择分类方法,查看不同分组下箱线图间的分布与差别。图形下面的表格,详细详述组间比较的显著性和假阳性率统计。
结果我们会看到本实验设计的分组方式有Bodysite, Subject, ReportAntibioticUse,只有身体位置各组间差异明显,且下面统计结果也存在很多组间的显著性差异。
在这个数据集中,没有连续的样本元数据列(例如,DaysSinceExperimentStart)与α多样性相关联,所以我们这里不测试这类关联。如果你有兴趣执行这类测试(对于这个数据集或其他数据集),可以使用qiime diversity alpha-correlation命令。
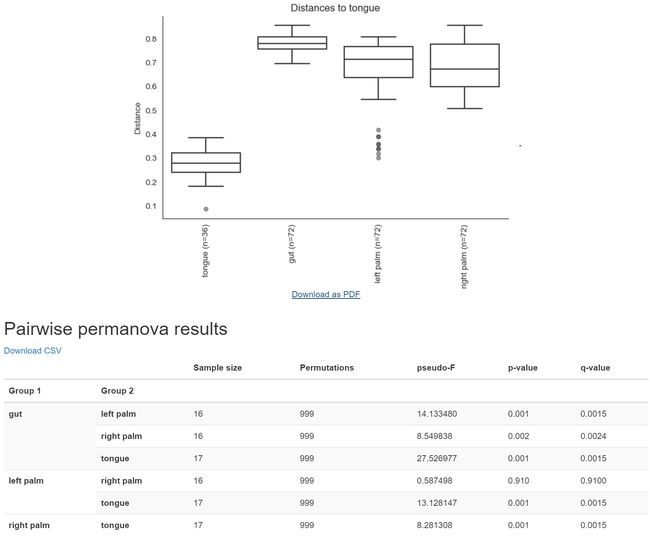
接下来,我们将使用PERMANOVA方法(首先在Anderson 2001年的文章中描述)beta-group-significance分析分类元数据背景下的样本组合。以下命令将测试一组样本之间的距离,是否比来自其他组(例如,舌头、左手掌和右手掌)的样本彼此更相似,例如来自同一身体部位(例如肠)的样本。如果你用这个命令的--p-pairwise参数,它将执行成对检验,结果将允许我们确定哪对特定组(例如,舌头和肠)彼此不同是否显著不同。这个命令运行起来可能很慢,尤其是当使用--p-pairwise参数,因为它是基于置换检验的。因此,我们将在元数据的特定列上运行该命令,而不是在其适用的所有元数据列上运行该命令。这里,我们将使用两个示例元数据列将此应用到未加权的UniFrac距离,如下所示。
qiime diversity beta-group-significance \
--i-distance-matrix core-metrics-results/unweighted_unifrac_distance_matrix.qza \
--m-metadata-file sample-metadata.tsv \
--m-metadata-column BodySite \
--o-visualization core-metrics-results/unweighted-unifrac-body-site-significance.qzv \
--p-pairwise
qiime diversity beta-group-significance \
--i-distance-matrix core-metrics-results/unweighted_unifrac_distance_matrix.qza \
--m-metadata-file sample-metadata.tsv \
--m-metadata-column Subject \
--o-visualization core-metrics-results/unweighted-unifrac-subject-group-significance.qzv \
--p-pairwise
输出可视化结果:
core-metrics-results/unweighted-unifrac-body-site-significance.qzv: 查看 | 下载core-metrics-results/unweighted-unifrac-subject-group-significance.qzv: 查看 | 下载
图8. 不同部分组内和组间差异显著性分析箱线图+统计表
问题
受试者之间的关联和微生物组成的差异在统计学上是否显著?身体部位呢?哪些特定的身体部位对彼此有显著的不同?
同样,我们对于这个数据集所拥有的连续样本元数据中没有一个与样本组成相关,因此这里我们不会测试这些关联。如果你对执行这些测试感兴趣,那么可以使用qiime metadata distance-matrix、qiime diversity mantel和qiime diversity bioenv命令组合使用。
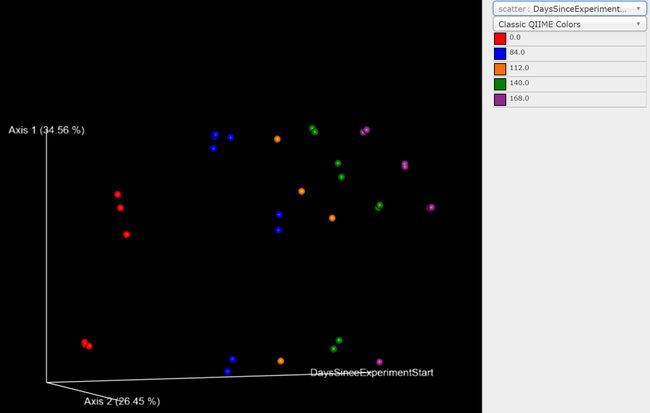
最后,排序是在样本元数据分组间探索微生物群落组成差异的流行方法。我们可以使用Emperor工具在示例元数据背景下探索主坐标(PCoA)绘图。虽然我们的core-metrics-phylogenetic命令已经生成了一些Emperor图,但我们希望传递一个可选的参数--p-custom-axes,这对于探索时间序列数据非常有用。用于core-metrics-phylogeny的PCoA结果也是可用的,这使得很容易与Emperor生成新的可视化。我们将为未加权的UniFrac和Bray-Curtis的PCoA结果生成Emperor图,以便所得到的图将包含主坐标1、主坐标2和实验开始以来的天数(days since the experiment start)的轴。我们将使用最后一个轴来探索这些样本是如何随时间变化的。
qiime emperor plot \
--i-pcoa core-metrics-results/unweighted_unifrac_pcoa_results.qza \
--m-metadata-file sample-metadata.tsv \
--p-custom-axes DaysSinceExperimentStart \
--o-visualization core-metrics-results/unweighted-unifrac-emperor-DaysSinceExperimentStart.qzv
qiime emperor plot \
--i-pcoa core-metrics-results/bray_curtis_pcoa_results.qza \
--m-metadata-file sample-metadata.tsv \
--p-custom-axes DaysSinceExperimentStart \
--o-visualization core-metrics-results/bray-curtis-emperor-DaysSinceExperimentStart.qzv
输出可视化:
- core-metrics-results/unweighted-unifrac-emperor-DaysSinceExperimentStart.qzv: 查看 | 下载
- core-metrics-results/bray-curtis-emperor-DaysSinceExperimentStart.qzv: 查看 | 下载
图9. 探索样本在第1/2主轴和时间上的分布,调整右侧着色方式和颜色方案可方便观察研究的分类或时间序列结果。
问题:Emperor图是否支持我们在这里执行的其他β多样性分析?(提示:对不同实验元数据进行点着色。)
问题:在未加权的UniFrac和Bray-Curtis PCoA图中,你观察到了哪些差异?
Alpha稀释取线
Alpha rarefaction plotting
在本节中,我们将使用qiime diversity alpha-rarefaction可视化器来探索α多样性与采样深度的关系。该可视化器在多个采样深度处计算一个或多个α多样性指数,范围介于1(可选地--p-min-depth控制)和作为--p-max-depth提供值之间。在每个采样深度,将生成10个抽样表,并对表中的所有样本计算alpha多样性指数计算。迭代次数(在每个采样深度计算的稀疏表)可以通过--p-iterations来控制。在每个采样深度,将为每个样本绘制平均多关性值,如果提供样本元数据--m-metadata-file参数,则可以基于元数据对样本进行分组。
qiime diversity alpha-rarefaction \
--i-table table.qza \
--i-phylogeny rooted-tree.qza \
--p-max-depth 4000 \
--m-metadata-file sample-metadata.tsv \
--o-visualization alpha-rarefaction.qzv
输出可视化:
- alpha-rarefaction.qzv: 查看 | 下载
图10. 查看按body site分组下observed otus的稀疏箱线图,注意观察图中变化以及下面对应样本数据的图。
可视化将有两个图。顶部图是α稀疏图,主要用于确定样品的丰度是否已被完全观察或测序。如果图中的线条在沿x轴的某个采样深度处看起来“平坦”(即,接近于零的斜率),这表明收集超过该采样深度的附加序列不太可能导致对附加特征的观察。如果绘图中的线条没有变平,这可能是因为尚未充分观察样本的丰富度(因为收集的序列太少),或者它可能是在数据中仍然存在许多测序错误(这被误认为是新的多样性)的指标。
当通过元数据对样本进行分组时,此可视化中的底部绘图非常重要。它说明了当特征表被细化到每个采样深度时,每个组中剩余的样本数量。如果给定的采样深度d大于样本s的总频率(即,针对样本s获得的序列数),则不可能计算采样深度d下样本s的多样性。在顶部绘图将不可靠,因为它将计算基于相对少的样本。因此,当通过元数据对样本进行分组时,必须查看底部图表,以确定顶部图表中显示的数据是否可靠的。
注意:提供的
--p-max-depth参数的值应该通过查看上面创建的table.qzv文件中呈现的“每个样本的测序量”信息来确定。一般来说,选择一个在中位数附近的值似乎很好用。如果得到的稀疏图中的线看起来没有变平,那么你可能希望增加该值。如果由于大于最大采样深度而丢失了许多样本,则减少该值。
问题1:当通过“BodySite”列信息对样本进行分组并查看“observed_otus”指数的α稀疏图时,哪些身体部位显示出足够的多样性覆盖(即稀疏曲线趋于平缓)?在这些身体部位似乎存在多少序列变异?
问题2:当通过“BodySite”对样本进行分组并查看“observed_otus”指数的α稀疏图时,“右手掌(right palm)”样本的线看起来在40左右变平,但随后跳到大约140。你认为这里发生了什么?(提示:一定要查看顶部和底部的细节。)
译者注问题2答案:左手掌的多样性从突然40跳至140,而对应的样本量从9个下降为3个(由于测序深度不足)。仅有3次生物学重复样本量太少,偶然性太大,导致的结果波动可信度不高。问题1很简单,自己看图吧可以想出答案。
物种组成分析
在这一节中,我们将开始探索样本的物种组成,并将其与样本元数据再次组合。这个过程的第一步是为FeatureData[Sequence]的序列进行物种注释。我们将使用经过Naive Bayes分类器预训练的,并由q2-feature-classifier插件来完成这项工作。这个分类器是在Greengenes 13_8 99% OTU上训练的,其中序列被修剪到仅包括来自16S区域的250个碱基,该16S区域在该分析中采用V4区域的515F/806R引物扩增并测序。我们将把这个分类器应用到序列中,并且可以生成从序列到物种注释结果关联的可视化。
注意:物种分类器根据你特定的样品制备和测序参数进行训练时表现最好,包括用于扩增的引物和测序序列的长度。因此,一般来说,你应该按照使用
q2-feature-classifier的训练特征分类器的说明来训练自己的物种分类器。我们在数据资源页面上提供了一些通用的分类器,包括基于Silva的16S分类器,不过将来我们可能会停止提供这些分类器,而让用户训练他们自己的分类器,这将与他们的序列数据最相关。
下载物种注释数据库制作的分类器:无法下载记得后台回复"qiime2"获得备用下载链接
wget \
-O "gg-13-8-99-515-806-nb-classifier.qza" \
"https://data.qiime2.org/2018.11/common/gg-13-8-99-515-806-nb-classifier.qza"
物种注释和可视化
qiime feature-classifier classify-sklearn \
--i-classifier gg-13-8-99-515-806-nb-classifier.qza \
--i-reads rep-seqs.qza \
--o-classification taxonomy.qza
qiime metadata tabulate \
--m-input-file taxonomy.qza \
--o-visualization taxonomy.qzv
输出结果:
- taxonomy.qza: 物种注释结果
- gg-13-8-99-515-806-nb-classifier.qza: 分类器的训练结果
可视化结果:
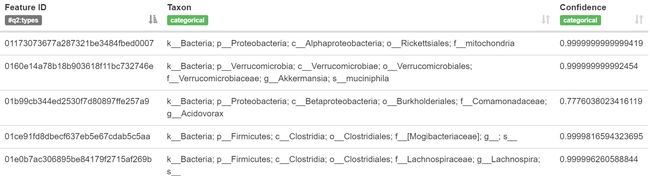
- taxonomy.qzv: 物种注释可视化。查看 | 下载
图11. ID对应的物种分类和置信度
问题:回想一下,
rep-seqs.qzv可视化允许你轻松地对NCBI nt数据库BLAST每个特性的序列。使用此处创建的可视化和taxonomy.qzv可视化,将几个特性物种分配与最佳BLAST命中的分类进行比较,结果有多相似?如果它们不同,它们在什么分类学层次上开始不同(例如,物种、属、科…)?
接下来,我们可以用交互式条形图查看样本的分类组成。使用以下命令绘图成堆叠柱状图,然后打开可视化。
qiime taxa barplot \
--i-table table.qza \
--i-taxonomy taxonomy.qza \
--m-metadata-file sample-metadata.tsv \
--o-visualization taxa-bar-plots.qzv
结果:
taxa-bar-plots.qzv: 查看 | 下载
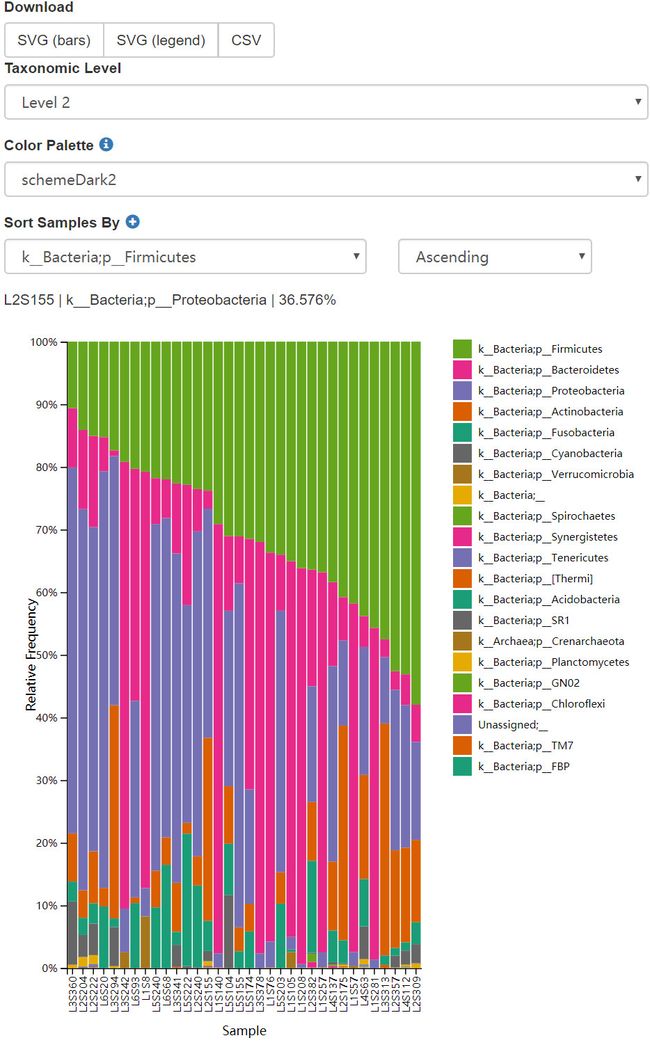
图12. 门水平样本堆叠柱状图、按Firmicutes排序。可切换不同分类级别、选择10余种本色方案; 切换排序各类和方向。
问题:在物种注释第二级可视化样本(在本分析中对应于门级别),然后按
BodySite、Subject、然后按DaysSinceExperimentStart对样本进行排序。在BodySite中不同部位都有哪些优势门类?在DaysSinceExperimentStart0和后面的时间点之间,你是否观察到两个组之间的任何一致的变化规律?
使用ANCOM差异丰度分析
Differential abundance testing with ANCOM
ANCOM可用于识别不同样本组中丰度差异的特征。与任何生物信息学方法一样,在使用ANCOM之前,你应该了解ANCOM的假设和局限性。我们建议在使用这种方法之前先回顾一下ANCOM的论文 https://www.ncbi.nlm.nih.gov/pubmed/26028277。
注意:差异丰度检验在微生物学分析中是一个热门的研究领域。有两个QIIME 2插件可用:
q2-gneiss和q2-composition。本节使用q2-composition,但是如果你想了解更多,还有一个教程在另外的数据集上使用q2-gneiss,在后面有详细介绍。
ANCOM是在q2-composition插件中实现的。ANCOM假设很少(小于约25%)的特征在组之间改变。如果你期望在组之间有更多的特性正在改变,那么就不应该使用ANCOM,因为它更容易出错(I类和II类错误都有可能增加)。因为我们预期身体部位的许多特征都会发生变化,所以在本教程中,我们将过滤完整的特征表后只包含肠道样本。然后,我们将应用ANCOM来确定哪种(如果有的话)序列变体在我们两个受试者的肠道样本中丰度存在差异。
我们将首先创建一个只包含肠道样本的特征表。(要了解关于筛选的更多信息,请参阅数据筛选教程。)
qiime feature-table filter-samples \
--i-table table.qza \
--m-metadata-file sample-metadata.tsv \
--p-where "BodySite='gut'" \
--o-filtered-table gut-table.qza
输出对象:
- gut-table.qza:只包含肠道样本的OTU表
ANCOM基于每个样本的特征频率对FeatureTable[Composition]进行操作,但是不能容忍零。为了构建组成对象,必须提供一个添加伪计数(一种遗失值插补方法)的FeatureTable[Frequency]对象,这将产生FeatureTable[Composition]对象。
qiime composition add-pseudocount \
--i-table gut-table.qza \
--o-composition-table comp-gut-table.qza
输出结果:
- comp-gut-table.qza: 组成型特征表
接下来可用ANCON对两组的特征进行丰度差异的比较了。
qiime composition ancom \
--i-table comp-gut-table.qza \
--m-metadata-file sample-metadata.tsv \
--m-metadata-column Subject \
--o-visualization ancom-Subject.qzv
输出结果:
ancom-Subject.qzv: 按Subject分类比较结果。查看 | 下载
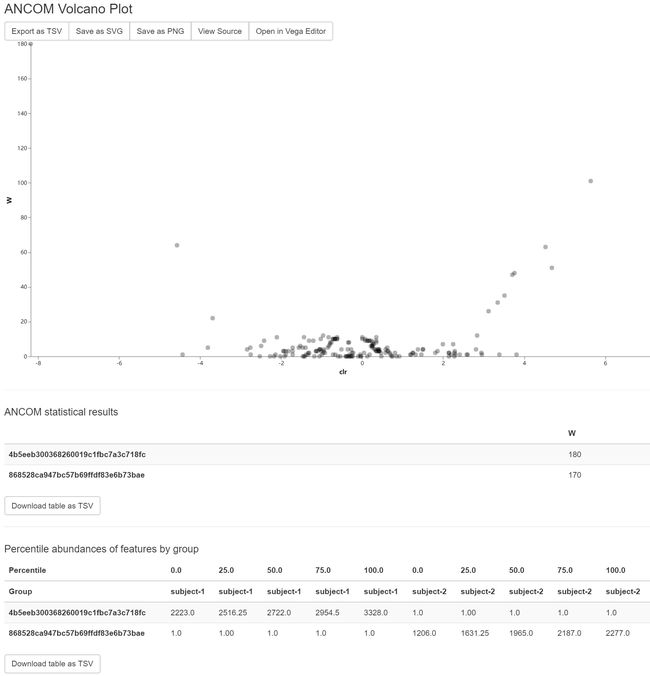
图13. 交互火山图展示组间差异特征。鼠标悬停在特征点上,可显示特征名称和对应的具体坐标。下面有每个显著差异特征的统计结果,以及组内分位数表格。
问题:哪个ASV在分组间差异很大?每个序列变体在哪个分组中更丰富?这些序列变体的分类是什么?(要回答最后一个问题,你需要参考本教程中生成的另一个可视化。)
我们也经常对在特定的分类学层次上执行差异丰度检验。为此,我们可以在感兴趣的分类级别上折叠FeatureTable[Frequency]中的特性,然后重新运行上述步骤。在本教程中,我们将特征表折叠到属级别(即Greengenes分类法的第6级)。
qiime taxa collapse \
--i-table gut-table.qza \
--i-taxonomy taxonomy.qza \
--p-level 6 \
--o-collapsed-table gut-table-l6.qza
qiime composition add-pseudocount \
--i-table gut-table-l6.qza \
--o-composition-table comp-gut-table-l6.qza
qiime composition ancom \
--i-table comp-gut-table-l6.qza \
--m-metadata-file sample-metadata.tsv \
--m-metadata-column Subject \
--o-visualization l6-ancom-Subject.qzv
输出对象:
- gut-table-l6.qza: 按属水平折叠的特征表
- comp-gut-table-l6.qza: 属水平筛选肠样本的相对丰度组成表
输出可视化结果:
- l6-ancom-Subject.qzv: 查看 | 下载

图14. 交互火山图展示组间差异属。鼠标悬停在特征点上,可显示属名称和对应的具体坐标。下面表格为每个显著差异属的统计结果,以及组内分位数表格。
问题:哪个属在不同组间有丰富的差异?哪一组每个属比较丰富?
译者简介
刘永鑫,博士。2008年毕业于东北农大微生物学专业。2014年中科院遗传发育所获生物信息学博士学位,2016年博士后出站留所工作,任宏基因组学实验室工程师,目前主要研究方向为宏基因组学、数据分析与可重复计算和植物微生物组、QIIME 2项目参与人。发于论文12篇,SCI收录9篇。2017年7月创办“宏基因组”公众号,目前分享宏基因组、扩增子原创文章300+篇,代表博文有《扩增子图表解读、分析流程和统计绘图三部曲》,关注人数3万+,累计阅读400万+。
Reference
- https://qiime2.org/
- Bolyen E, Rideout JR, Dillon MR, Bokulich NA, Abnet C, Al-Ghalith GA, Alexander H, Alm EJ, Arumugam M, Asnicar F, Bai Y, Bisanz JE, Bittinger K, Brejnrod A, Brislawn CJ, Brown CT, Callahan BJ, Caraballo-Rodríguez AM, Chase J, Cope E, Da Silva R, Dorrestein PC, Douglas GM, Durall DM, Duvallet C, Edwardson CF, Ernst M, Estaki M, Fouquier J, Gauglitz JM, Gibson DL, Gonzalez A, Gorlick K, Guo J, Hillmann B, Holmes S, Holste H, Huttenhower C, Huttley G, Janssen S, Jarmusch AK, Jiang L, Kaehler B, Kang KB, Keefe CR, Keim P, Kelley ST, Knights D, Koester I, Kosciolek T, Kreps J, Langille MG, Lee J, Ley R, Liu Y, Loftfield E, Lozupone C, Maher M, Marotz C, Martin BD, McDonald D, McIver LJ, Melnik AV, Metcalf JL, Morgan SC, Morton J, Naimey AT, Navas-Molina JA, Nothias LF, Orchanian SB, Pearson T, Peoples SL, Petras D, Preuss ML, Pruesse E, Rasmussen LB, Rivers A, Robeson, II MS, Rosenthal P, Segata N, Shaffer M, Shiffer A, Sinha R, Song SJ, Spear JR, Swafford AD, Thompson LR, Torres PJ, Trinh P, Tripathi A, Turnbaugh PJ, Ul-Hasan S, van der Hooft JJ, Vargas F, Vázquez-Baeza Y, Vogtmann E, von Hippel M, Walters W, Wan Y, Wang M, Warren J, Weber KC, Williamson CH, Willis AD, Xu ZZ, Zaneveld JR, Zhang Y, Zhu Q, Knight R, Caporaso JG. 2018. QIIME 2: Reproducible, interactive, scalable, and extensible microbiome data science. PeerJ Preprints 6:e27295v2 https://doi.org/10.7287/peerj.preprints.27295v2
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外2600+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA