Author: Marijori A. Matzke and Rebecca A. Mosher
Journal: Nature Review Genetics
doi: 10.1038/nrg3683
link: RNA-directed DNA methylation: an epigenetic pathway of increasing complexity
Abstract
Abstract | RNA-directed DNA methylation (RdDM) is the major small RNA-mediated epigenetic pathway in plants. RdDM requires a specialized transcriptional machinery that comprises two plant-specific RNA polymerases — Pol IV and Pol V — and a growing number of accessory proteins, the functions of which in the RdDM mechanism are only partially understood. Recent work has revealed variations in the canonical RdDM pathway and identified factors that recruit Pol IV and Pol V to specific target sequences. RdDM, which transcriptionally represses a subset of transposons and genes, is implicated in pathogen defence, stress responses and reproduction, as well as in interallelic and intercellular communication.
RNA指导的DNA甲基化是植物中小RNA参与的表观遗传调控的主要方式。RdDM的实现需要由两个植物特有的两个RNA聚合酶Pol IV and Pol V以及大量的辅助蛋白构成的转录复合体的参与,这些辅助蛋白的功能在RdDM途径中的作用还没有完全搞明白。最近的工作揭示了经典的RdDM途径的变化,同时鉴定出可以将Pol IV and Pol V募集到目标基因上的一些因子。RdDM可以在转录水平抑制转座子,基因(表达),参与到了植物抗病,胁迫应答,再生以及与等位基因间(interallelic)和细胞间的通信。
小RNA调控基因表达
Overview
RNA interference (RNAi) is an umbrella term that describes gene silencing phenomena triggered by small RNAs.
- (post-transcription gene silencing, PTGS)在细胞质中,siRNA通过在转录后加工过程中对complementary mRNAs的降解和抑制来沉默基因表达.
- (transcriptional gene silencing, TGS)在细胞核内,小RNA通过对基因组同源区域的表观遗传修饰例如DNA胞嘧啶甲基化和组蛋白甲基化来导致基因沉默。
- 分为两组:小干扰
RNA small interfering RNAs,siRNA 和PIWI-interacting RNAs,piRNAs,其中siRNAs广泛存在于动物植物真菌中,但是piRNAs只存在于后生动物中,在植物和真菌中不存在,在植物汇中由小RNA介导的DNA甲基化是小RNA参与到表观遗传主要的方式。
RdDM
In plants, the major siRNA-mediated epigenetic pathway is RNA-directed DNA methylation (RdDM), which is the subject of this Review. Initially detected in plants that were infected with RNA pathogens , RdDM was later shown to require siRNAs and core RNAi proteins.
RdDM通路是植物特有的,因为整个通路围绕着两个植物特有的与RNA聚合酶II相关的酶,分别是聚合酶IV和聚合酶V。
RNA pol IV 和Pol V详细介绍
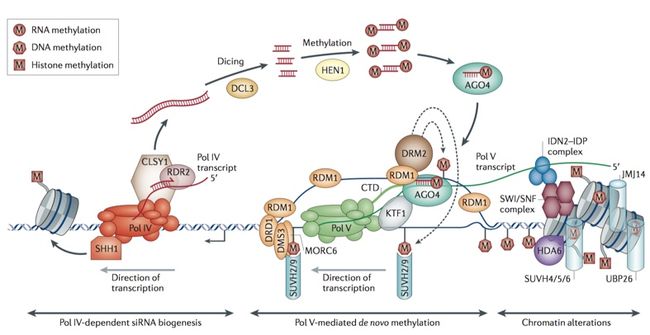
- DNA甲基化是一种重要的表观遗传学修饰。在植物中,DNA甲基化发生于CG,CHG和CHH序列。DNA甲基化在这些序列的建立都依赖于一条RNA介导的DNA甲基化(RNA-directed DNA methylation,RdDM)途径。RNA聚合酶Pol IV和Pol V在RdDM途径中分别负责产生小干扰RNA(small interfering RNAs,siRNAs)和长非编码RNA(Long Intergenic Noncoding RNAs, IGN RNAs)。siRNA和AGO4结合并与IGN RNA互补配对,招募甲基转移酶DRM2(Domain Rearranged Methyltransferase 2)至靶标位点建立DNA甲基化。
- Box1 RNA polymerases IV and V
RNA聚合酶有三大类: RNA聚合酶 I → 核糖体大亚基; RNA聚合酶 II → mRNA前体; RNA聚合酶III → tRNA和5S rRNA, Pol IV和Pol V与 Pol II 同源,是植物特有的,参与到RdDM。首先PolII PolIV和PolV均有12个亚基,大部分都是共有的,除了PolII的nuclear RNA polymerase B,NRPB; Pol IV的NRPD以及Pol V的NRPE。其中比较大的亚基是NRPD1和NRPE1,分别于NRPD2及NRPE2构成了催化核心,与polII 的NRPB1不同,在催化中心和碳末端CTDs存在保守氨基酸序列的代换和缺失,这可能就是他们参与到RdDM机制的原因,在NRPB1的CTDs区域有一段7肽重复,但是在NRPD1和NRPE1中这些重复是不存在的,在这些7肽重复中包含了一个基序(motif),这个基序通常出现在被称作有缺陷的叶绿体和叶片的蛋白中DeCL,事实上,DeCL蛋白通常参与到rRNA的生理过程,但是NRPD1和NRPE1中的CTDs的功能未知。NRPE1中延长的CTD同时包含了Trp-Gly或者Gly-Trp的重复序列,这些重复序列是AGO蛋白的结合区域,可以和AGO4结合,而这个结构决定了Pol V参与到siRNA介导的从头甲基化。
在Pol II中NRPB1和nRPB2亚基会结合NRPB5和NRPB9B会形成一个钳型结构,在转录的时候可以抓住DNA,Pol V的NRPE5和NRPE9B的功能表明这或许是适应新的转录模板的特殊结构特征或者染色质修饰。在体外实验中,Pol IV和PolV可以根据一个由RNA-DNA共同构成的模板来转录一个有RNA引物引发的转录本;此外,Pol IV还可以转录出一段RNA-RNA模板。尽管体外Pol IV和pol V的模板已知,但是一些有一些与他们传统功能不同的,例如作为非常规的内切酶或者外切酶,或者是一个染色质外的模板应该被考虑。与Pol II相似,Pol IV和Pol V需要一些因子将他们从细胞质带入细胞核中,酵母同源IWR1已经被证明可以将Pol II运输到细胞核,在RdDM缺陷型突变体中,Pol IV 和Pol V的入核的方式与Pol II的方式相似。
过程简述
首先由Pol IV 转录成长的双链RNA(long dsRNAs),然后在DICER_LIKE3, (DCL3)作用下加工成siRNAs,然后输出到细胞质中;接下来,这些siRNAs的一条链装载到AGO4,然后再运回到细胞核,根据碱基互补配对原则这些siRNA会靶定Pol V转录的新生转录骨架上(nascent scaffold transcripts..真的布吉岛怎么翻译,但可以确定是一段非编码RNA?)最终,这个复合体会招募DNA甲基转移酶介导所有包含CG,CHG和CHH(H代表A,C或者T)序列位点的甲基化。最终结果会导致由Pol V转录的基因座上的转录本的沉默,特别是在转座子位置或者DNA重复序列处。这种机制与裂殖酵母中的TGS截然相反.
裂殖酵母的TGS中的RNA Pol II 同时转录出siRNAs的前体和吸附siRNAs的支架RNA(scaffold RNA),然后再招募沉默感受复合体(silencing effector complex),与诱导DNA甲基化方面相比,这些沉默感受符合体通过对组蛋白3第9位上的赖氨酸进行甲基化(H3K9me)实现转录沉默和染色体异质化。通常发生在
pericentromeric repeats区域,或者一些发育调节基因developmentally regulated genes及逆转座子retrotransposons部位。
Canonical RdDM pathway mechanisms
Pol IV dependent siRNA biogensis
Pol VI缺陷突变体证明超过90%的24nt-siRNAs来自Pol VI,而这些24nt-siRNAs会引起甲基化事件。
- Pol VI结合到TE和重复序列上的机制尚不完全清楚; 首先
SAWADEE HOMEODOMAIN HOMOLOGUE 1 (SHH1)会招募Pol VI, SHH会通过串联的Tudor-like fold结合到甲基化的H3K9和没有甲基化的H3K4。 - 尽管没有在体内发现Pol IV的转录本,但是可以推断出Pol IV可以在他的target基因座上转录出单链RNAs。该ssRNA会被
RNA-dependent RNA polymerase, RDR2复制形成dsRNAs。 -
The chromatin remodeller CLASSY 1 (CLSY1)也参与到该过程。 - DCL3(dicer)将合成的dsRNAs加工为24nt-siRNAs,然后经过3‘-OH的甲基化后装载在
AGO4,该过程是由HEN1完成的。
Tips : AGO4还存在其他分支,比如部分冗余的AGO6,以及在再生组织中特异表达的AGO9
Pol V-mediated de novo methylation
- 三磷酸化的或者5'端被封住缺少poly(A)尾的Pol V的转录本被认为可以提供一个支架RNA和siRNAs互作,并且招募其他沉默因子。与Pol IV一样,具体的机制尚不清楚,与之结合的DNA共有序列也没有被发现;但很多ChIP-seq的结果显示大部分Pol V被定位在胞嘧啶甲基化的转座子和重复序列上,同时发现有24nt-siRNAs的存在。所以说明Pol V介导的RdDM应该发生在这些区域。
- Pol V 介导的RdDM通常发生在常染色质区域,特别是小的 “年轻” 的基因间区转座子,或者存在于启动子区域,外显子,内含子区域的重复序列。RdDM靶标的位置与前面预测的PolIV PolV和PolII推测的一致,位于常染色质主要的基因转录区域。此外,Pol V在基因启动子区域的富集也说明了它继承了Pol II在基因上有的偏好性。
- RdDM似乎在某种程度上不会发生在富含大转座子的近着丝粒端的异染色质区域。相反,该区域的修饰(H3K9me and DNA甲基化),更倾向于一种siRNA独立的方式并且依赖于染色质重塑蛋白(chromatin remodeller DECREASED DNA METHYLATION1, DDM1)、DNA5端胞嘧啶甲基转移酶(DNMT1),class enzyme 甲基转移酶(MET1,也叫DMT1),还有植物特异甲基转移酶(CMT2)和(CMT3)。
- SUVH2、SUVH9和SUVR2是SU(var)组蛋白甲基转移酶家族的成员,它们有助于将Pol V招募到一些靶序列中。suvh2和suvh9不能催化组蛋白甲基化,但它们可以通过其SRA(SET and RING-associated)结构域与甲基化的DNA结合,从而有助于polV与染色质的相互作用平台,染色质包含一些预先存在的甲基化。PolV的转录与染色体的结合是在DDR复合体协助下完成的。DDR复合体的成员:CLSY1-related putative chromatin remodeller DEFECTIVE IN RNA_DIRCTED DNA METHYLATION1(DRD1);DEFECTIVE IN MERISTEM SILENCING3(DMS3),DMS3是一个维持染色体构像的铰链蛋白;还有一个在RdDM通路中起到多种作用的小的植物特异蛋白:RNA-DIRCTED DNA METHYLATION1(RDM1)。
- Pol V 与NRPE1互作来招募AGO4蛋白,他们都含有AGO hook motif,这个基序就是与AGO。PolV的motif位于C-端特殊的大亚基上,KTF1则是一个疑似的包含AGO hook motif的延伸因子。在Pol V介导的转录中,结合在AGO4上的siRNA会和PolV新转录的转录本根据碱基互补配对结合,然后招募结构重排甲基转移酶2(DRM2)进而对基因组同源区域的DNA进行从头甲基化,RDM1在招募DRM2的过程中起到关键作用,因为它是唯一一个被证明和AGO4和DRM2都能互作的蛋白,同时他还可以结合单链DNA。RDM1在RdDM机制中可能还会扮演更多的角色。
- INVOLVED IN DE NOVO2(IDN2)
IDN2是一个双链RNA结合蛋白,和SUPPRESSOR OF GENE SILENCING 3,SGS3有关,同时IDN2与PTGS也有关。 IDN2可以和两个冗余的旁系同源产物(redundant paralogues)IDP1和IDP2形成复合体。IDN2-IDP复合体可以稳定siRNAs和PolV支架RNAs结合的稳定性,同时还可以通过和SWI/SNF染色质重塑复合体的互作来改变核小体定位,从而加速RdDM机制的完成。
Interplay with chromatin features
组蛋白修饰
大约70%的RdDM的target几乎都被H3K9me修饰过,这样可以形成一个DNA甲基化的反馈循环来加强基因沉默,比如SUVH4(kryptonite)SUVH5和SUVH6催化H3K9me,与CMT3催化的非CG-甲基化紧密相关,然而CMT3在染色体上结合的位点是H3K9me,因此建立一个自我加强的loop可以长期保持表观修饰。
一些RdDM的target需要一些可以去除激活标记的组蛋白修饰酶来维持DNA甲基化和促进H3K9的甲基化,这些激活标记包括乙酰化,H3K4me和H2B泛素化等。HDA6与MET1结合来维持CG甲基化并且通过乙酰化组蛋白来维持H3K9me。Jumonji C-type histone demethylase JUMONJI14(JMJ14)脱甲基酶以及两个相关的组蛋白脱甲基酶LYSINE-SPECIFIC HISTONE DEMETHYLASE 1(LDL1)和LDL2一起维持DRM2指导的DNA甲基化,可能是通过去除靶标位置的H3K4me。对于非CG甲基化及靶标位置H3K9me的维持需要H2B的去泛素化。
高阶染色质结构
首先两个MORC-型ATPases在RdDM通路中起到重要作用,在拟南芥中,MORC6作为ATPase将DMS3变为SMCHD1,这个蛋白是个cohesin-like蛋白,在大鼠中会导致X染色体的去激活。MORC1和MORC6缺失植物突变体表现出轻微的DNA甲基化和组蛋白修饰抑制尽管在靶标位点失去了对转录组的沉默,这表明他们的作用应该是导致染色质凝结,有研究支持了这个说法,一项研究表明这些突变体在着丝粒周围的异染色质表现出去凝结化。