最强降维模型t-SNE vs 最常用降维模型PCA(下)
异常点检测
前文讲到tsne在高维度数据有着绝对的优势,能够碾压其余降维模型,在低维度的数据也毫不逊色任何一种。但是缺点也很明显,性能的代价往往是时间,它的时间复杂度过高。
tsne降维完分块的特点很明显,但也有一些飘来飘去的点,这些点叫做异常点,异常点的剔除在日常生活中非常常见。比如机器零件的异常,食品,饮料等部分指标异常等。
t-SNE和PCA降维后都有异常点,我们试着来分析分析,这些异常点如何检测以及通过这些点来判断降维的效果。
常见的检测异常点有4种异常点检测方法,One-Class SVM(一分类向量机,非高斯分布)、EllipticEnvelope(基于高斯概率密度的异常点检测)、Isolation Forest(基于集成学习方法异常点检测)、LocalOutlierFactor(基于密度的局部异常因子),本文重点介绍基于高斯概率密度的异常点检测。因为博主曾经写过关于高斯概率密度分布的博客,有一定的联系。
实战
一样的原理不作分析,直接进入实战。首先就是导入相关包。
from sklearn.manifold import TSNE
from sklearn.datasets import load_iris,load_digits
from sklearn.decomposition import PCA
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
用的还是手写数字数据集和鸢尾花数据集。
在博主上一篇博客详细介绍了,如何tsne与pca方法进行降维,一些设置的参数如n_components=2表示降到2维,这些就不再赘述。但值得注意的是,博主这里将数据集放入模型后得到的2维点又和标签合并起来了,最终将数组变成dataframe形式。这是为何呢?这样做是方便下面异常点检测,运用dataframe格式比数组来得方便些。注意一下这里数组的合并,np.hstack是专门在行上的合并,例如[1,2]与[3]合并就变成[1,2,3]。
digits = load_digits()
X_digits_tsne = TSNE(n_components=2, random_state=33).fit_transform(digits.data)
X_digits_pca = PCA(n_components=2).fit_transform(digits.data)
X_digits_tsne = np.hstack((X_digits_tsne, digits.target.reshape(-1, 1)))
X_digits_pca = np.hstack((X_digits_pca, digits.target.reshape(-1, 1)))
X_digits_tsne = pd.DataFrame(X_digits_tsne)
X_digits_pca = pd.DataFrame(X_digits_pca)
上面是对手写数字数据集的预处理,下面同样的也是预处理,对于鸢尾花数据集的预处理,其功能和上面一样。最后处理完的数据格式也和上面一样。

输出一些数据看看格式,第一列是x,第二列是y,第三列是标签,现在数据是dataframe格式。
from sklearn.covariance import EllipticEnvelope
ad_model = EllipticEnvelope(contamination=0.19)
这里导入检测异常点的高斯概率密度模型。先将EllipticEnvelope实例化,其中最重要的一个参数就是contamination,它的默认值是0.1,其实一般情况下0.1就是效果很好的了。但是为了显示效果,博主特地放大了一点,夸张表示能够更好看出区别。
这个参数的意思有点类似dbscan模型的半径参数,读者可以理解为以这样一个比例的圆,不断飘逸搜索附近的点看看是否符合这个比例半径的距离。如果是就是同类,不是就是异常点。是不是有点聚类的意思呢?(因为博主水平有限,不知这里这样解释是否合理,如果有大佬纠错,我将感激不尽!)
plt.figure(figsize=(16, 7), dpi=100)
plt.subplot(1, 2, 1)
for i in range(10):
a = X_digits_tsne.iloc[:,:2][X_digits_tsne.iloc[:,2]==float(i)]
a = a.values
ad_model.fit(a)
y_predict = ad_model.predict(a)
plt.scatter(a[:,0], a[:,1], marker='x', label='original_data')
plt.scatter(a[:,0][y_predict==-1], a[:,1][y_predict==-1],
marker='o', facecolor='none',
edgecolors='red', s=150, label='anomaly_data')
这里就是画图了。首先用a提取出dataframe的坐标。此时a就是所有坐标的数组。用刚刚创建的高斯概率密度模型的实例化进行训练a。得到预测结果y_predict。
首先我们画出原始点original_data,以x形状表示。然后我们画异常点,如果是异常点,那么y_predict会显示为-1.正常点是1.那么就可以用dataframe特有的取数据方式来取出异常点。也就是下一行的scatter的作用,就是画异常点,圈起来,圈圈不填充就是参数facecolor=‘none’,圈圈的边为红色。大小为150.
同样的我们对pca得到的手写数字数据集进行判断异常点。
代码十分相似,故不再赘述。
plt.subplot(1, 2, 2)
for i in range(10):
a = X_digits_pca.iloc[:,:2][X_digits_pca.iloc[:,2]==float(i)]
a = a.values
ad_model.fit(a)
y_predict = ad_model.predict(a)
plt.scatter(a[:,0], a[:,1], marker='x', label='original_data')
plt.scatter(a[:,0][y_predict==-1], a[:,1][y_predict==-1],
marker='o', facecolor='none',
edgecolors='red', s=150, label='anomaly_data')
plt.savefig('D:/桌面/5.png')
最后我们得到以下图片。可以看到左图很多明显属于分块的点也被圈起来了,这是因为contamination过大,前文讲到博主这里是做了夸张的处理,为了更好的观察效果。但是每一个块的中心还能很容易的分辨出来。但是pca这边呢。一盘散沙一样的。效果非常差。几乎每个地方都有红圈。
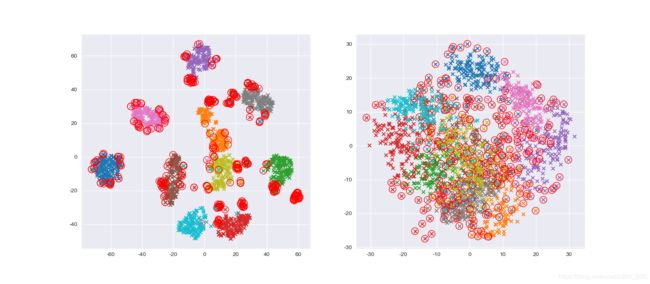
因此我们得到结论,在高纬度的数据集上,tsne降维模型有着绝对的优势能够碾压其余降维模型。细心的读者已经发现,博主载入了鸢尾花数据但是没有画出来,博主偷个懒。留给读者当个小实战任务吧!看看读者们画出来的效果是不是tsne完爆了pca呢?