R: 使用CHAMP包进行甲基化数据的差异分析(QC, CNV, DMP, DMR等)
CHAMP: 甲基化数据的差异分析
本文介绍如何使用CHAMP包对自己的甲基化数据进行分析(QC, CNV, DMP, DMR等)
包的安装
setwd('E:/wu/R')
#~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~#
source("http://bioconductor.org/biocLite.R")
##if faild, try the following one
install.packages("BiocInstaller",repos="http://bioconductor.org/packages/3.7/bioc")
library(BiocInstaller)
##安装一些依赖包
biocLite(c('kernlab',"minfi","ChAMPdata","Illumina450ProbeVariants.db","sva","IlluminaHumanMethylation450kmanifest","limma","RPMM","DNAcopy","preprocessCore","impute","marray","wateRmelon","goseq","plyr","GenomicRanges","RefFreeEWAS","qvalue","isva","doParallel","bumphunter","quadprog","shiny","shinythemes","plotly","RColorBrewer","DMRcate","dendextend","IlluminaHumanMethylationEPICmanifest","FEM","matrixStats","missMethyl","combinat"))
##安装ChAMP
biocLite("ChAMP")
#~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~~
library('ChAMP')
安装好依赖的包之后,就可以进行分析了。在分析过程中,如果有提示说缺少某某包,那就另外再使用biocLite安装一下即可。
数据导入
#载入数据
testDir = "E:/wu/R/OE2018Q1084N_CNV/raw"
myLoad <- champ.load(testDir,arraytype = 'EPIC')
其中,raw文件夹内的文件为自己的850K芯片原始文件
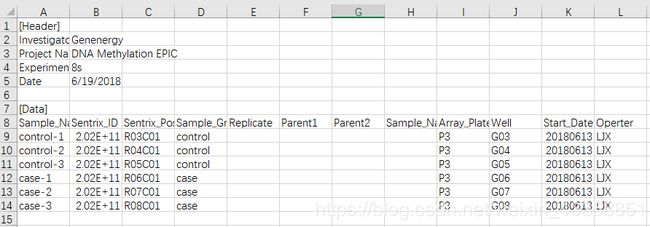
Sample_Sheet.csv的内容如下:
质控分析
champ.QC()
该命令会直接在R的工作路径下生成一个叫CHAMP_QCimages 的文件夹,里面为质控绘图结果。
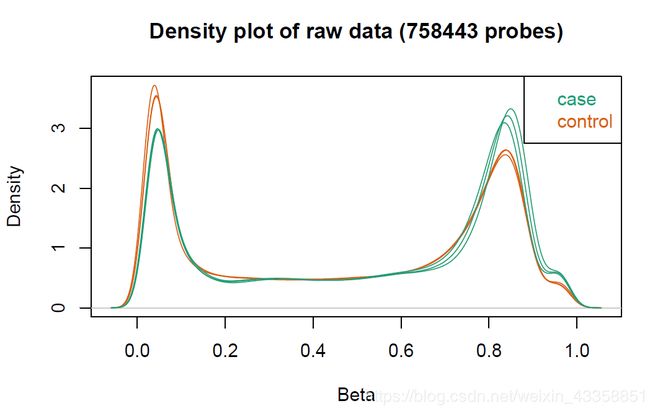
三张图如下所示:
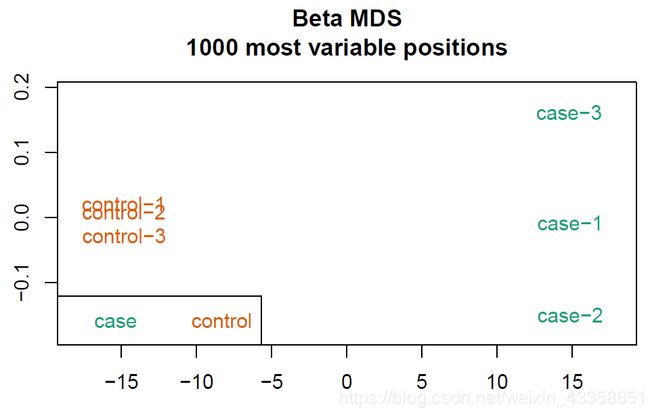
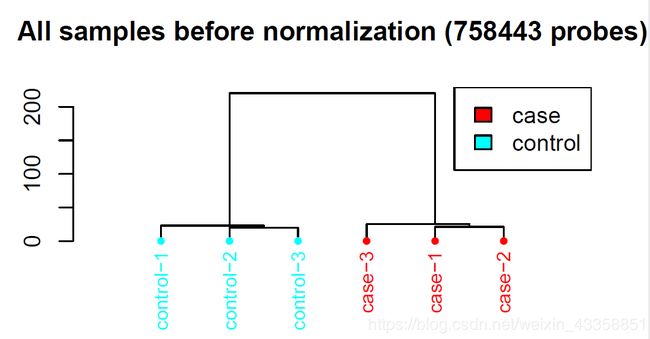
标准化
myNorm <- champ.norm(arraytype="EPIC")
##myNorm <- champ.norm(beta=myLoad$beta,arraytype="EPIC",cores=5)
##cores默认3
#保存标准化数
write.csv(myNorm,file="./Normalization Data.csv",quote=F,row.names = T)
说明:该过程会自动生成一个CHAMP_Normalization 的文件夹,但是有时候往往是空的,可以使用上述代码自己保存标准化数据。
CNV 分析
##CNV分析
myCNA <- champ.CNA(intensity=myLoad$beta,pheno=myLoad$pd$Sample_Group,controlGroup='control',arraytype="EPIC")
#保存差异CNV结果
write.csv(myCNA$groupResult,file="./CNV_analysis_result.csv",quote=F,row.names = F)
该过程会自动生成一个CHAMP_CNA 文件夹,里面是实验组每个样本的CNV绘图结果,如下图所示。
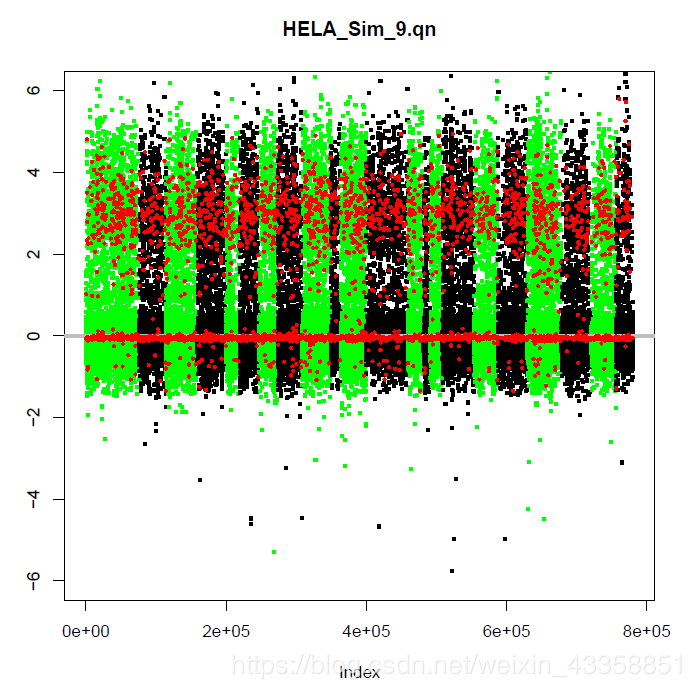
另外还可以做实验组的整体变化情况
myCNA <- champ.CNA(intensity=myLoad$beta,pheno=myLoad$pd$Sample_Group,sampleCNA = FALSE,controlGroup='control',arraytype="EPIC")
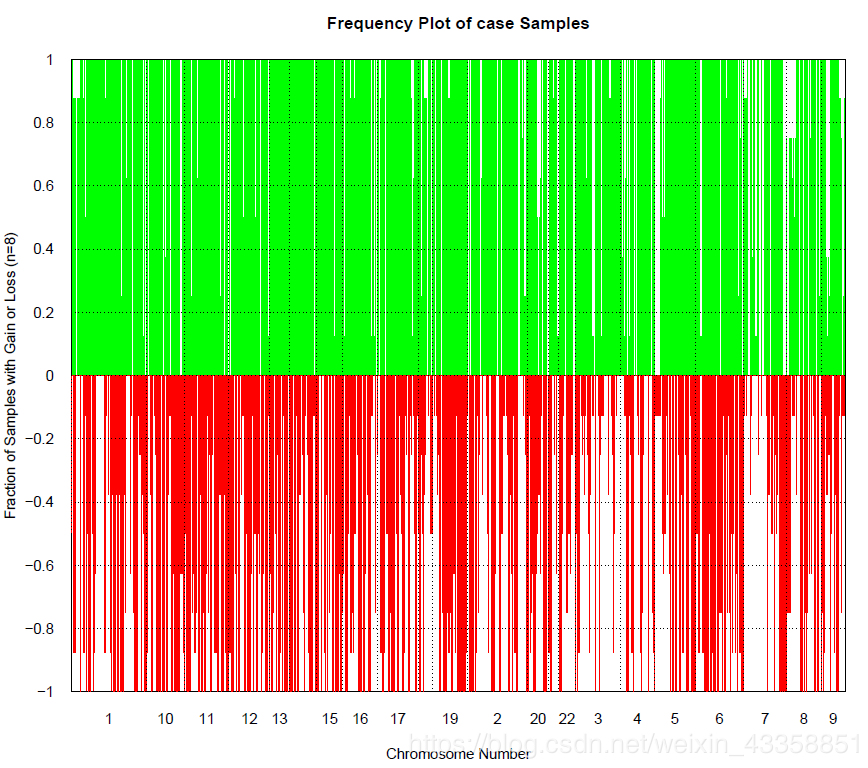
这个图还是比较丑的,没有conumee包绘制的图漂亮。后续会介绍conumee这个包的使用。
DMP分析
##DMP分析
myDMP <- champ.DMP(arraytype="EPIC")
##交互式结果展示
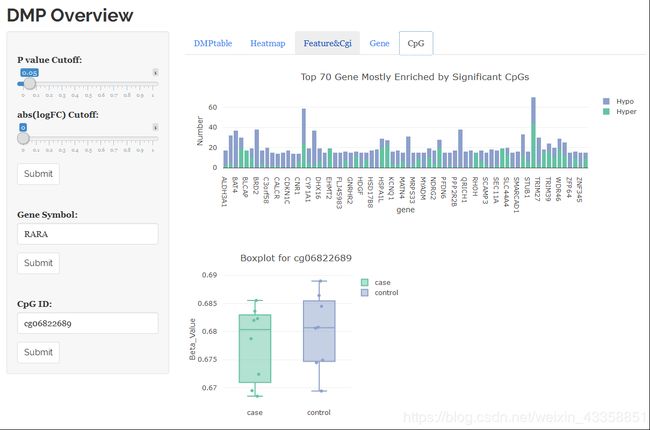
DMP.GUI()
弹出一个交互式界面,可以进行查询等操作。以下是一些界面的示意图。
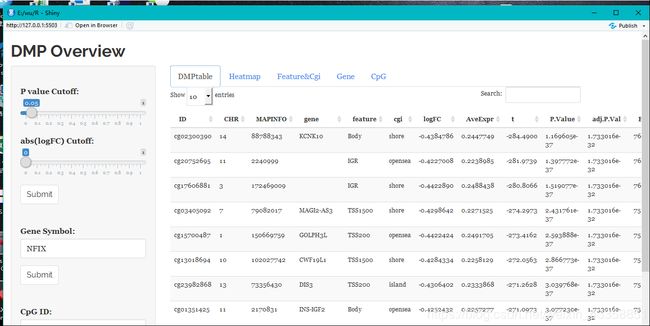
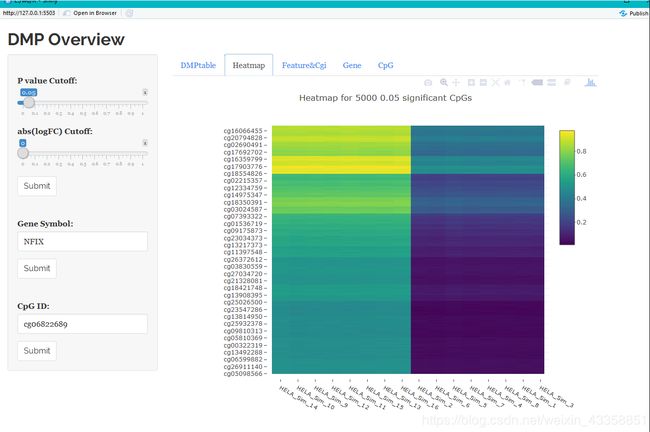
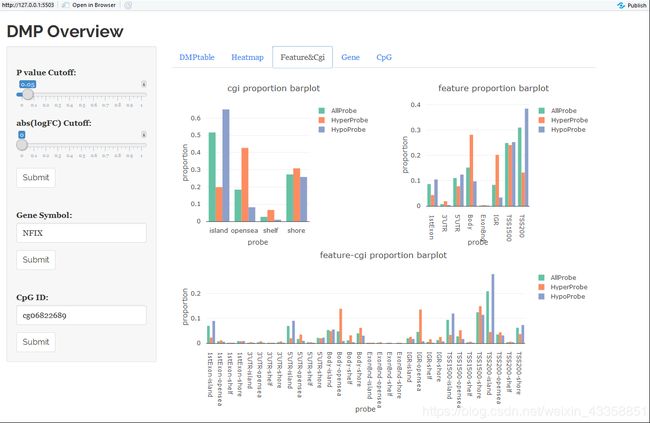
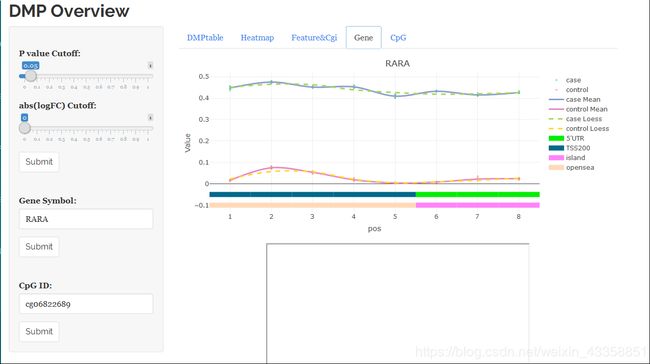
DMR分析
myDMR <- champ.DMR(arraytype="EPIC")
DMR.GUI()
也是非常 简单的,结果与DMP分析结果类似,也是交互式界面,这里就不演示结果了。
其他的具体细节可以参考CHAMP官方网站:
http://www.bioconductor.org/packages/release/bioc/html/ChAMP.html