Cytoscape制作带bar图和pie图节点的网络图
采用cytoscape制作带bar图和pie图节点的网络图
作者:中科院微生物所 陈亮博士
本教程旨在告诉大家如何使用cytoscape根据Node信息表格制作带有barplot信息节点的网络图。以安装文件夹下的样例数据为例。
软件安装(装过请跳过)
Java安装
Cytoscape的使用需要依赖Java环境,根据不同的版本选择相应的Java程序,Cytoscape 3.4.0使用Java8,不再支持Java6和Java7。不同操作系统选择相应的Java版本下载安装,详见 https://www.java.com/zh_CN/ 。
Cytoscape安装
Cytoscape 同样有适用于不同操作平台的版本,安装方法不尽相同。所有的版本都可以从 http://cytoscape.org/ 网站下载。Windows系统下双击exe应用程序开始安装;Linux和Mac OS X系统运行sh文件安装。
打开Cytoscape
本文以Cytoscape_v3.4.0为例,不同版本操作类似,略有不同请自行按思路进行修改进行。
- 打开cytoscape并载入一个网络。
从主菜单选择 File → Import → Network → File…, 然后选择安装文件夹下的sampleData文件夹里的galFiltered.sif数据并载入。
- 采用Network Analyzer产生一些节点或边的统计量。
从主菜单选择 Tools → Network Analyzer → Network Analysis → Analyze Network…, 最后点击 OK。计算结果将以常规表格的形式展示在结果部分。此部分也可以根据自己需要导入Node信息表格。
- 制作一个新的Style。
选左侧择控制面板(Control Panel)部分的Style选项卡。点击 Options 按钮(左上方一个下三角形按扭),并选择Create New Style,然后填写一个名字作为你自己的新的style。如style1
- 显示
Image/Chart 1选项
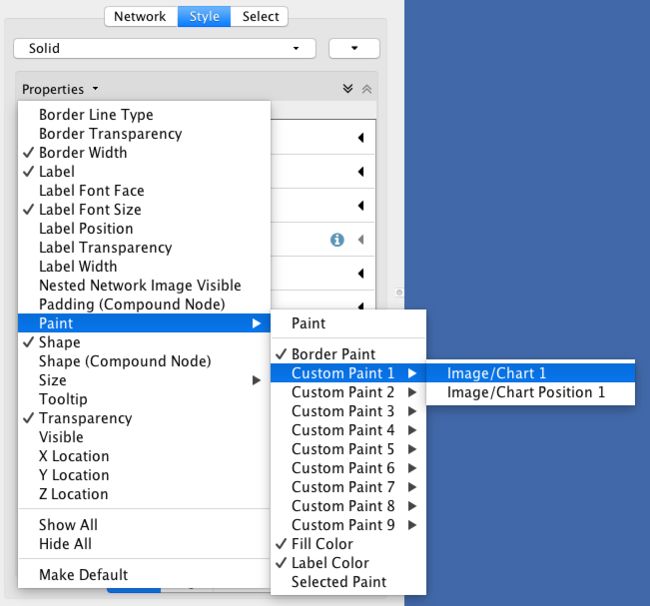
查看Properties面板下是否存在Image/Chart 1 选项,如果没有,可以通过Properties下拉选项来添加,步骤为Properties → Paint → Custom Paint 1 → Image/Chart 1。
- 单击 Image/Chart 1 选项处默认格子以打开Graphics 会话窗口。
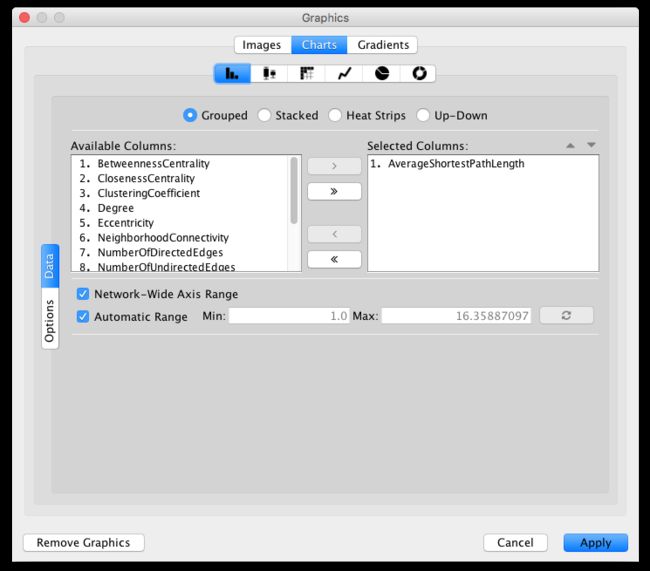
- 单击Chart选项,并确认Bar chart选项已经被选择。若想制作pie图,此处选择pie chart选项。
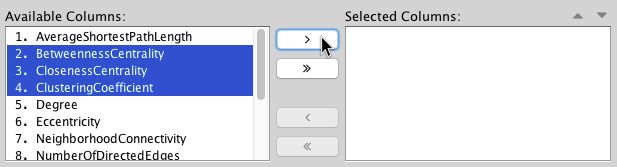
- 现在你可以根据自己需要来选择Node table中要展示的数据了。Available Columns窗口展示了所有可以用于作图的数据。
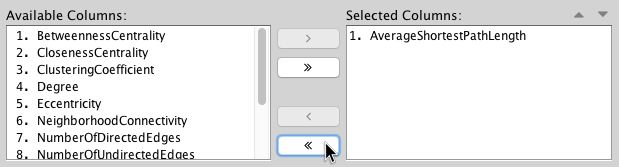
首先单击Remove All 按钮移除当前所有被选择的列(默认情况下,cytoscape会选择Available Columns的第一列)。
然后从Available Columns选择所有的Betweennesscentrality、ClosenessCentrality性质和ClusteringCoefficient列,并单击添加按钮,数据将被添加到右侧的选择列窗口。
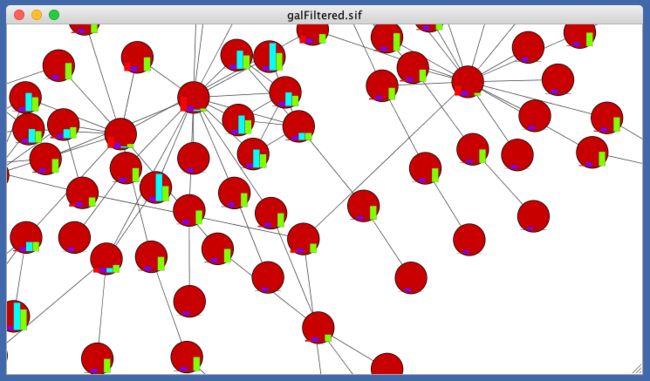
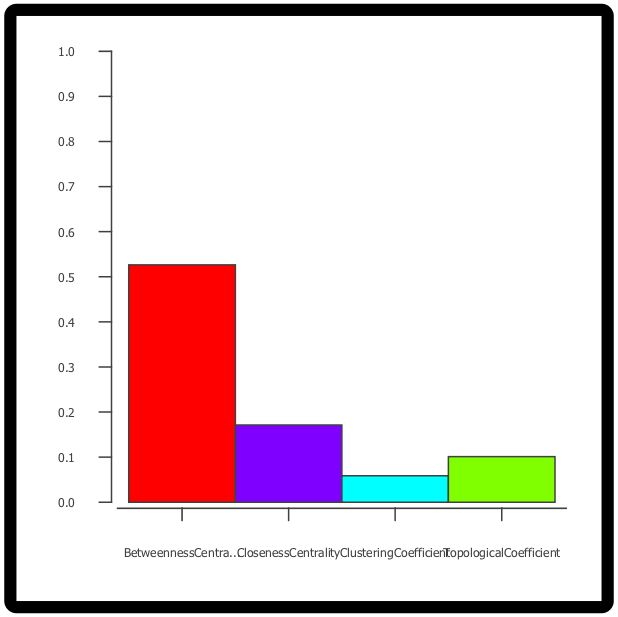
- 点击右下角的Apply应用按钮,被选择的数据将以bar plot的形式展示在节点上。
- 修改结点颜色和形状
从图上可以看出圆形的节点并不适合展示bar plot图,因此我们可以将节点的形状改为正方形,填充颜色改为白色。方法为左侧属性中的Fill Color和Shape选项,最左边的按扭进行点击修改。
- 查看结点
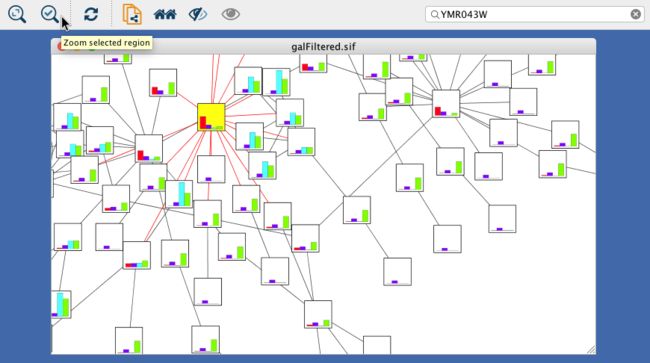
单击选择一个节点,选中时为黄色高亮。并选择上面工具栏中Zoom selected region放大查看细节(点击旁边的Zoom out挖扭退回查看完整网络),也可以根据节点名字从右上方的搜索框搜索感兴趣的节点。
- 根据自己需要改变一些默认参数
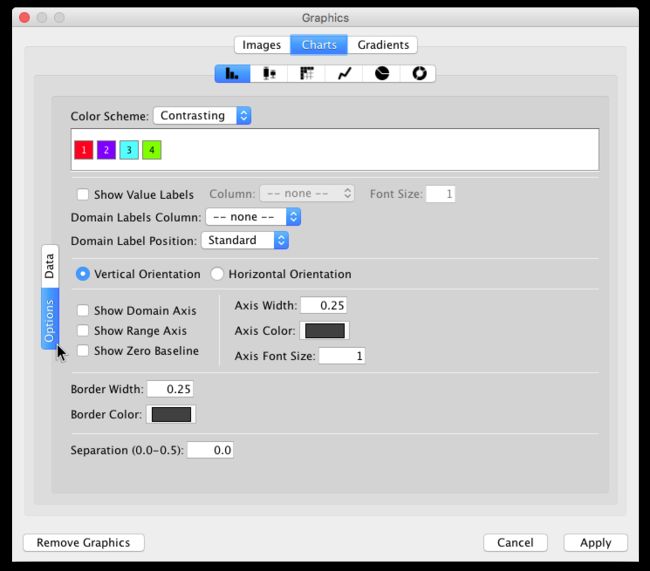
再次单击 Image/Chart 1 选项处默认格子以打开Graphics 会话窗口。选择左下方的 Options按钮,然后可以根据自己需求设置颜色、标签、展示或者隐藏坐标轴、改变线宽和增加bar之间的距离。选择Show Domain Axis 和 Show Range Axis选项并在此点击应用,我们可以看到bar plot增加了x轴和y轴。
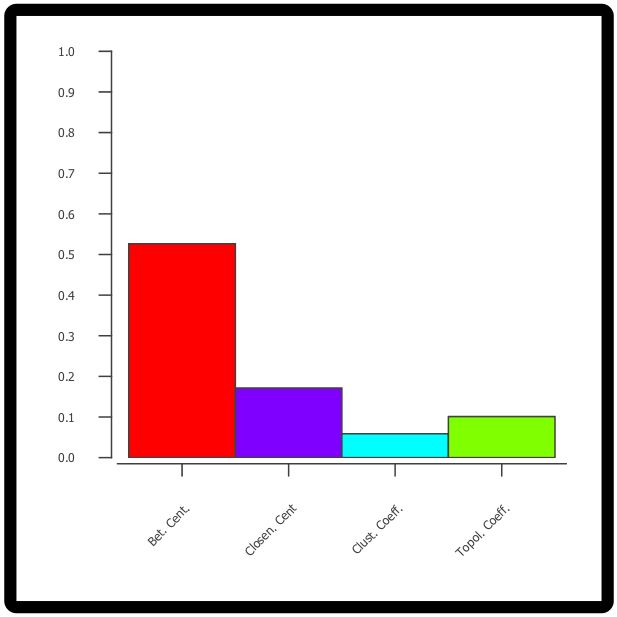
- 修改美化bar图标签
此时我们看到bar默认的label在图上显示出来并不好看,实际上我们可以根据自己需要来改变此标签。
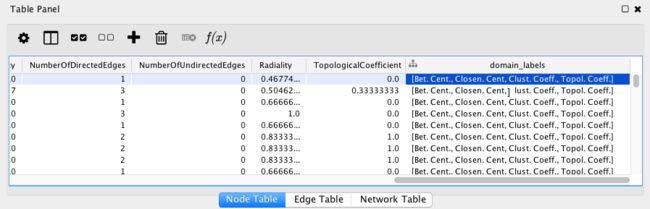
在Table面板(网络图下方)处,我们点击加号,选择 New List Column → String,并命名为domain_labels。在新增加列(托到下方滚动条至最右端)的任意单元格双击进入编辑模式,然后键入[“Bet. Cent.”,”Closen. Cent”,”Clust. Coeff.”,”Topol. Coeff.”],然后右击相同的单元格,选择Apply to entire column。
再次打开bar plot编辑面板,选择Options按钮,在Domain Labels Column 下拉框中选择 “domain_labels”列,在Domain Labels Position下拉框中选择UP 45度,然后应用。结果如下图所示。
参考文献
http://manual.cytoscape.org/en/stable/Styles.html
猜你喜欢
- 10000+: 菌群分析
宝宝与猫狗 提DNA发Nature 实验分析谁对结果影响大 Cell微生物专刊 肠道指挥大脑 - 系列教程:微生物组入门 Biostar 微生物组 宏基因组
- 专业技能:生信宝典 学术图表 高分文章 不可或缺的人
- 一文读懂:宏基因组 寄生虫益处 进化树
- 必备技能:提问 搜索 Endnote
- 文献阅读 热心肠 SemanticScholar Geenmedical
- 扩增子分析:图表解读 分析流程 统计绘图
- 16S功能预测 PICRUSt FAPROTAX Bugbase Tax4Fun
- 在线工具:16S预测培养基 生信绘图
- 科研经验:云笔记 云协作 公众号
- 编程模板: Shell R Perl
- 生物科普: 肠道细菌 人体上的生命 生命大跃进 细胞暗战 人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外2000+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA