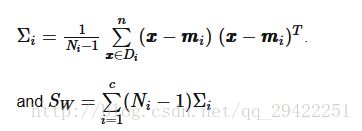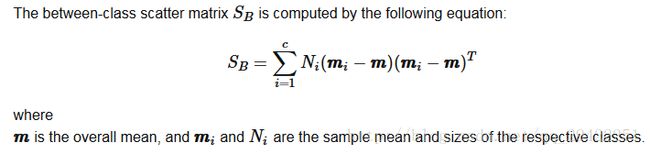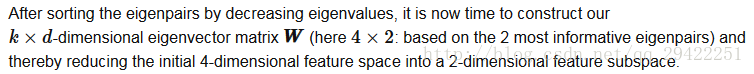LDA(Linear Discriminant Analysis)
1.介绍
线性判别分析(LDA)最常用作模式分类和机器学习应用的预处理步骤中的降维技术。目标是将数据集投影到具有良好类可分性的较低维空间上,以避免过度拟合(“维数灾难”)并降低计算成本。
Ronald A. Fisher于1936年提出了线性判别式(在分类问题中使用多重测量),并且它还具有一些实际用途作为分类器。原始的线性判别式被描述为一个2类问题,并且后来被CR Rao在1948年推广为“多类线性判别分析”或“多判别分析”(在生物分类问题中利用多重测量)。
LDA的目标往往是将特征空间(数据集n维样本)投影到较小的子空间k(其中k≤n-1),同时保持类别的判别信息。一般来说,降维不仅有助于降低给定分类任务的计算成本,而且可以在最小化参数估计误差(“维度诅咒”)中避免过拟合。
1.1 主成分分析与线性判别分析
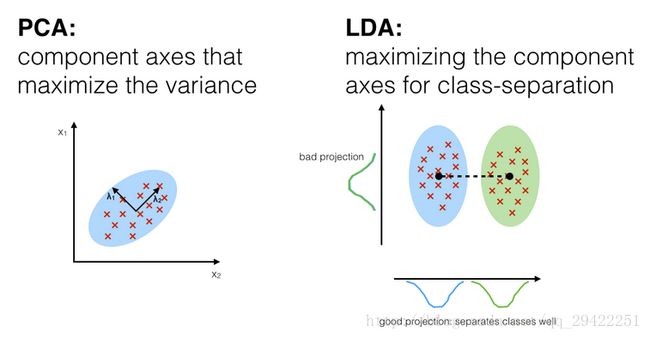
线性判别分析(LDA)和主成分分析(PCA)都是线性变换技术,通常用于降维。 PCA可以被描述为“无监督”算法,因为它“忽略”类标签,其目标是找到使数据集方差最大化的方向(所谓的主成分)。与PCA相反,LDA是“监督的”并计算方向(“线性判别式”),该方向的轴线能够最大化分割多个类别。
虽然LDA在类别标签已知的情况下,多分类任务中优于PCA可能听起来很直观,但情况并非总是如此。
例如,使用PCA或LDA后的图像识别的分类精度比较显示,如果每个类别的样本数量相对较少,则PCA往往优于LDA。在实践中,同时使用LDA和PCA也并不少见:例如,PCA降维后再用LDA进行降维。
1.2 什么是“好”特征子空间?
让我们假设,将d维的数据集投影到k维空间中(where k
我们将从我们的数据集中计算特征向量(分量),并将用散度矩阵来统计它们(即类间散度矩阵和类内散度矩阵)中。 每一个特征向量都与特征值相关联,该特征值告诉我们关于特征向量的“长度”或“幅度”。
如果我们观察到所有特征值都具有相似的幅度,那么这可能是一个很好的指标,即我们的数据已经投影到“良好”特征空间。而在另一种情况下,如果某些特征值比其他特征值大得多,我们可能有兴趣只保留具有最高特征值的特征向量,因为它们包含有关我们数据分布的更多信息。反之亦然,接近于0的特征值信息量较少,我们可能会考虑删除那些用于构造新特征子空间的特征值。
1.3 5步总结LDA方法
下面列出了执行线性判别分析的5个一般步骤,我们将在以下部分更详细地探讨它们。
- 计算数据集中不同类别的d维平均向量
- 计算散度矩阵(类间散度和类内散度)
- 计算散度矩阵的特征向量(e1,e2,....,ed)和对应的特征值(λ1,λ2....,λd)
- 对特征值进行降序,选择具有最大特征值的k个特征向量来形成d x k维矩阵W(每列代表一个特征向量)
- 使用 d x k 维特征向量将原数据集投影到新的子空间中,可以使用矩阵相乘表示:Y=X x W, X是一个n x d 维矩阵,n表示样本个数,y是变换矩阵,将数据集从n x d维空间转换到新的子空间中
2 准备数据
2.1 Iris 数据集
对于下面的教程,我们将使用已存入UCI机器学习存储库中的“Iris”数据集(https://archive.ics.uci.edu/ml/datasets/Iris).
Iris数据集包含来自三种不同物种的150种鸢尾花的测量结果。
Iris数据集中包含以下三个类:
- Iris-setosa (n=50)
- Iris-versicolor (n=50)
- Iris-virginica (n=50)
Iris数据集的四个特征:
- sepal length in cm
- sepal width in cm
- petal length in cm
- petal width in cm

feature_dict = {i:label for i,label in zip(
range(4),
('sepal length in cm',
'sepal width in cm',
'petal length in cm',
'petal width in cm', ))}
2.2 查看数据集中的数据
import pandas as pd
import numpy as np
from sklearn.preprocessing import LabelEncoder
df = pd.io.parsers.read_csv(
filepath_or_buffer='https://archive.ics.uci.edu/ml/machine-learning-databases/iris/iris.data',
header=None,
sep=',',
)
#df.to_csv('out.csv')
#print df
X = df[[0,1,2,3]].values
#print X
df.columns = [l for i,l in sorted(feature_dict.items())] + ['class label']
#print df.columns
df.dropna(how="all", inplace=True) # to drop the empty line at file-end
print df.tail()
y = df['class label'].values
#print y
enc = LabelEncoder()
label_encoder = enc.fit(y)
y = label_encoder.transform(y) + 1
print y
label_dict = {1: 'Setosa', 2: 'Versicolor', 3:'Virginica'}
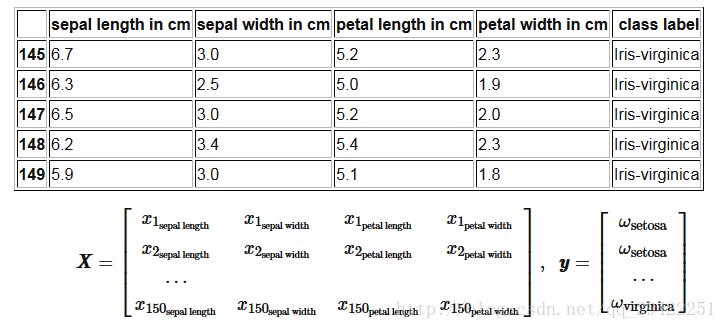
由于使用数值更方便,我们将使用scikit-learn库中的LabelEncode将类标签转换为数字:1,2和3。
2.3 直方图和特征选取
为了粗略地了解我们的三个类别ω1,ω2和ω3的样本是如何分布的,让我们将一维直方图中对四个不同特征的分布进行可视化.(下面代码有问题)
%matplotlib inline
from matplotlib import pyplot as plt
import numpy as np
import math
fig, axes = plt.subplots(nrows=2, ncols=2, figsize=(12,6))
for ax,cnt in zip(axes.ravel(), range(4)):
# set bin sizes
min_b = math.floor(np.min(X[:,cnt]))
max_b = math.ceil(np.max(X[:,cnt]))
bins = np.linspace(min_b, max_b, 25)
# plottling the histograms
for lab,col in zip(range(1,4), ('blue', 'red', 'green')):
ax.hist(X[y==lab, cnt],
color=col,
label='class %s' %label_dict[lab],
bins=bins,
alpha=0.5,)
ylims = ax.get_ylim()
# plot annotation
leg = ax.legend(loc='upper right', fancybox=True, fontsize=8)
leg.get_frame().set_alpha(0.5)
ax.set_ylim([0, max(ylims)+2])
ax.set_xlabel(feature_dict[cnt])
ax.set_title('Iris histogram #%s' %str(cnt+1))
# hide axis ticks
ax.tick_params(axis="both", which="both", bottom="off", top="off",
labelbottom="on", left="off", right="off", labelleft="on")
# remove axis spines
ax.spines["top"].set_visible(False)
ax.spines["right"].set_visible(False)
ax.spines["bottom"].set_visible(False)
ax.spines["left"].set_visible(False)
axes[0][0].set_ylabel('count')
axes[1][0].set_ylabel('count')
fig.tight_layout()
plt.show()
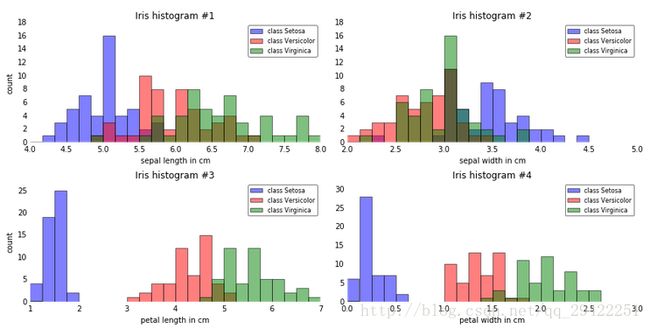
从查看这些简单的图形表征可以看出,花瓣的长度和宽度可能更适合三种花类之间的潜在特征。在实践中,不只是通过投影(这里:LDA)来降低维度,这里有一种很好的替代方法,那就是特征选择技术。对于像Iris这样的低维数据集,浏览这些直方图已经非常有用。另一个简单但非常有用的技术是使用特征选择算法;如果您有兴趣,我可以在这里更详细地描述顺序特征选择算法,而scikit-learn也可以很好地选择其他方法。对于不同方法的高级总结,我写了一篇关于“过滤器,包装器和用于特征选择的嵌入式方法之间有什么区别?”的短文。
2.4 正态性假设
应该提到的是,LDA假定数据服从正态分布,每个类别具有统计独立的特征以及相同的协方差矩阵。但是,这仅适用于LDA,因为如果这些假设被违反,则用于降维的分类器的LDA也可以相当好地工作。即使对于分类任务,LDA似乎也可以对数据的分布非常稳健。
3. 5步探讨LDA
在我们完成了几个准备步骤之后,我们的数据终于可以用于实际的LDA了。实际上,降维的LDA只是典型机器学习或模式分类任务的另一个预处理步骤。
步骤1:计算d维平均向量
在这第一步中,我们将首先简单计算3个不同花类的平均向量mi, (i=1,2,3)
np.set_printoptions(precision=4)
#print y
#print 'y==1','\n',X[y==1]
#print 'y==2','\n',X[y==2]
mean_vectors = []
for cl in range(1,4):
mean_vectors.append(np.mean(X[y==cl], axis=0))
print 'Mean Vector class %s:',cl, mean_vectors[cl-1]Mean Vector class 1: [ 5.006 3.418 1.464 0.244]
Mean Vector class 2: [ 5.936 2.77 4.26 1.326]
Mean Vector class 3: [ 6.588 2.974 5.552 2.026]步骤2:计算散度矩阵
现在,我们将计算两个4×4维矩阵:类内和类间散度矩阵。2.1 类内散度矩阵
S_W = np.zeros((4,4))
for cl,mv in zip(range(1,4), mean_vectors):
class_sc_mat = np.zeros((4,4)) # scatter matrix for every class
for row in X[y == cl]:
row, mv = row.reshape(4,1), mv.reshape(4,1) # make column vectors
class_sc_mat += (row-mv).dot((row-mv).T)
S_W += class_sc_mat # sum class scatter matrices
print('within-class Scatter Matrix:\n', S_W)within-class Scatter Matrix:
[[ 38.9562 13.683 24.614 5.6556]
[ 13.683 17.035 8.12 4.9132]
[ 24.614 8.12 27.22 6.2536]
[ 5.6556 4.9132 6.2536 6.1756]]或者,我们也可以在类内散度矩阵中添加尺度因子1/N-1来计算中类别协方差矩阵,于是,我们的公式变为;
其中Ni是相应类别的样本大小(这里是50),并且在这种特殊情况下,因为所有类别都具有相同的样本大小,所以我们采用无偏估计Ni-1个样本作为分母。
然而,由此产生的特征空间将是相同的(相同的特征向量,只有特征值按等比例缩放)。
2.2 类间散度矩阵
overall_mean = np.mean(X, axis=0)
S_B = np.zeros((4,4))
for i,mean_vec in enumerate(mean_vectors):
n = X[y==i+1,:].shape[0]
mean_vec = mean_vec.reshape(4,1) # make column vector
overall_mean = overall_mean.reshape(4,1) # make column vector
S_B += n * (mean_vec - overall_mean).dot((mean_vec - overall_mean).T)
print('between-class Scatter Matrix:\n', S_B)
between-class Scatter Matrix:
[[ 63.2121 -19.534 165.1647 71.3631]
[ -19.534 10.9776 -56.0552 -22.4924]
[ 165.1647 -56.0552 436.6437 186.9081]
[ 71.3631 -22.4924 186.9081 80.6041]]S−1WS
步骤3:求解矩阵广义特征值问题
eig_vals, eig_vecs = np.linalg.eig(np.linalg.inv(S_W).dot(S_B))
for i in range(len(eig_vals)):
eigvec_sc = eig_vecs[:,i].reshape(4,1)
print('\nEigenvector {}: \n{}'.format(i+1, eigvec_sc.real))
print('Eigenvalue {:}: {:.2e}'.format(i+1, eig_vals[i].real))
Eigenvector 1:
[[-0.2049]
[-0.3871]
[ 0.5465]
[ 0.7138]]
Eigenvalue 1: 3.23e+01
Eigenvector 2:
[[-0.009 ]
[-0.589 ]
[ 0.2543]
[-0.767 ]]
Eigenvalue 2: 2.78e-01
Eigenvector 3:
[[ 0.179 ]
[-0.3178]
[-0.3658]
[ 0.6011]]
Eigenvalue 3: -4.02e-17
Eigenvector 4:
[[ 0.179 ]
[-0.3178]
[-0.3658]
[ 0.6011]]
Eigenvalue 4: -4.02e-17如果我们正在执行降维的LDA,则特征向量很重要,因为它们将形成新特征子空间的新轴;相关的特征值是特别感兴趣的,因为它们会告诉我们新的“轴”是如何“有信息”的。让我们简单地仔细检查我们的计算,并在下一节中详细讨论特征值。
检查计算的特征向量-特征值
for i in range(len(eig_vals)):
eigv = eig_vecs[:,i].reshape(4,1)
np.testing.assert_array_almost_equal(np.linalg.inv(S_W).dot(S_B).dot(eigv),
eig_vals[i] * eigv,
decimal=6, err_msg='', verbose=True)
print('ok')
ok步骤4:为新的特征子空间选择线性判别式
4.1 通过对特征值降序来排列特征向量
从介绍中还记得,我们不仅仅希望将数据投影到子空间中,以提高类的可分性,而且还希望降低我们的特征空间的维数(特征向量将形成这个新特征子空间的轴)。
但是,特征向量仅定义新轴的方向,因为它们具有相同的单位长度1。
因此,为了确定我们想要为我们的低维子空间丢弃哪些特征向量,我们必须看看特征向量的相应特征值。粗略地说,具有最低特征值的特征向量具有关于数据分布的最少信息,而那些是我们想要放弃的特征向量。
常用的方法是将特征向量从最高到最低对应的特征值进行排序,并选择最大的k个特征值所对应的特征向量。
# Make a list of (eigenvalue, eigenvector) tuples
eig_pairs = [(np.abs(eig_vals[i]), eig_vecs[:,i]) for i in range(len(eig_vals))]
# Sort the (eigenvalue, eigenvector) tuples from high to low
eig_pairs = sorted(eig_pairs, key=lambda k: k[0], reverse=True)
# Visually confirm that the list is correctly sorted by decreasing eigenvalues
print('Eigenvalues in decreasing order:\n')
for i in eig_pairs:
print(i[0])
Eigenvalues in decreasing order:
32.2719577997
0.27756686384
5.01086753497e-15
3.90511902684e-15 现在,我们用百分比来表示“解释性差异“:
print('Variance explained:\n')
eigv_sum = sum(eig_vals)
for i,j in enumerate(eig_pairs):
print('eigenvalue {0:}: {1:.2%}'.format(i+1, (j[0]/eigv_sum).real))
Variance explained:
eigenvalue 1: 99.15%
eigenvalue 2: 0.85%
eigenvalue 3: 0.00%
eigenvalue 4: 0.00%4.2 根据最大的特征值选择K个特征向量
上面描述:将原始数据从4维空间中投影到2维空间中
W = np.hstack((eig_pairs[0][1].reshape(4,1), eig_pairs[1][1].reshape(4,1)))
print('Matrix W:\n', W.real)
Matrix W:
[[ 0.2049 -0.009 ]
[ 0.3871 -0.589 ]
[-0.5465 0.2543]
[-0.7138 -0.767 ]]步骤5:将样本转换到新的子空间中
X_lda = X.dot(W)
assert X_lda.shape == (150,2), "The matrix is not 150x2 dimensional."
#print X_ldafrom matplotlib import pyplot as plt
def plot_step_lda():
ax = plt.subplot(111)
for label,marker,color in zip(
range(1,4),('^', 's', 'o'),('blue', 'red', 'green')):
plt.scatter(x=X_lda[:,0].real[y == label],
y=X_lda[:,1].real[y == label],
marker=marker,
color=color,
alpha=0.5,
label=label_dict[label]
)
plt.xlabel('LD1')
plt.ylabel('LD2')
leg = plt.legend(loc='upper right', fancybox=True)
leg.get_frame().set_alpha(0.5)
plt.title('LDA: Iris projection onto the first 2 linear discriminants')
# hide axis ticks
plt.tick_params(axis="both", which="both", bottom="off", top="off",
labelbottom="on", left="off", right="off", labelleft="on")
# remove axis spines
ax.spines["top"].set_visible(False)
ax.spines["right"].set_visible(False)
ax.spines["bottom"].set_visible(False)
ax.spines["left"].set_visible(False)
plt.grid()
plt.tight_layout
plt.show()
plot_step_lda()
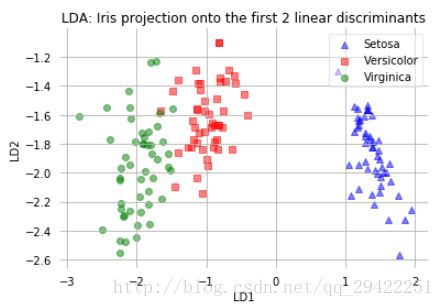
上面的散点图表示我们通过LDA构建的新特征子空间。我们可以看到第一个线性判别式“LD1”很好地分离了这些类。然而,第二个判别式“LD2”没有增加太多有价值的信息,当我们查看排列的特征值时,我们其实已经可以预测分类的结果
原文地址: http://sebastianraschka.com/Articles/2014_python_lda.html#lda-in-5-steps