Python无监督学习3
聚类
聚类(clustering)将数据划分为组,这些组叫作簇。聚类算法为每个数字分配一个数字,表示这个点属于哪个簇。
1.K均值聚类
它试图找到聚类的簇中心,将每个数据点分配到最近的簇中心,然后每个簇中心设置为分配的所有点的平均值。
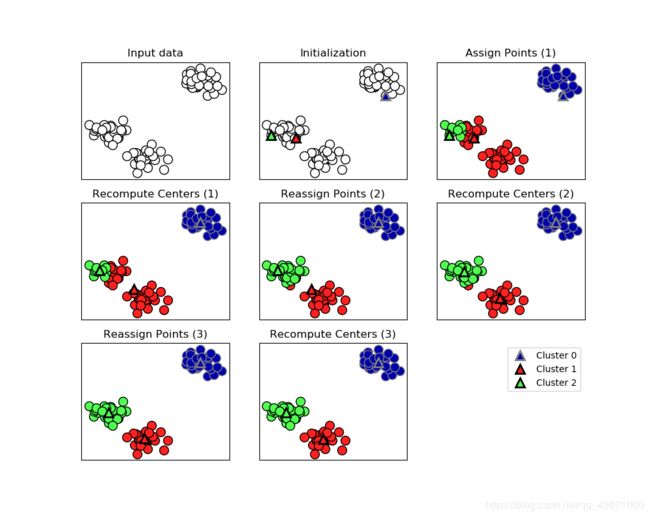
mglearn.plots.plot_kmeans_algorithm()
plt.show()先初始化。分配数据点,重新计算中心,一共迭代了3次。
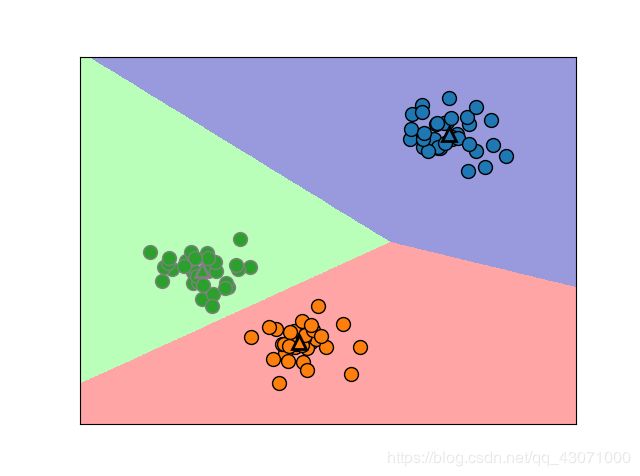
mglearn.plots.plot_kmeans_boundaries()
plt.show()会发现分类的那个线线好像不是垂直平分线找两个点的最短距离来的。那是怎么来的呢?
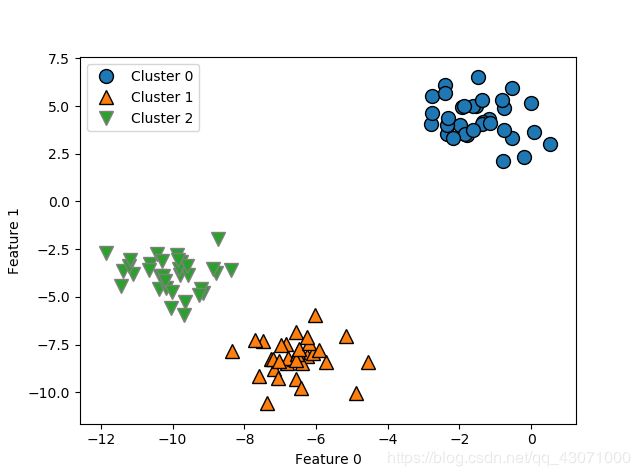
from sklearn.datasets import make_blobs
from sklearn.cluster import KMeans
# 生成模拟的二维数据
X, y = make_blobs(random_state=1)
# 构建聚类模型
kmeans = KMeans(n_clusters=3)
kmeans.fit(X)运行期间,为X中的每个训练数据点分配一个簇标签,我们可以在 kmeans.labels_ 属性里找到标签。
print("Cluster memberships:\n {}".format(kmeans.labels_))这里分为3个类,所以标签为0,1,2。我们也可以用 predict 方法为新数据分配标签,但现有模型不会改变。
print(kmeans.predict(X))这里,标签本身没有先验意义。可能每一次簇的分类都不一样,原因在于初始化的随即性质!!!初始化,憨批!
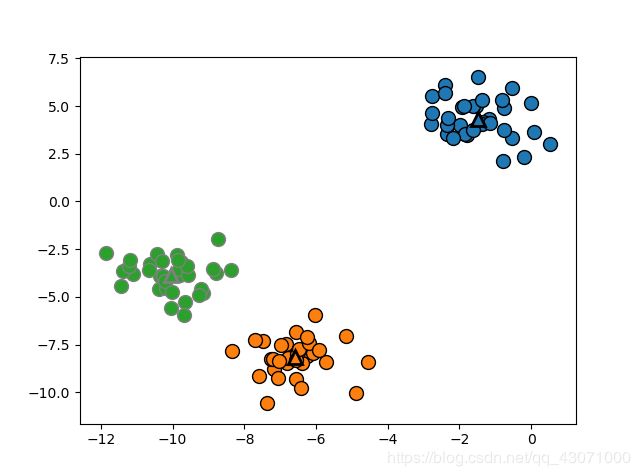
mglearn.discrete_scatter(X[:, 0], X[:, 1], kmeans.labels_, markers='o')
mglearn.discrete_scatter(
kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1], [0, 1, 2],
markers='^', markeredgewidth=2)
plt.show()我们可以使用更多或更少的簇中心。
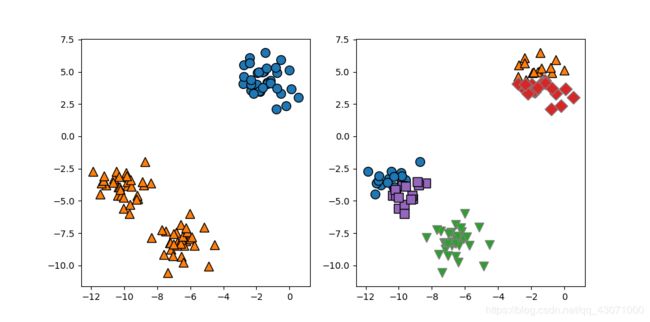
X, y = make_blobs(random_state=1)
fig, axes = plt.subplots(1, 2, figsize=(10, 5))
# 使用2个簇中心:
kmeans = KMeans(n_clusters=2)
kmeans.fit(X)
assignments = kmeans.labels_
mglearn.discrete_scatter(X[:, 0], X[:, 1], assignments, ax=axes[0])
# 使用5个簇中心:
kmeans = KMeans(n_clusters=5)
kmeans.fit(X)
assignments = kmeans.labels_
mglearn.discrete_scatter(X[:, 0], X[:, 1], assignments, ax=axes[1])
plt.show()1.1k均值失败的案例
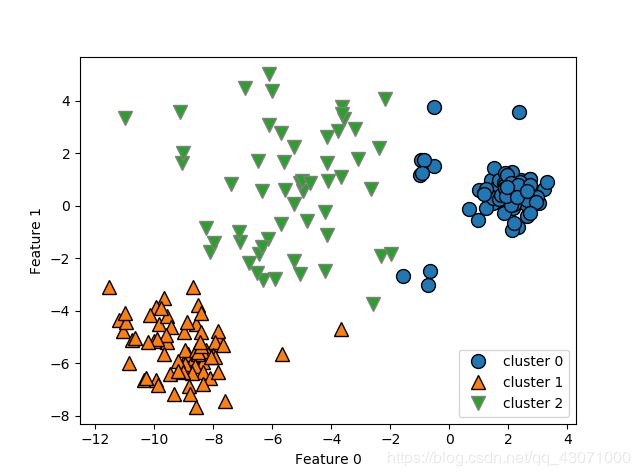
即使我们知道分类的个数,它也不一定就能很好的找到他们。每个簇仅由其中心定义,这意味着每个簇都是凸形(convex)。因此,k均值只能找到相对简单的形状。而且它总是将簇之间的边界刚好画在簇中心的中间位置。
X_varied, y_varied = make_blobs(n_samples=200,
cluster_std=[1.0, 2.5, 0.5],
random_state=170)
y_pred = KMeans(n_clusters=3, random_state=0).fit_predict(X_varied)
mglearn.discrete_scatter(X_varied[:, 0], X_varied[:, 1], y_pred)
plt.legend(["cluster 0", "cluster 1", "cluster 2"], loc='best')
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()你会发现左下角和右上角的簇会包含一些离他们很远的点,因为他们的边界找的簇中心中间的位置呢。k均值假设所有方向对每个簇都很重要,所以如果是一个长条条它只是某个方向比较重要,其他方向重要性不大,那么k均值就无法处理这种情况。
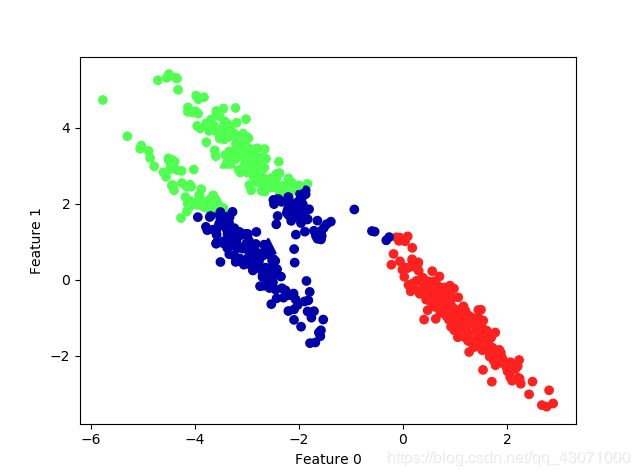
# 生成一些随机分组数据
X, y = make_blobs(random_state=170, n_samples=600)
rng = np.random.RandomState(74)
# 变换数据使其拉长
transformation = rng.normal(size=(2, 2))
X = np.dot(X, transformation)
# 将数据聚类成3个簇
kmeans = KMeans(n_clusters=3)
kmeans.fit(X)
y_pred = kmeans.predict(X)
# 画出簇分配和簇中心
plt.scatter(X[:, 0], X[:, 1], c=y_pred, cmap=mglearn.cm3)
plt.scatter(kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1],
marker='^', c=[0, 1, 2], s=100, linewidths=2, cmap=mglearn.cm3)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()通俗点就是k均值无法识别非球簇。比如下面也是。
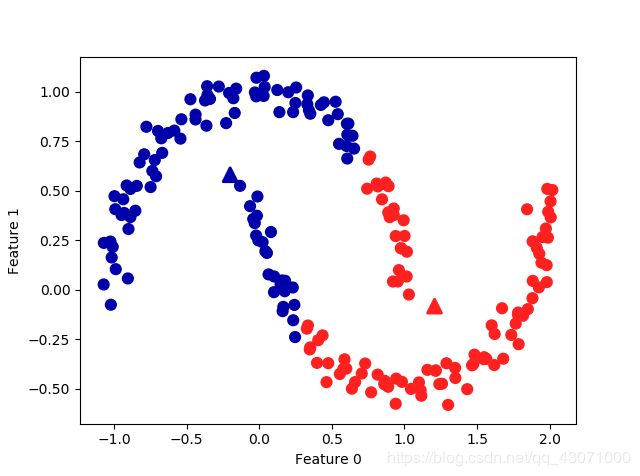
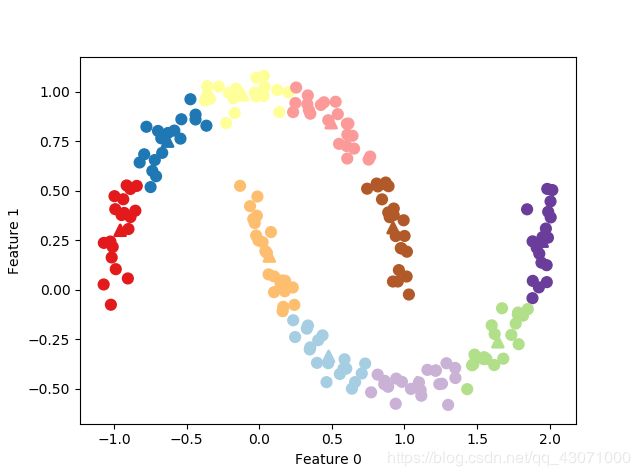
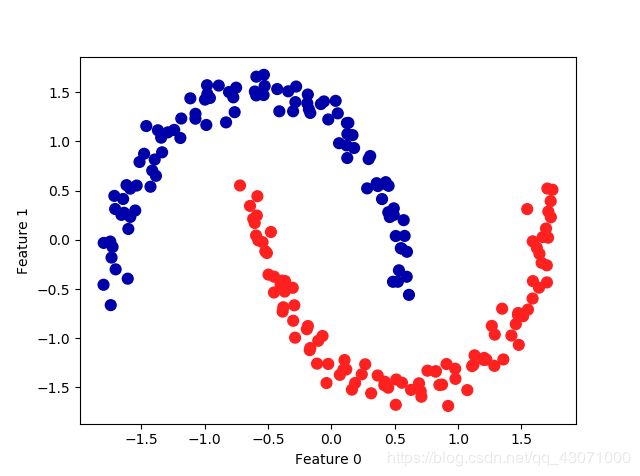
# 生成模拟的two_moons数据(这次的噪声较小)
from sklearn.datasets import make_moons
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据聚类成2个簇
kmeans = KMeans(n_clusters=2)
kmeans.fit(X)
y_pred = kmeans.predict(X)
# 画出簇分配和簇中心
plt.scatter(X[:, 0], X[:, 1], c=y_pred, cmap=mglearn.cm2, s=60)
plt.scatter(kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1],
marker='^', c=[mglearn.cm2(0), mglearn.cm2(1)], s=100, linewidths=2)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()k均值小弟弟无法分类呢~
1.2.矢量量化,或者将k均值看作分解
k均值和分解方法存在一些相似之处。PCA试图找到数据中方差最大的方向,而NMF试图找到累加的分量,这通常对应于数据的“极值”或“部分”。与之相反,K均值则尝试利用簇中心来表示每个数据点。这种用单一分量表示的观点,被称为矢量量化(vector quantization)。
from sklearn.datasets import fetch_lfw_people
from sklearn.decomposition import NMF
from sklearn.decomposition import PCA
people = fetch_lfw_people(min_faces_per_person=20, resize=0.7)
image_shape = people.images[0].shape
mask = np.zeros(people.target.shape, dtype=np.bool)
for target in np.unique(people.target):
mask[np.where(people.target == target)[0][:50]] = 1
X_people = people.data[mask]
y_people = people.target[mask]
X_train, X_test, y_train, y_test = train_test_split(
X_people, y_people, stratify=y_people, random_state=0)
nmf = NMF(n_components=100, random_state=0)
nmf.fit(X_train)
pca = PCA(n_components=100, random_state=0)
pca.fit(X_train)
kmeans = KMeans(n_clusters=100, random_state=0)
kmeans.fit(X_train)
X_reconstructed_pca = pca.inverse_transform(pca.transform(X_test))
X_reconstructed_kmeans = kmeans.cluster_centers_[kmeans.predict(X_test)]
X_reconstructed_nmf = np.dot(nmf.transform(X_test), nmf.components_)
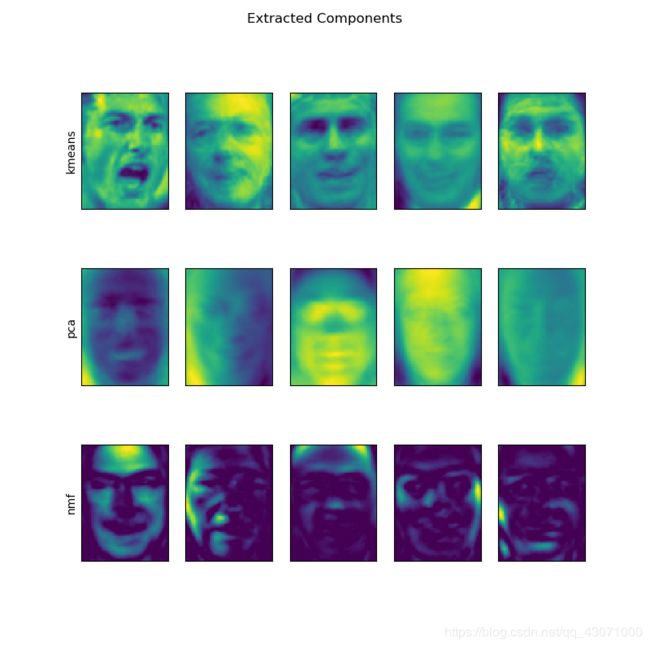
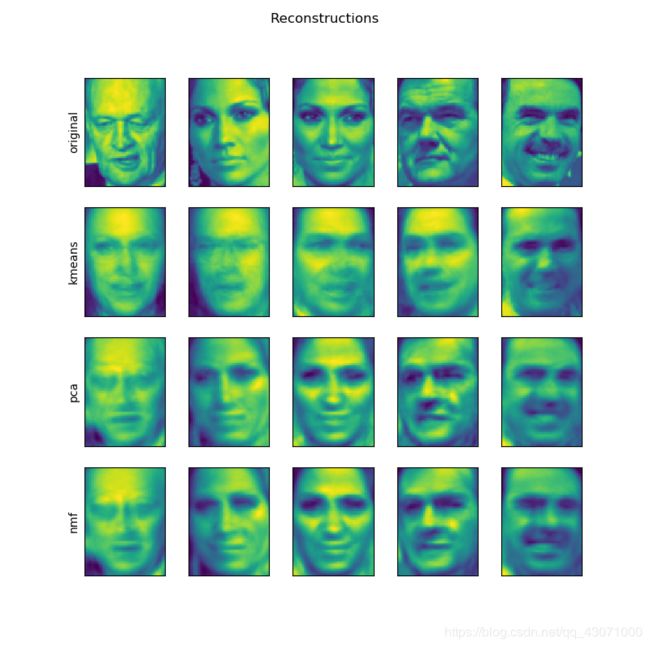
fig, axes = plt.subplots(3, 5, figsize=(8, 8),
subplot_kw={'xticks': (), 'yticks': ()})
fig.suptitle("Extracted Components")
for ax, comp_kmeans, comp_pcam, comp_nmf in zip(
axes.T, kmeans.cluster_centers_, pca.components_, nmf.components_):
ax[0].imshow(comp_kmeans.reshape(image_shape))
ax[1].imshow(comp_pcam.reshape(image_shape))
ax[2].imshow(comp_nmf.reshape(image_shape))
axes[0, 0].set_ylabel("kmeans")
axes[1, 0].set_ylabel("pca")
axes[2, 0].set_ylabel("nmf")
fig, axes = plt.subplots(4, 5, subplot_kw={'xticks': (), 'yticks': ()},
figsize=(8, 8))
fig.suptitle("Reconstructions")
for ax, orig, rec_kmeans, rec_pca, rec_nmf in zip(
axes.T, X_test, X_reconstructed_kmeans, X_reconstructed_pca, X_reconstructed_nmf):
ax[0].imshow(orig.reshape(image_shape))
ax[1].imshow(rec_kmeans.reshape(image_shape))
ax[2].imshow(rec_pca.reshape(image_shape))
ax[3].imshow(rec_nmf.reshape(image_shape))
axes[0, 0].set_ylabel("original")
axes[1, 0].set_ylabel("kmeans")
axes[2, 0].set_ylabel("pca")
axes[3, 0].set_ylabel("nmf")
plt.show()对于二维数据two_moons,利用PCA和NMF我们无能为力,降到意味损失太多数据,但可以通过使用更多的簇中心让k均值去变现它们,不错。
from sklearn.datasets import make_moons
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据聚类成2个簇
kmeans = KMeans(n_clusters=10, random_state=0)
kmeans.fit(X)
y_pred = kmeans.predict(X)
# 画出簇分配和簇中心
plt.scatter(X[:, 0], X[:, 1], c=y_pred, s=60, cmap='Paired')
plt.scatter(kmeans.cluster_centers_[:, 0], kmeans.cluster_centers_[:, 1], s=60,
marker='^', c=range(kmeans.n_clusters), linewidths=2, cmap='Paired')
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()
print("Cluster memberships:\n {}".format(y_pred))现在有10个分量,只有对应的簇中心的特征不为0,其余特征均为0。利用这个10维表示,现在可以用线性模型来划分了。将到每个簇中心的距离作为特征,还可以得到一种变现里更强的数据表示。
distance_features = kmeans.transform(X)
print("Distance feature shape: {}".format(distance_features.shape))
print("Distance features:\n {}".format(distance_features))
k均值运行速度蛮快的,可以轻松扩展到大型数据集,scikit-learn 甚至在 MiniBatchMeans 类中包含了一种更具可扩展性的变体。k均值的缺点之一在于它依赖于随机初始化,默认情况,scikit-learn 随机10次,返回最好的结果。k均值还有一个缺点,对簇形状的约束性较强。
2.凝聚聚类
凝聚聚类(agglomerative clustering)指的是许多基于相同原则构建的聚类算法,原则:算法先声明每个点是自己的簇,然后再合并两个最相似的簇,直到满足某种停止准则(簇的个数),好像那个二叉树从下往上啊。链接(linkage)准则,规定如何度量 “最相似的簇” ,这种度量总是定义在两个现有的簇之间。scikit-learn 有以下三个选项:
ward 默认选项。ward挑选两个簇来合并,使得所有簇中的方差增加最小。得到大小差不多的簇。
average averagr 链接将簇中所有点之间平均距离最小的两个簇合并。
complete complete 链接(也称最大链接)将簇中点之间最大距离最小的两个簇合并。
ward适用大多数数据集,如果簇中成员个数非常不同,average 或 complete 可能会好。
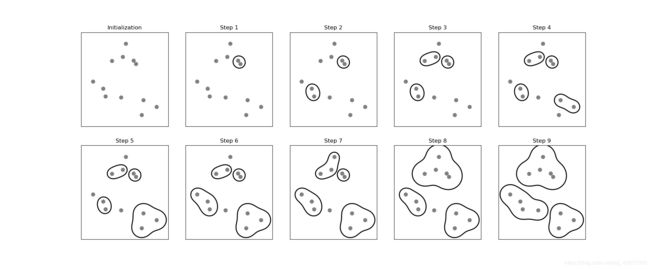
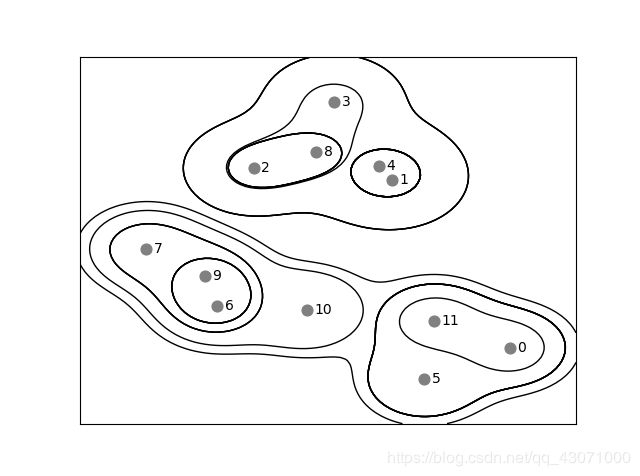
mglearn.plots.plot_agglomerative_algorithm()
plt.show()把它圈起来了,就理解为一个点这样子嘛? 停止准则是3个簇。凝聚算法无法做预测的,因此 AgglomerativeClustering 没有predict 方法,可以改用 fit_predict 方法。
from sklearn.cluster import AgglomerativeClustering
X, y = make_blobs(random_state=1)
agg = AgglomerativeClustering(n_clusters=3)
assignment = agg.fit_predict(X)
mglearn.discrete_scatter(X[:, 0], X[:, 1], assignment)
plt.legend(["Cluster 0", "Cluster 1", "Cluster 2"])
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()完美聚类~
2.1.层次聚类与树状图
凝聚聚类生成了所谓的层次聚类(hierarchical clustering),聚类过程迭代进行。叠加显示呢
mglearn.plots.plot_agglomerative()
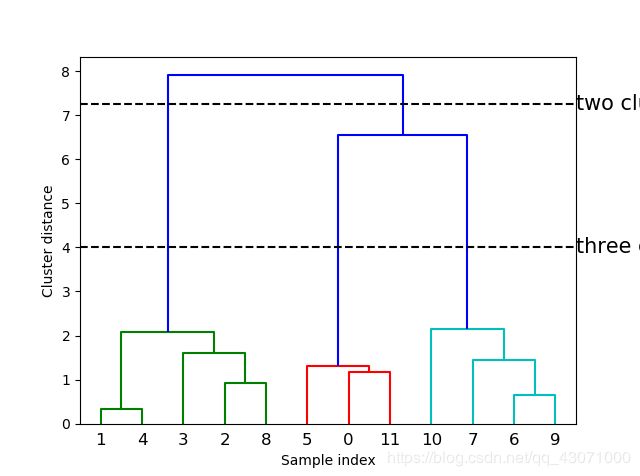
plt.show()这种可视化,我不知道数字的意思,它依赖于数据的二维性质。还有一个可视化工具,树状图(dendrogram),可以处理所谓数据,得用 SciPy,SciPy 提供一个函数,接受数据数组X并计算一个链接数组(linkage array),它对层次聚类的相似度进行编码,然后将链接提供给 dendrograma 函数绘制树状图。
# 从SciPy中导入dendrogram函数和ward聚类函数
from scipy.cluster.hierarchy import dendrogram, ward
X, y = make_blobs(random_state=0, n_samples=12)
# 将ward聚类应用于数据数组X
# SciPy的ward函数返回一个数组,指定执行凝聚聚类时跨越的距离
linkage_array = ward(X)
# 现在为包含簇之间距离的linkage_array绘制树状图
dendrogram(linkage_array)
# 在树中标记划分成两个簇或三个簇的位置
ax = plt.gca()
bounds = ax.get_xbound()
ax.plot(bounds, [7.25, 7.25], '--', c='k')
ax.plot(bounds, [4, 4], '--', c='k')
ax.text(bounds[1], 7.25, 'two clusters', va='center', fontdict={'size': 15})
ax.text(bounds[1], 4, 'three clusters', va='center', fontdict={'size': 15})
plt.xlabel("Sample index")
plt.ylabel("Cluster distance")
plt.show()每个分支的长度表示被合并的簇之间的距离!这个距离是簇中心点之间的距离嘛?? 然而凝聚聚类无法分离 two-moons 这种的。
3.DBSCAN
另一个非常有用的聚类算法是DBSCAN(density-based spatial clustering of applications with noise, 即 “ 具有噪声的基于密度的空间聚类应用 ” )。主要优点是不需要用户先验地设置簇的个数,可以划分具有复杂形状的簇,还可以找出不属于任何簇的点,虽然比k均值慢,但也可以扩展到大型数据集。
原理是识别特征空间的 “ 拥挤 ” 区域的点,他们被称为密集(dense)区域,由较空的区域分开密集的区域。在密集区域内的点被称为核心样本(core sample,或核心点),如果在距一个给定数据点 eps 距离内至少有 min_samples 个数据点,那么这个数据点就是核心样本。它将彼此距离小于 eps 的核心样本放到同一个簇中。
一共由三种类型的点:核心点、与核心点的距离在eps之内的点(叫做边界点,boundary point)和噪声。对访问顺序轻度依赖,因此如果多次运行,那么边界点可能与不止一个簇的核心样本相邻。而且这玩意儿与凝聚聚类类似也不可以预测,所以hi有fit_predict方法用来预测呢。
from sklearn.cluster import DBSCAN
X, y = make_blobs(random_state=0, n_samples=12)
dbscan = DBSCAN()
clusters = dbscan.fit_predict(X)
print("Clusters memeberships:\n {}".format(clusters))所有数据点都被分配为了标签-1,这代表噪声。我们选取不同的 eps 和 min_samples 来康康。
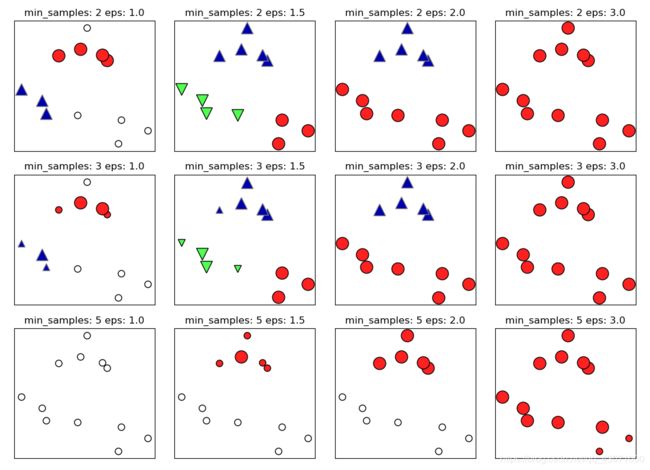
mglearn.plots.plot_dbscan()
plt.show()eps 决定点与点之间 “ 接近 ” 的含义,min_samples 决定簇的最小尺寸。使用 StandardScaler 或 MinMaxScaler 对数据进行缩放之后,有时候更容易找到 eps 的较好取值,因为使用这些缩放技术将确保所有特征具有相似的范围。
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import DBSCAN
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据缩放成平均值为0、方差为1
scaler = StandardScaler()
scaler.fit(X)
X_scaled = scaler.transform(X)
dbscan = DBSCAN()
clusters = dbscan.fit_predict(X_scaled)
# 绘制簇分配
plt.scatter(X_scaled[:, 0], X_scaled[:, 1], c=clusters, cmap=mglearn.cm2, s=60)
plt.xlabel("Feature 0")
plt.ylabel("Feature 1")
plt.show()
如果将 eps 减小到0.2(默认值为0.5), 我们将得到8个簇,增大到0.7,导致只有一个簇。因此需谨慎处理返回的簇分配。
4.聚类算法的对比和评估
上面讨论了k均值、凝聚聚类和 DBSCAN 算法,现在比较一下呢
4.1.用真实值评估聚类
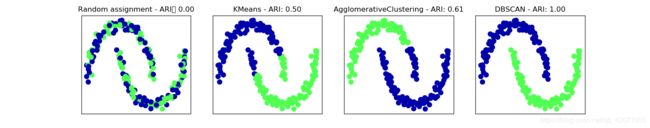
有一个指标也可用于评估聚类算法相对于真实聚类的结果,其中最重要的是调整 rand 指数(adjusted rand index,ARI)和归一化互信息(normalized mutual information,NMI),二者都给出了定量的度量,其最佳取值为1,0表示不相关的聚类(虽然 ARI 可以取负值)。下面对比
from sklearn.metrics.cluster import adjusted_rand_score
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import AgglomerativeClustering
from sklearn.cluster import DBSCAN
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据缩放成平均值为0、方差为1
scaler = StandardScaler()
scaler.fit(X)
X_scaled = scaler.transform(X)
fig, axes = plt.subplots(1, 4, figsize=(15, 3), subplot_kw={'xticks': (), 'yticks': ()})
# 列出要使用的算法
algorithms = [KMeans(n_clusters=2), AgglomerativeClustering(n_clusters=2), DBSCAN()]
# 创建一个随机的簇分配,作为参考
random_state = np.random.RandomState(seed=0)
random_clusters = random_state.randint(low=0, high=2, size=len(X))
# 绘制随机分配
axes[0].scatter(X_scaled[:, 0], X_scaled[:, 1], c=random_clusters, cmap=mglearn.cm3, s=60)
axes[0].set_title("Random assignment - ARI: {:.2f}".format(adjusted_rand_score(y, random_clusters)))
for ax, algorithm in zip(axes[1:], algorithms):
# 绘制簇分配和簇中心
clusters = algorithm.fit_predict(X_scaled)
ax.scatter(X_scaled[:, 0], X_scaled[:, 1], c=clusters, cmap=mglearn.cm3, s=60)
ax.set_title("{} - ARI: {:.2f}".format(algorithm.__class__.__name__,
adjusted_rand_score(y, clusters)))
plt.show()为什么不用 accuracy_score 呢,因为评估的目的不是簇标签是否与真实值相同,标签本身毫无意义----而是数据点们是否在一个簇里。
from sklearn.metrics.cluster import adjusted_rand_score
from sklearn.metrics import accuracy_score
# 这两种点标签对应于相同的聚类
clusters1 = [0, 0, 1, 1, 0]
clusters2 = [1, 1, 0, 0, 1]
# 精度为0,因为二者标签完全不同= =
print("Accuracy: {:.2f}".format(accuracy_score(clusters1, clusters2)))
# 调整 rand 分数为1,因为二者聚类完全相同
print("ARI: {:.2f}".format(adjusted_rand_score(clusters1, clusters2)))2.在没有真实值的情况下评估聚类
在实践中,我们通常没有正确的聚类结果呢。所以如果我们直到了数据的正确聚类,那么可以hi用这一信息构建一个监督模型(比如分类器)。因此,使用类似 ARI 和 NMI 的指标通常进有助于开发算法,但对评估应用是否成功没有帮助。
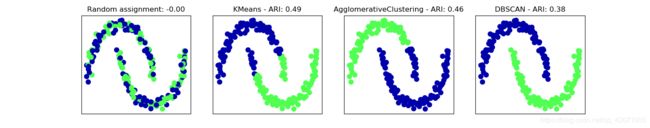
有一些聚类的评分指标并不需要真实值,比如轮廓系数(silhouette coeffcient),但它们在时间中的效果并不好。轮廓分数计算一个簇的紧致度,但它不允许复杂的形状。
from sklearn.metrics.cluster import silhouette_score
from sklearn.preprocessing import StandardScaler
from sklearn.cluster import AgglomerativeClustering
from sklearn.cluster import DBSCAN
X, y = make_moons(n_samples=200, noise=0.05, random_state=0)
# 将数据缩放成平均值为0、方差为1
scaler = StandardScaler()
scaler.fit(X)
X_scaled = scaler.transform(X)
fig, axes = plt.subplots(1, 4, figsize=(15, 3), subplot_kw={'xticks': (), 'yticks': ()})
# 创建一个随机的簇分配,作为参考
random_state = np.random.RandomState(seed=0)
random_clusters = random_state.randint(low=0, high=2, size=len(X))
# 绘制随机分配
axes[0].scatter(X_scaled[:, 0], X_scaled[:, 1], c=random_clusters, cmap=mglearn.cm3, s=60)
axes[0].set_title("Random assignment: {:.2f}".format(silhouette_score(X_scaled, random_clusters)))
algorithms = [KMeans(n_clusters=2), AgglomerativeClustering(n_clusters=2), DBSCAN()]
for ax, algorithm in zip(axes[1:], algorithms):
# 绘制簇分配和簇中心
clusters = algorithm.fit_predict(X_scaled)
ax.scatter(X_scaled[:, 0], X_scaled[:, 1], c=clusters, cmap=mglearn.cm3, s=60)
ax.set_title("{} - ARI: {:.2f}".format(algorithm.__class__.__name__,
silhouette_score(X_scaled, clusters)))
plt.show()k均值分数最高。对于评估聚类,稍好的策略是使用基于鲁棒性的(robustness-based)聚类指标。这种指标先向数据中添加噪声,或者使用不同的参数设定,然后运行算法,并对结果进行比较。就是说你瞎几把往里丢屎,它还行的话,那是真的牛逼!
4.3.在人脸数据集上比较算法
将使用数据的特征脸表示,它由包含100个成分的PCA(whitten=True)生成:
# 从lfw数据种提取特征脸,并对数据进行变换
from sklearn.decomposition import PCA
pca = PCA(n_components=100, whiten=True, random_state=0)
from sklearn.datasets import fetch_lfw_people
people = fetch_lfw_people(min_faces_per_person=20, resize=0.7)
image_shape = people.images[0].shape
mask = np.zeros(people.target.shape, dtype=np.bool)
for target in np.unique(people.target):
mask[np.where(people.target == target)[0][:50]] = 1
X_people = people.data[mask]
y_people = people.target[mask]
X_people = X_people / 255.
pca.fit_transform(X_people)
X_pca = pca.transform(X_people)与原始像素相比,这是对人脸图像的一种语义更强的表示,计算速度也更快。
用 DBSCAN 分析人脸数据集。
# 使用默认参数的 DBSCAN
dbscan = DBSCAN()
labels = dbscan.fit_predict(X_pca)
print("Unique labels: {}".format(np.unique(labels)))所有的数据集返回的标签都是-1,即噪声。因此考虑增大 eps 和减小 min_samples 。首先变 min_samples。
dbscan = DBSCAN(min_samples=3)
labels = dbscan.fit_predict(X_pca)
print("Unique labels: {}".format(np.unique(labels)))还是不行哦
dbscan = DBSCAN(min_samples=3, eps=15)
labels = dbscan.fit_predict(X_pca)
print("Unique labels: {}".format(np.unique(labels)))现在得到了单一簇和噪声点,接下来进一步查看
# print("Unique labels: {}
# 计算所有簇中的点数和噪声中的点数
# bincount不允许负值,所以我们需要加1
# 结果中的第一个数字对应于噪声点
print("Number of points per clusters: {}".format(np.bincount(labels + 1)))噪声点只有32个,现在查看噪声点呢
noise = X_people[labels == -1]
fig, axes = plt.subplots(3, 9, subplot_kw={'xticks': (), 'yticks': ()}, figsize=(12, 4))
for image, ax in zip(noise, axes.ravel()):
ax.imshow(image.reshape(image_shape), vmin=0, vmax=1)
plt.show()都是一些奇奇怪怪的图像呢。这种类型分析----尝试找出 “ 奇怪的那一个 ” ----被称为异常值检测(outlier detection)。we need slove problems...接下来我们看一下 eps 不同取值对应的结果呢
for eps in [1, 3, 5, 7, 9, 11, 13]:
print("\neps={}".format(eps))
dbscan = DBSCAN(eps=eps, min_samples=3)
labels = dbscan.fit_predict(X_pca)
print("Clusters present: {}".format(np.unique(labels)))
print("Clusters sizes: {}".format(np.bincount(labels + 1)))较大的簇从来都没有超过一个,这表明数据中没有两类或三类非常不同的人脸图像,每个图或多或少的都有点相似的。
dbscan = DBSCAN(min_samples=3, eps=7)
labels = dbscan.fit_predict(X_pca)
for clusters in range(max(labels) + 1):
mask = labels == clusters
n_images = np.sum(mask)
fig, axes = plt.subplots(1, n_images, figsize=(n_images * 1.5, 4),
subplot_kw={'xticks': (), 'yticks': ()})
for image, label, ax in zip(X_people[mask], y_people[mask], axes):
ax.imshow(image.reshape(image_shape), vmin=0, vmax=1)
ax.set_title(people.target_names[label].split()[-1])
plt.show()就是由很多张图,我不知道怎么显示在一张图上我就不放出来了= =在每个簇内,人脸方向和面部表情也是固定的。有些簇包含多个人的面孔,但它们的方向和表情类似。接下来使用k均值,因为DBSCAN无法创建多于一个较大的簇,凝聚聚类和K均值更可能创建均与大小的簇。
用k均值分析人脸数据
# 用k均值提取簇
km = KMeans(n_clusters=10, random_state=0)
labels_km = km.fit_predict(X_pca)
print("Clusters sizes k-means: {}".format(np.bincount(labels_km)))k均值聚类的簇更加均匀。为了可视化,由于我们是在PCA 生成的表示中进行聚类,因此我们需要使用 pca.inverse_transdorm 将簇中心旋转回到原始空间并可视化:
# 用k均值提取簇
km = KMeans(n_clusters=10, random_state=0)
labels_km = km.fit_predict(X_pca)
# print("Clusters sizes k-means: {}".format(np.bincount(labels_km)))
fig, axes = plt.subplots(2, 5, subplot_kw={'xticks': (), 'yticks': ()}, figszie=(12, 4))
for center, ax in zip(km.cluster_centers_, axes.ravel()):
ax.imshow(pca.inverse_transform(center).reshape(image_shape), vmin=0, vmax=1)
plt.show()k均值找到的簇中心是非常平滑的人脸,因为每个簇中心都是 64 到 386 张人脸的平均。使用降维的 PCA,可以增加图像的平滑度。接下来对每个簇中心我们给出簇中5张最典型和最不典型的图像。
km = KMeans(n_clusters=10, random_state=0)
labels_km = km.fit_predict(X_pca)
mglearn.plots.plot_kmeans_faces(km, pca, X_pca, X_people, y_people, people.target_names)
plt.show()因为k均值对所有的数据点进行划分,所以 “ 非典型的 ” 点与簇中心不太相似,而且它们的分配有些随意,且k均值没有噪声概念。
用凝聚聚类分析人脸数据集
from sklearn.cluster import AgglomerativeClustering
# 用ward凝聚聚类提取簇
agglomerative = AgglomerativeClustering(n_clusters=10)
labels_agg = agglomerative.fit_predict(X_pca)
print("Clusters sizes agglomerative clustering: {}".format(np.bincount(labels_agg)))相比k均值,簇分布均匀程度稍差。通过ARI度量凝聚聚类和k均值的划分是否相似
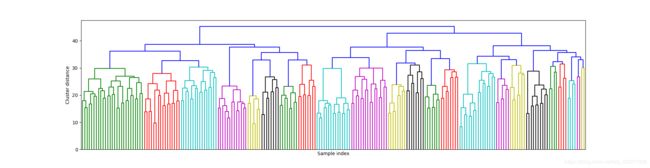
print("ARI: {:.2f}".format(adjusted_rand_score(labels_agg, labels_km)))只有0.09,说明它们的共同点很少。因为对于k均值,远离簇中心的点似乎没什么共同点。接下来画树状图并限制树的深度。
from scipy.cluster.hierarchy import dendrogram, ward
linkage_array = ward(X_pca)
# 现在我们为包含簇之间距离的linkage_array绘制树状图
plt.figure(figsize=(20, 5))
dendrogram(linkage_array, p=7, truncate_mode='level', no_labels=True)
plt.xlabel("Sample index")
plt.ylabel("Cluster distance")
plt.show()似乎没有一个特别适合的簇的数量,因为DBSCAN的结果是试图将所有的点都聚集在一起。我们将10个簇可视化,记住这玩意儿没有簇中心的概念。
n_clusters = 10
for cluster in range(n_clusters):
mask = labels_agg == cluster
fig, axes = plt.subplots(1, 10, subplot_kw={'xticks': (), 'yticks': ()}, figsize=(15, 8))
axes[0].set_ylabel(np.sum(mask))
for image, label, asdf, ax in zip(X_people[mask], y_people[mask], labels_agg[mask], axes):
ax.imshow(image.reshape(image_shape), vmin=0, vmax=1)
ax.set_title(people.target_names[label].split()[-1], fontdict={'fontsize': 9})
plt.show()图= =不放了我的图是分开的10张太多了= =
# 用ward凝聚聚类提取簇
agglomerative = AgglomerativeClustering(n_clusters=40)
labels_agg = agglomerative.fit_predict(X_pca)
print("clusters sizes agglomerative clustering: {}".format(np.bincount(labels_agg)))
n_clusters = 40
# 手动挑选“有趣的”簇
for cluster in [10, 13, 19, 22, 36]:
mask = labels_agg == cluster
fig, axes = plt.subplots(1, 15, subplot_kw={'xticks': (), 'yticks': ()}, figsize=(15, 8))
cluster_size = np.sum(mask)
axes[0].set_ylabel("#{}: {}".format(cluster, cluster_size))
for image, label, asdf, ax in zip(X_people[mask], y_people[mask], labels_agg[mask], axes):
ax.imshow(image.reshape(image_shape), vmin=0, vmax=1)
ax.set_title(people.target_names[label].split()[-1], fontdict={'fontsize': 9})
for i in range(cluster_size, 15):
axes[i].set_visible(False)
plt.show()小结
聚类的应用于评估是一个非常定性的过程,这三种聚类都可以控制聚类的粒度(granularity)。k均值可以用簇的平均值表示,被看作一种分解方法,每个数据点都有其簇中心表示。DBSCAN可以检测噪声点,允许簇具有复杂的形状,簇不是那么平均呢。凝聚聚类可以提供数据的层次结构,清晰明了。