细思极恐——R语言forestplot包画meta分析群体药动学常用森林图
细思极恐——R语言forestplot包画meta分析群体药动学常用森林图
今天,笔者想分享一下最近科研作图的经历,最主要的就是用于群体药动学模型建立的森林图,其百度百科定义为:
森林图是以统计指标和统计分析方法为基础,用数值运算结果绘制出的图型。它在平面直角坐标系中,以一条垂直的无效线(横坐标刻度为1或0)为中心,用平行于横轴的多条线段描述了每个被纳入研究的效应量和可信区间(confidence interval,CI),用一个棱形(或其它图形)描述了多个研究合并的效应量及可信区间。
这里我们尝试用Rstudio进行作图,脱离传统GraphPad, Origin等工具(Data Science No.1)
library(tidyverse)
library(forestplot)
library(readxl)
导入tidyverse框架,这里主要用dplyr, tibble这两个包,readxl用于读入Excel文件,forestplot包是集成森林图的作图包,最主要函数就是forestplot,可用以下命令查看帮助文档:
?forestplot
vignette("forestplot")
博客中使用的数据为自己的模拟数据,过程如下,实际操作过程中用NONMEN等专业软件的生成数据:
# simulation data
simulation <- tibble(rnorm(20, 8, 1), rnorm(20, 6, 2),
rnorm(20, 7, 1.5), rnorm(20, 9, 3),
rnorm(20, 0.3, 0.03), rnorm(20, 0.3, 0.1),
rnorm(20, 0.03, 0.02), rnorm(20, 0.041, 0.01),
rnorm(20, 0.15, 0.07), rnorm(20, 0.12, 0.06))
for (i in seq_along(simulation)) {
simulation[[i]]
simulation[[i]] <- abs(simulation[[i]])
print(simulation[[i]])
}
colnames(simulation) <- c("V-FFM", "V-Fatmass", "V2-FFM", "V2-Fatmass", "CL-FFM",
"CL-Fatmass", "CL2-FFM", "CL2-Fatmass", "CL3-FFM", "CL3-Fatmass")
write.csv(simulation, "222.csv")
再其下是referene data,也就是参照数据,使用如下数据进行标准化,产生森林图所需基线,我们使用的变量为药代动力学常用的两个表观分布容积Volume, 以及三个清除率Clearance
# reference data
ref <- tibble(
V = 7.769933759,
V2 = 9.583607374,
CL = 0.250863168,
CL2 = 0.041241008,
CL3 = 0.137454759
)
然后是读入数据集,通常来说直接读入格式化的实验数据即可,也可读入乱序数据用dplyr包进行清洗,这个工作比较繁琐,我后续会将规整的模拟数据集传上GITHUB供有兴趣的同学使用:
# dataset
df <- read_xlsx("simulation.xlsx")
df
其下是数据标准化,即根据上述的reference data进行归一化:
# standadization
df_CI <- tibble(mean = NA, lower = NA, upper = NA)
for (i in seq_along(df)) {
if (i <= 2) {
df_CI <- rbind(df_CI, c(t.test(df[[i]])[[5]], t.test(df[[i]])[[4]][[1]], t.test(df[[i]])[[4]][[2]]) / ref$V)
} else if (i <= 4) {
df_CI <- rbind(df_CI, c(t.test(df[[i]])[[5]], t.test(df[[i]])[[4]][[1]], t.test(df[[i]])[[4]][[2]]) / ref$V2)
} else if (i <= 6) {
df_CI <- rbind(df_CI, c(t.test(df[[i]])[[5]], t.test(df[[i]])[[4]][[1]], t.test(df[[i]])[[4]][[2]]) / ref$CL)
} else if (i <= 8) {
df_CI <- rbind(df_CI, c(t.test(df[[i]])[[5]], t.test(df[[i]])[[4]][[1]], t.test(df[[i]])[[4]][[2]]) / ref$CL2)
} else {
df_CI <- rbind(df_CI, c(t.test(df[[i]])[[5]], t.test(df[[i]])[[4]][[1]], t.test(df[[i]])[[4]][[2]]) / ref$CL3)
}
}
df_CI <- filter(df_CI, !is.na(mean))
df_CI
最后得到的效应值以及t检验置信区间如下:
# A tibble: 10 x 3
mean lower upper
1 1.04 0.971 1.11
2 0.796 0.706 0.885
3 0.685 0.616 0.754
4 0.958 0.819 1.10
5 1.18 1.13 1.23
6 1.21 1.03 1.38
7 0.880 0.671 1.09
8 0.945 0.812 1.08
9 1.04 0.849 1.22
10 0.914 0.666 1.16
上述工作全部做好则可以准备森林图绘制所需的标签值rownames,这里注意,要得到规整格式的标签,需要在留白位置设置NA,如以下代码中Effect,95% CI等标签对应的最左一列是没有输出的,则应设置NA,paste是连接字符串的函数,也可用stringr::str_c()等,数据集同理应增加一个空白行NA留给最上面的标签行。
forestplot函数中的参数重要的为labeltext, mean, lower, upper,后三项用test_data数据集的三列直接传入,test[,]选择操作也可以用dplyr::select()和dplyr::filter()完成。
grid设置80%和120%的线条
clip设置坐标轴,这里被xticks覆盖懒得改了
xticks设置x坐标轴的标度
txt_gp是个高级操作,用gpar进行赋值,其中cex为文本字体大小,ticks为坐标轴大小,xlab为坐标轴文字字体大小
graph.pos控制图形输出再第几列,比如设置4则前三列为文字,图形在最后面
fn.ci_norm设置置信区间中间图形的形状
lty.ci设置置信区间线条样式,7号是实线
lwd.ci则是置信区间线条宽度
ci.vertices.height则是置信区间两端小竖线的长度
# for row names
rownames <- cbind(c(NA , names(df)),
c("Effect", paste(trunc(df_CI$mean * 100, 2), "%", sep = "")),
c("95% CI", paste("(", trunc(df_CI$lower * 100, 2), "%-", trunc(df_CI$upper * 100, 1), "%)", sep = "")))
test_data <- rbind(rep(NA, 3), df_CI)
forestplot(rownames,
test_data[,c("mean", "lower", "upper")],
zero = 1,
grid = structure(c(0.8, 1.2), gp = gpar(col = "steelblue", lty = 2)),
clip = c(0.5, 1.25),
xticks = c(0.5, 0.8, 1, 1.2, 1.5),
txt_gp = fpTxtGp(ticks = gpar(cex = 1), xlab = gpar(cex = 1.5), cex = 1.2), # configure fontsize
boxsize = 0.3,
graph.pos = 2, # position of the plot
col=fpColors(box = "blue"),
line.margin = .1,
fn.ci_norm = fpDrawCircleCI,
lty.ci = 7, # Confidential Intervals
lwd.ci = 3,
ci.vertices.height = 0.15)
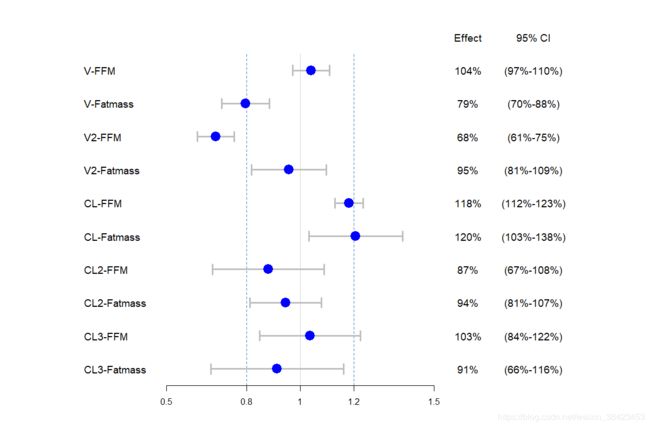
这样一个森林图就完成了。
笔者接着bb几句,自国内某211信息管理信息系统专业毕业后进入国内某顶级药学院修硕士,但做的仍然是偏数据挖掘的工作,接下来不晓得是留校直博还是出国深造,出国也不晓得是读博还是读研。
前些日子去美帝伯克利和UCSF逛了一圈,学校不错,酒吧不错,后续还是想关注澳洲,新西兰,英法德的留学,新西兰奥克兰大学Data Science博士应该是首选,但没定下心,可能最后读不了博只能读个硕镀金,大家有什么好的想法希望提供给我,不甚感激。笔者R语言开发工程师,懂Python也懂一点JAVA,C++,英语雅思6.5左右,半年后再考应该可以上7.5甚至8,希望不把自己奶死~
以上代码已上传GITHUB点此进入