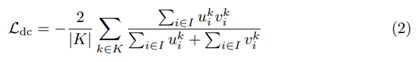nnU-Net: 基于U-Net医学图像分割技术的自适应框架
**
nnU-Net: 基于U-Net医学图像分割技术的自适应框架
**
https://arxiv.org/pdf/1809.10486.pdf
作者:Fabian Isensee
提要
U-Net于2015年推出。凭借其简单直接的架构,它迅速发展成为医学图像分割中的常用基准。 但是,U-Net缺乏对新问题的适应性,包括在精确架构,预处理,训练和推理方面等缺乏自由度。 这些选择的结果彼此影响,严重拖累整体性能。
本文介绍nnU-Net(“ no new-Net”),它是基于2D和3D U-Net的鲁棒自适应的框架。 我们提议删除许多多余的网络设计,着重于模型的性能和通用性。 我们在医学分割十项全能挑战(the Medical Segmentation Decathlon challenge)上评估nnU-Net,该挑战衡量十个学科的分割性能,这些学科包括不同的实体,图像模态,图像几何形状和数据集大小,并且不允许在数据集之间进行手动调整。
在提交结果时,nnUNet在7个一阶段比赛中,在线得分榜中获得最高的平均dice损失得分。(除了BrainTumour)
关键词:语义分割,医学图像,U-Net
1介绍
目前,医学图像分割主要由深度卷积神经网络(CNN)主导。 然而,每个细分的分割任务似乎都需要专门的网络结构和训练方法,才能获得具有竞争力的表现[1,2,3,4,5]。 这导致了大量论文其实只在较小的数据集上验证实验而没有普适性,这使得研究人员越来越难以判断一个新技术是否能在新数据集上实现其承诺优势的方法。医学分割十项全能挑战就是专门针对这个问题而产生的:比赛参与者被要求开发一个分割算法,能够普遍适用于遍布人体的十个器官的数据集上。这个算法可以动态自适应于不同的数据集上,但是要求绝对是全自动的,全过程无人工参与的。 这个挑战包括两个连续的阶段:1,开发阶段。这个阶段,开发者得到7个公开的数据集用以开发优化他们的算法,用所开发的固定的最终的算法来提交相应的7个的数据集的验证集。2,测试阶段,算法将在3个未公开的数据集上做测试。 我们认为一些看似先进的模型修改的技术,实际上会在特定的问题上发生过拟合,或者由于不那么完美的验证集,而导致非最优的解决办法。使用U-Net作为基准的模型,几个例子,在新的问题和数据集上使用时,要求做一些相应的方法上的调整。这个横跨了很多有关自由度的方面。尽管模型本身是相当简单直接的,方法也是常常被当作是一个基准,但是我们相信关于精确的网络结构,预处理,训练,和后处理的不同选择,以及这些选择之间的彼此影响,常常导致U-Net的表现低于基准。
此外,模型结构上的修改是非常容易被展示出来成是有效的,特别是当这个任务本身实际上还没有达到一个最优点。这就给模型的修改这个操作很多的空间来提升模型的表现。 在我们的先期实验中,这些模型修改实际上不能够提升模型的表现,如果模型本身已经是在一个最优的情况下。这就让我们相信,那些除了修改模型之外的操作,实际上对于模型的表现得提升,更加的重要,而这一点,常常被忽视。
在这篇论文中,我们提出一个叫做nnU-Net的模型框架,这里面包括三个主要的U-Net模型,只在原始的U-Net模型上做了一些微小的变动。我们忽略了很多新出现的技术,比如跳跃连接,密集连接,注意力机制等。nnU-Net会根据给定的图片格式,自动适应自身结构。更重要的是,这个框架全面地规定了模型使用所需要的一切步骤。这些步骤通常会影响模型的表现。比如,预处理:重采样,正则,训练:损失,优化器,数据增强。Inference:patch-based策略和ensembling,以及可能需要的后处理,比如执行成分单向连接(enforcing single connected components)
2 方法
2.1网络结构
医学图像通常包括三个维度,这就是为什么我们提出一套包括2d U-Net,3d U-Net和U-Net cascade的这么一个框架。2d和3d U-Net产生全解析度(resolution)的分割结果,而U-Net cascade首先产生一个低解析度的分割结果,然后再精修。和原始的U-Net相比我们的模型几乎没有修改,我们的主要注意力放在了如何设计一个全自动的训练流程。U-Net [6]是成功的编码器-解码器网络,近年来受到了很多关注。 其编码器部分的工作方式与传统分类CNN相似,因为它以减少的空间信息为代价连续地聚合语义信息。 由于在分割中,语义信息和空间信息对于网络的成功都是至关重要的,因此必须以某种方式恢复丢失的空间信息。 U-Net通过解码器执行此操作,解码器从“ U”的底部接收语义信息,并将其与通过跳过连接直接从编码器获得的高分辨率特征图重新组合。 与其他分割网络不同,例如FCN [9]和DeepLab的先前迭代[10],这使得U-Net可以很好地分割精细结构。和原始U-Net类似,在编码器部分,我们在池化层之间使用简单的卷积层,在解码器部分,我们使用转置卷积。与原始U-Net不同的是,我们是用的激活函数是leaky ReLU而不是ReLU(-1e-2),用instance normalization替换更加流行的batch normalization。
2D U-Net 直觉地,2d U-Net不是最完美的解决办法,因为z轴上的信息没有被利用起来。但是有证据显示,3d卷积操作又会使得模型的表现下降,如果数据集是各向异性的话。
3D U-Net 这个貌似是一个很好的解决办法,对于3d的图像数据来说。在完美的条件下,我们会选择这样的结构,来适应整个病人的数据。但是在实际的情况下,这会受到显存的限制,这使得我们只能用一个一个的包(image patches)来训练。当这个问题对于小型的数据集来说问题不大,(小是相对于每个病人的体素(voxel)的数量),比如大脑肿瘤,海马体和前列腺数据。但是对于大型的数据集比如肝,就不行了。这是因为现有结构下的视野的限制,一个小的视野不能够提供足够的信息,来将肝和其他的器官做分离。
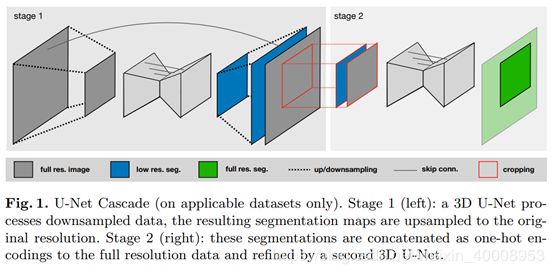
U-Net Cascade 为了解决这个3d U-Net在大数据集上的问题,我们额外地提出了一个模型,叫做cascade模型。一个3d U-Net模型首先在一个downsampled的图像上训练(stage1)这个分割的结果然后unsampled到原始的尺寸上去,然后作为额外的输入通道,以one hot encode的方式输入到第二个3d U-Net上去。第二个训练的是全解析度的。见图1
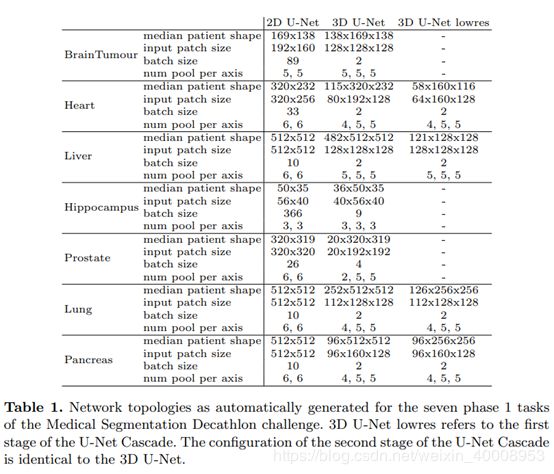
网络拓扑的动态适应 由于图像大小的巨大差异(肝脏的平均形状为482×512×512,海马的平均形状为36×50×35),输入patch的大小和每个轴的合并操作数(因此也就隐含了卷积层的数目)必须针对每个数据集自动进行调整,以充分整合空间信息。除了适应图像几何形状外,还有一些技术约束,例如要考虑的可用内存。在这方面,我们的指导原则是动态权衡batch size与网络容量之间的关系,如下所示:
我们从网络结构配置开始。对于2D U-Net,此输入patch大小为256×256,batch size为42,最高层30个特征图(每个下采样,特征图的数量加倍)。我们自动将这些参数调整为每个数据集的中值平面大小(在这里我们使用平面内间距最小的平面,对应于最高的分辨率),以便网络有效地训练整个切片。我们将网络配置为沿每个轴池化,直到该轴的要素图大小小于8(但最多不超过6个池化操作)。就像2D U-Net一样,我们的3D U-Net在最高分辨率的图层上使用30个特征图。
在这里,我们从输入切片为大小128×128×128和batch size为2的基本配置开始。由于内存的限制,我们不会将输入切片的体积增加到超过1283体素,而是匹配输入的patch size的长宽比与以像素为单位的数据集的中值大小相匹配。(Due to memory constraints, we do not increase the input patch volume beyond 1283 voxels, but instead match the aspect ratio of the input patch size to that of the median size of the dataset in voxels.)如果数据集的中位数形状小于1283,则我们将中位数形状用作输入patch size并增加批次大小(以便处理的体素总数与128×128×128相同,并且batch size为2)。就像2D U-Net一样,我们沿每个轴池化(最多5次),直到特征图的size为8。
对于任何网络,我们将每个优化程序步骤处理的体素总数(定义为输入patch size乘以batch size)限制为数据集的最大5%。对于超过5%的情况,我们将减小batch size(下限为2)。表2.1列出了为第1阶段数据集生成的所有网络拓扑。
2.2 预处理
预处理是我们的方法所组成的全自动分割流程的一部分,因此,下面介绍的步骤无需任何用户干预即可执行。裁剪所有数据都裁剪到非零值区域。这对大多数数据集(如肝脏CT)没有影响,但会减少颅骨剥脱(?)MRI的大小(从而减少计算负担)。
Resampling 重新采样CNN本身不了解体素间距。在医学图像中,不同的扫描仪或不同的采集协议通常会导致数据集具有不同的体素间距。为了使我们的网络能够正确学习空间语义,将所有患者重新采样到其各自数据集的中值体素间距,其中三阶样条插值用于图像数据,最近邻插值用于相应的分割蒙版。
是否需要经过U-Net级联模型,由以下方法确定:如果重采样数据的中值形状具有3D U-Net可以将其作为输入切片处理的体素的4倍以上(批大小为2) ,则有资格获得U-Net级联,并且还会对该数据集重新采样以降低分辨率。这是通过将体素间距(降低分辨率)增加2倍直到满足上述条件来完成的。如果数据集是各向异性的,则首先对高分辨率轴进行下采样,直到它们与低分辨率轴匹配为止,然后才同时对所有轴进行下采样。阶段1的以下数据集满足要求,因此触发了U-Net级联的使用:心脏,肝脏,肺和胰腺。
Normalization 归一化因为CT扫描的强度等级是固定的,所以所有CT图像都会根据整个数据集的统计信息自动归一化:如果数据集的相应json描文件中的模态描述表示是“ CT”,则所有在分割蒙版中位置对应出现的强度值收集训练数据集中的所有数据,并通过裁剪这些强度值的[0.5,99.5]百分位数对整个数据集进行归一化,然后根据所有收集的强度值的平均值和标准偏差对z-score进行归一化。对于MRI或其他图像模式(即,如果在模式中未找到“ CT”字符串),则将简单的z-score归一化分别应用于患者。如果裁剪使数据集中的患者平均大小(体素)减少1/4或更多,则仅在非零元素的掩码内进行归一化,并且掩码外的所有值均设置为0。
2.3 训练流程
所有模型都是从头开始训练,并在训练集上使用五重交叉验证进行评估。 我们结合dice和交叉熵损失来训练网络:
![]()
对于在几乎整个患者身上运行的3D U-Net(如果不需要cascade,则是U-Net cascade的第一阶段和3D U-Net),我们计算batch里每个样本的dice损失,并计算batch中的平均值。 对于所有其他网络,我们将batch中的样本(samples)解释为伪体积(volume),并计算批次中所有体素的dice损失。
这里使用的dice损失公式是[14]中提出的多类别变体的改编。 根据过去的经验[13,1],我们倾向于采用这种形式,而不是其他变体[8,15]。 dice损失的实现如下:
其中u是网络的softmax输出,v是标签分割图的一个热编码。 u和v都具有形状I×K,其中i∈I是训练patch/batch的像素数,而k∈K是类别。对于所有实验,我们使用Adam优化器,其初始学习率为3×10−4。 我们将一个时期定义为250个训练批次的迭代。 在训练过程中,我们保留验证损失(l v MA)和训练损失(l t MA)的指数移动平均值。 只要在过去30个时期内lt MA至少没有提高5×10-3,学习率就会降低5倍。如果lv MA在30 ep内没有提高5×10-3,则训练会自动终止。 持续60个时期,但学习率小于10−6之前不会停止。
数据增强
从有限的训练数据训练大型神经网络时,必须格外小心,以防止过拟合。我们通过利用各种数据增强技术来解决此问题。在训练过程中,以下增强技术在飞行中被应用:随机旋转,随机缩放,随机弹性变形,伽玛校正增强和镜像。数据扩充是通过我们自己的内部框架完成的,该框架可从www.github.com/MIC-DKFZ/batchgenerators公开获得。我们分别为2D和3D U-Net定义了一组数据增强参数。这些参数不会在数据集之间修改。如果3D U-Net的输入面片大小的最大边缘长度大于最短长度的两倍,则应用三维数据增强可能不是最佳选择。对于适用此标准的数据集,我们改用2D数据增强,然后逐个切片地将其应用于每个样本。U-Net级联的第二阶段接收上一步的分段作为附加输入通道。为了防止强烈的共适应(co-adaptation),我们应用了随机的形态运算符(腐蚀,扩张,打开,关闭),并随机删除了这些分段的连接部分。
Patch Sampling
为了提高网络培训的稳定性,我们强制要求取样中的三分之一以上的样本包含至少一个随机选择的前景类。
2.4 Inference
由于我们训练的是基于patch,所有测试也都基于patch进行。 由于网络精度朝着patch的边界降低,因此,当汇总patch的预测时,我们将靠近中心的体素的权重设置为高于接近边界的体素。 patch选择为按patch大小/ 2重叠,所有patch通过沿所有轴做镜像来进一步利用测试时间做数据增强。将分块的预测和测试时间数据的增强相结合,可以进行细分,在每个细分中,通过汇总多达64个预测(在使用3D U-Net的中心),可以获得每个体素的决策。 对于测试案例,我们使用从训练集交叉验证中获得的五个网络作为整体,以进一步提高模型的鲁棒性。
2.5 后处理
对训练数据进行所有地面真相分段标签的关联组件分析。 如果某个类在所有情况下都位于单个连接的组件内,则这个类将被解释为是数据集的常规属性。 因此,将在相应数据集的预测图像上自动删除除了最大组件以外的所有该类组件。
2.6组装与提交
为了进一步提高分割性能和鲁棒性,我们为每个数据集整合了三个模型中两个模型的所有可能组合。 对于最终提交,将自动选择在训练集交叉验证中达到最高平均前景dice得分的模型。
3 实验和结果
我们对阶段1数据集使用五重交叉验证来优化我们的网络拓扑。 表2中汇总了我们的第一阶段交叉验证结果以及相应的测试集结果。-表明U-Net Cascade不适用于数据集(根据我们的标准),因为它已经完全可用 由3D U-Net的输入patch大小覆盖。 用于最终提交的模型以粗体突出显示。 尽管平台允许提交多个测试集,但我们认为这样做是不好的做法。 因此,我们只提交了一次,并报告了该次提交的结果。
如表2所示,我们的阶段1交叉验证结果在测试集上得到了恢复,表明不存在过度拟合的问题。遭受性能下降的唯一数据集是BrainTumour。 此阶段1数据集的数据源于BRATS挑战[16],在BRATS挑战中,验证和测试之间的这种性能下降是常见现象,并归因于相应数据分布有较大差异。
4 讨论
在本文中,我们提出了医学领域的nnU-Net分割框架,该框架直接围绕原始U-Net架构[6]构建,并动态地适应任何给定数据集。 基于我们的假设,即非架构修改可能比最近提出的某些架构修改强大得多,该框架的本质是对自适应预处理,训练方案和测试的全面设计。适应新任务所需的所有设计选择均以全自动方式完成,无需人工干预。 对于每个任务,nnU-Net会针对三个不同的自动配置的U-Net模型自动运行五重交叉验证,并且选择具有最高平均前景dice得分的模型(或整体)进行最终提交。 在“医学十项全能”比赛上,我们证明nnU-Net在7个高度不同的医学数据集的保留测试集上具有竞争力,在所有任务的所有类别(BrainTumour除外)上均获得最高的平均dice得分。
我们承认,训练三个模型并为每个数据集独立选择最佳模型并不是最干净的解决方案。 未来,可在训练之前研究适当的启发式方法,以找到给定数据集的最佳模型。 我们当前的趋势倾向于U-Net Cascade(如果无法应用Cascade,则倾向于3D U-Net),唯一(接近)的例外是前列腺和肝脏任务。 此外,我们在许多设计选择中的额外好处(例如,使用Leaky ReLU代替常规ReLU)以及在此挑战的背景下未正确验证我们的数据扩充的参数。 因此,未来的工作将集中在通过消融研究来系统地评估所有设计选择。