分布式空间分析引擎-Simba架构分析与源码阅读之SpatialJoin实现与总结
在分区器和索引部分铺垫了很多,其实Simba中Spatial join算子的部分是真正利用前面的结构来有效降低计算量的逻辑,也是simba最大的亮点。simba主要实现了三类spatial join算子:
KNN query:select * from table IN KNN ($target) within ($k);
Distance join:SELECT * FROM R JOIN S ON (R.x - S.x) * (R.x - S.x) + (R.y - S.y) * (R.y - S.y)
<= 10.0 * 10.0
KNN join:SELECT * FROM point1 AS p1 KNN JOIN point2 AS p2 ON POINT(p2.x, p2.y) IN KNN(POINT(p1.x, p1.y), 10).
出于篇幅原因,本章会对每类算子选择一个算子具体讲述代码和算法原理,对于其余算子仅会分析代码实现,具体的算法原理可以参考simba的论文。在本章的介绍中,会把参数join的两部分数据分别用leftRDD和rightRDD表示。
KNN Query
Simba基于two-level index对KNN Query进行了特别的优化,值得注意的是,不同的索引对于KNN Query的优化方式有所不同,作者所实现的效果最好的KNN算子需要配合提前建立RTree索引。另外KNN query算子并没有作为单独一个类出现,KNN query部分代码放在了org.apache.spark.sql.simba.index.IndexedRelationScan类的doExceute()函数的RTreeIndexedRelation优化逻辑的部分,在由physicalPlan生成RDD的时候执行。KNN Query算子的主要逻辑如下:
1)首先会根据global index未给定点找到最近的几个partition,然后调用knnGlobalPrune方法粗略找到第一个KNN候选集。在knnGlobalPrune方法中会首先调用PartitionPruningRDD,基于给定的paritionID裁剪不满足要求的partition;然后在裁剪后的每个分区内部调用local inedx的KNN方法找到最近的k个点,再把每个裁剪分区的k个点进行归并,去距离最近的k个,生成候选集tmp_ans,并计算候选集与点之间的距离theta作为第二次的裁剪;
def knnGlobalPrune(global_part: Set[Int]): Array[InternalRow] = {
val pruned = new PartitionPruningRDD(rtree._indexedRDD, global_part.contains)
pruned.flatMap{ packed =>
var tmp_ans = Array[(Shape, Int)]()
if (packed.index.asInstanceOf[RTree] != null) {
tmp_ans = packed.index.asInstanceOf[RTree]
.kNN(query_point, k, keepSame = false)
}
tmp_ans.map(x => packed.data(x._2))
}.takeOrdered(k)(ord)
}
// first prune, get k partitions, but partitions may not be final partitions
val global_part1 = rtree.global_rtree.kNN(query_point, k, keepSame = false).map(_._2).toSet
val tmp_ans = knnGlobalPrune(global_part1) // to get a safe and tighter bound
val theta = evalDist(tmp_ans.last, query_point, column_keys, rtree.isPoint)采用上面这样的逻辑的原因是:首先选择与目标点最近的、含有至少k个点的partition,以这些parition到目标点的最远距离theta作为裁剪依据。到目标点的距离大于theta的分区内的点一定不属于到目标点最近的k个点。
上图中每个矩形代表一个数据分区,步骤1)就是找到深灰色的部分,并计算红色圆形的半径,深灰色内至少含有k点,所以到目标点最近的k个点一定全部位于红色圆形内的部分,也就一定位于浅灰色的分区内,步骤2)就是找到浅灰色分区的部分。
2)计算到半径theta之后,simba会调用global index的circleRange方法找到半径为theta圆相交的partitionID,进行裁剪得到第二个候选集tmp_knn_res;第3行部分的逻辑可以看做一个懒加载,如果两部分重合就不再计算,不重合说明数据可能位于红色圈内、深灰色矩形之外,需要继续计算。
val global_part2 = rtree.global_rtree.circleRange(query_point, theta).
map(_._2).toSet -- global_part1
val tmp_knn_res = if (global_part2.isEmpty) tmp_ans
else knnGlobalPrune(global_part2).union(tmp_ans).sorted(ord).take(k)3)在裁剪失败(候选集为空)或者上一步的结果大于给定阈值的场景下,会尝试利用global index的range方法计算所有与给定点的MBR相交的部分进行计算。
var global_part = rtree.global_rtree.range(queryMBR).map(_._2).toSeq
if (cir_ranges.nonEmpty){ // circle range
global_part = global_part.intersect(
rtree.global_rtree.circleRangeConj(cir_ranges).map(_._2)
)
}
val pruned = new PartitionPruningRDD(rtree._indexedRDD, global_part.contains)Distance Join
Distance Join部分代码位于org.apache.spark.sql.simba.execution.join包中,实现了DJSpark、CDJSpark、RDJSpark、BDJSpark四类Distance Join算子。
DJSpark
DJSpark的思路与SpatialHadoop的思路一致:1)分别对参与join的leftRDD和rightRDD按key值进行分桶;2)对各个分桶内的数据做两两组合,每个分桶的数据与其他分桶数据组合做nested loop join;3)数据合并。对应地simba中的算法实现如下:
1)分别把leftRDD和rightRDD进行STR分区(对应于hadoop的分桶),并对rightRDD建立RTree索引:
val (left_partitioned, left_mbr_bound) = STRPartition(left_rdd, dimension, num_partitions,
sample_rate, transfer_threshold, max_entries_per_node)
val (right_partitioned, right_mbr_bound) = STRPartition(right_rdd, dimension, num_partitions,
sample_rate, transfer_threshold, max_entries_per_node)
val right_rt = RTree(right_mbr_bound.zip(Array.fill[Int](right_mbr_bound.length)(0))
.map(x => (x._1._1, x._1._2, x._2)), max_entries_per_node)2)对与leftRDD的每个数据分区,根据RTree索引找到rightRDD中所有距离小于给定值的分区,并对这些分区对进行编号(tot):
left_mbr_bound.foreach { now =>
val res = right_rt.circleRange(now._1, r)
val tmp_arr = mutable.ArrayBuffer[Int]()
res.foreach {x =>
if (right_dup(x._2) == null) right_dup(x._2) = Array(tot)
else right_dup(x._2) = right_dup(x._2) :+ tot
tmp_arr += tot
tot += 1
}
left_dup(now._2) = tmp_arr.toArray
}3)对leftRDD和rightRDD的数据分区按归属于哪些分区对进行重分区,依据步骤2)生成的分区对编号生成左右两个新分区left_dup_partitioned和right_dup_partitioned,并执行zipPartitions算子,对相同编号的两个分区进行聚合:
val left_dup_rdd = left_partitioned.mapPartitionsWithIndex { (id, iter) =>
iter.flatMap {now =>
val tmp_list = bc_left_dup.value(id)
if (tmp_list != null) tmp_list.map(x => (x, now))
else Array[(Int, (Point, InternalRow))]()
}
}
val right_dup_rdd = right_partitioned.mapPartitionsWithIndex { (id, iter) =>
iter.flatMap {now =>
val tmp_list = bc_right_dup.value(id)
if (tmp_list != null) tmp_list.map(x => (x, now))
else Array[(Int, (Point, InternalRow))]()
}
}
val left_dup_partitioned = MapDPartition(left_dup_rdd, tot).map(_._2)
val right_dup_partitioned = MapDPartition(right_dup_rdd, tot).map(_._2)
left_dup_partitioned.zipPartitions(right_dup_partitioned) {(leftIter, rightIter) =>
val ans = mutable.ListBuffer[InternalRow]()
val right_data = rightIter.toArray
if (right_data.nonEmpty) {
val right_index = RTree(right_data.map(_._1).zipWithIndex, max_entries_per_node)
leftIter.foreach {now =>
ans ++= right_index.circleRange(now._1, r)
.map(x => new JoinedRow(now._2, right_data(x._2)._2))
}
}
ans.iterator
}
CDJSpark
CDJSpark是一种基于笛卡尔积的Distance Join算法,实现思路非常简单:
left.execute().cartesian(right.execute()).mapPartitions { iter =>
val joinedRow = new JoinedRow
iter.filter { row =>
val point1 = ShapeUtils.getShape(left_key, left.output, row._1).asInstanceOf[Point]
val point2 = ShapeUtils.getShape(right_key, right.output, row._2).asInstanceOf[Point]
point1.minDist(point2) <= r
}.map(row => joinedRow(row._1, row._2))
}先进行笛卡尔积,然后根据是否满足DIstance条件对结果filter。
RDJSpark
RDJSpark是simba的基于two-level index实现的Distance Join方法,实现思路如下:
1)将leftRDD基于STR分区器进行分区,并在划分出的MBR块上建立RTree索引:
val (left_partitioned, left_mbr_bound) =
STRPartition(left_rdd, dimension, num_partitions, sample_rate,
transfer_threshold, max_entries_per_node)
val left_part_size = left_partitioned.mapPartitions {
iter => Array(iter.length).iterator
}.collect()
val left_rt = RTree(left_mbr_bound.zip(left_part_size).map(x => (x._1._1, x._1._2, x._2)),
max_entries_per_node)2)将建立的global index广播出去:
val bc_rt = sparkContext.broadcast(left_rt)
3)leftRDD的通过索引为rightRDD每个数据点找到距离满足条件的候选MBR,生成right_dup=>(left partitionID, candidate point);
val right_dup = right_rdd.flatMap {x =>
bc_rt.value.circleRange(x._1, r).map(now => (now._2, x))
}4)leftRDD与rightRDD执行zippartition算子,在同一个数据分区内的数据,通过local index进行distance join。
BDJSpark
BDJSpark的思路与DJSpark的思路基本一致,可以看做是DJSpark的弱化版,因为在BDJSpark中仅仅将数据分桶两两组合,但并没有建立Rtree索引进行加速。代码逻辑如下:
1)数据分桶(分区),并将分桶两两组合:
val tot_rdd = left.execute().map((0, _)).union(right.execute().map((1, _)))
val tot_dup_rdd = tot_rdd.flatMap {x =>
val rand_no = new Random().nextInt(num_partitions)
var ans = mutable.ListBuffer[(Int, (Int, InternalRow))]()
if (x._1 == 0) {
val base = rand_no * num_partitions
for (i <- 0 until num_partitions)
ans += ((base + i, x))
} else {
for (i <- 0 until num_partitions)
ans += ((i * num_partitions + rand_no, x))
}
ans
}
val tot_dup_partitioned = MapDPartition(tot_dup_rdd, num_partitions * num_partitions)2)每个分桶对进行nested loop join:
left_data.foreach {left =>
right_data.foreach {right =>
if (left._1.minDist(right._1) <= r) {
joined_ans += new JoinedRow(left._2, right._2)
}
}
}相比于CDJSpark仅仅是做了下分区,没有做笛卡尔积。
KNN Join
KNN Join的代码位于org.apache.spark.sql.simba.execution.join包中,实现了CKJSpark、RKJSpark、BKJSpark、VKJSpark、ZKJSpark五类KNN Join算子。
BKJSpark
BKJSpark的思路是将数据进行分桶,然后将分桶两两组合进行数据分区内的过滤计算。代码逻辑如下:
1)数据分桶(分区),并将分桶两两组合:
val tot_rdd = left.execute().map((0, _)).union(right.execute().map((1, _)))
val tot_dup_rdd = tot_rdd.flatMap {x =>
val rand_no = new Random().nextInt(num_partitions)
val ans = mutable.ListBuffer[(Int, (Int, InternalRow))]()
if (x._1 == 0) {
val base = rand_no * num_partitions
for (i <- 0 until num_partitions)
ans += ((base + i, x))
} else {
for (i <- 0 until num_partitions)
ans += ((i * num_partitions + rand_no, x))
}
ans
}
val tot_dup_partitioned = MapDPartition(tot_dup_rdd, num_partitions * num_partitions)2)对每个分桶对内的数据两两组合,计算KNN,并通过reduceByKey算子聚合KNN结果:
left_data.foreach(left => {
var pq = new BoundedPriorityQueue[(InternalRow, Double)](k)(new DisOrdering)
right_data.foreach(right => pq += ((right._2, right._1.minDist(left._1))))
joined_ans += ((left._2, pq.toArray))
})
joined_ans.iterator
}.reduceByKey((left, right) => (left ++ right).sortWith(_._2 < _._2).take(k), num_partitions)
.flatMap {
now => now._2.map(x => new JoinedRow(now._1, x._1))
}CKJSpark
CKJSpark的思路是对leftRDD与rightRDD进行笛卡尔积来计算KNN结果:1)leftRDD与rightRDD计算笛卡尔积;2)笛卡尔积的结果计算每对left tuple与right tuple的距离;3)执行reduceByKey对于leftRDD的每个key找到距离最小的k个right key。
override protected def doExecute(): RDD[InternalRow] = {
val left_rdd = left.execute()
val right_rdd = right.execute()
left_rdd.map(row =>
(ShapeUtils.getShape(left_key, left.output, row).asInstanceOf[Point], row)
).cartesian(right_rdd).map {
case (l: (Point, InternalRow), r: InternalRow) =>
val tmp_point = ShapeUtils.getShape(right_key, right.output, r).asInstanceOf[Point]
l._2 -> List((tmp_point.minDist(l._1), r))
}.reduceByKey {
case (l_list: Seq[(Double, InternalRow)], r_list: Seq[(Double, InternalRow)]) =>
(l_list ++ r_list).sortWith(_._1 < _._1).take(k)
}.flatMapValues(list => list).mapPartitions { iter =>
val joinedRow = new JoinedRow
iter.map(r => joinedRow(r._1, r._2._2))
}
}RKJSpark
RKJSpark是基于simba的two-level index实现的KNN join算子,他的主要思路如下:
1)对leftRDD基于STR分区器进行分区,对rightRDD进行抽样并建立RTree索引:
val right_sampled = right_rdd
.sample(withReplacement = false, sample_rate, System.currentTimeMillis())
.map(_._1).collect().zipWithIndex
val right_rt = RTree(right_sampled, max_entries_per_node)
val dimension = right_sampled.head._1.coord.length
val (left_partitioned, left_mbr_bound) =
STRPartition(left_rdd, dimension, num_partitions, sample_rate,
transfer_threshold, max_entries_per_node)2)在已经被分区的leftRDD的每个分区内部基于STR算法重新划分MBR,这是为了缩小MBR的划分粒度,达到更好的优化效果。
val refined_mbr_bound = left_partitioned.mapPartitionsWithIndex {(id, iter) =>
if (iter.hasNext) {
val data = iter.map(_._1).toArray
def recursiveGroupPoint(entries: Array[Point], cur_dim: Int, until_dim: Int)
: Array[(Point, Double)] = {
val len = entries.length.toDouble
val grouped = entries.sortWith(_.coord(cur_dim) < _.coord(cur_dim))
.grouped(Math.ceil(len / dim(cur_dim)).toInt).toArray
if (cur_dim < until_dim) grouped.flatMap(now => recursiveGroupPoint(now, cur_dim + 1, until_dim))
else grouped.map {list =>
val min = new Array[Double](dimension).map(x => Double.MaxValue)
val max = new Array[Double](dimension).map(x => Double.MinValue)
list.foreach { now =>
for (i <- min.indices) min(i) = Math.min(min(i), now.coord(i))
for (i <- max.indices) max(i) = Math.max(max(i), now.coord(i))
}
val mbr = MBR(new Point(min), new Point(max))
var cur_max = 0.0
list.foreach(now => {
val cur_dis = mbr.centroid.minDist(now)
if (cur_dis > cur_max) cur_max = cur_dis
})
(mbr.centroid, cur_max)
}
}
recursiveGroupPoint(data, 0, dimension - 1).map(x => (x._1, x._2, id)).iterator
} else Array().iterator
}.collect()3)为步骤2)中生成的每个mbr依据RTree索引计算过滤阈值,过滤阈值的计算方法涉及复杂证明,请参照原论文:
val theta = new Array[Double](refined_mbr_bound.length)
for (i <- refined_mbr_bound.indices) {
val query = refined_mbr_bound(i)._1
val knn_mbr_ans = right_rt.kNN(query, k, keepSame = false)
theta(i) = knn_mbr_ans.last._1.minDist(query) + (refined_mbr_bound(i)._2 * 2.0)
}4)rightRDD过滤在任一mbr的theta阈值范围内的数据并重分区,使分区id与leftRDD一致:
val right_dup = right_rdd.flatMap(x => {
var list = mutable.ListBuffer[(Int, (Point, InternalRow))]()
val set = new mutable.HashSet[Int]()
for (i <- refined_mbr_bound.indices) {
val pid = refined_mbr_bound(i)._3
if (!set.contains(pid) && refined_mbr_bound(i)._1.minDist(x._1) < bc_theta.value(i)) {
list += ((pid, x))
set += pid
}
}
list
})
val right_dup_partitioned = MapDPartition(right_dup, left_mbr_bound.length).map(_._2)5)对leftRDD与过滤后的rightRDD执行zipPartition算子,在每个分区内部基于RTree索引进行KNN运算,汇总得到最终计算结果:
left_partitioned.zipPartitions(right_dup_partitioned) {
(leftIter, rightIter) =>
val ans = mutable.ListBuffer[InternalRow]()
val right_data = rightIter.toArray
if (right_data.length > 0) {
val right_index = RTree(right_data.map(_._1).zipWithIndex, max_entries_per_node)
leftIter.foreach(now =>
ans ++= right_index.kNN(now._1, k, keepSame = false)
.map(x => new JoinedRow(now._2, right_data(x._2)._2))
)
}
ans.iterator
}VKJSpark
VKJSpark的思路与RKJSpark有类似之处:首先对数据分区,然后为leftRDD的每个分区,根据rightRDD的分区与该分区的最小距离是否大于本分区内找到的KNN阈值找到匹配的数据分区集合,然后对leftRDD的每个数据分区与对应找到的分区集合计算结果并汇总。其代码逻辑如下:
1)对数据进行分区,Voronoi diagram是地理领域一个非常经典的空间划分方法:
val pivots = generatePivots(left_rdd.map(_._1).union(right_rdd.map(_._1)), num_of_pivots)
val bc_pivots = sparkContext.broadcast(pivots)
val left_with_pivots = left_rdd.mapPartitions(iter => iter.map(x => {
var nearestDist = Double.MaxValue
var ans = -1
val point = x._1
val local_pivots = bc_pivots.value
for (i <- local_pivots.indices){
val dist = point.minDist(local_pivots(i))
if (dist < nearestDist) {
nearestDist = dist
ans = i
}
}
(ans, x)
}))
val right_with_pivots = right_rdd.mapPartitions(iter => iter.map(x => {
var nearestDist = Double.MaxValue
var ans = -1
val point = x._1
val local_pivots = bc_pivots.value
for (i <- local_pivots.indices){
val dist = point.minDist(local_pivots(i))
if (dist < nearestDist) {
nearestDist = dist
ans = i
}
}
(ans, x)
}))
// calculate the number of records in every partition of the left table
val cell_size = left_with_pivots.aggregate(Array.fill[Int](num_of_pivots)(0))((tmp, now) => {
tmp(now._1) += 1
tmp
}, (left, right) => {
left.zip(right).map(x => x._1 + x._2)
})
val (grouping, pivot_to_group) = geoGrouping(pivots, cell_size, num_partitions)
val left_partitioned = VoronoiPartition(left_with_pivots, pivot_to_group, num_partitions)
val right_partitioned = VoronoiPartition(right_with_pivots, pivot_to_group, num_partitions) 首先生成给定数目的轴值:先随机生成第一个轴值,然后每次选取与选定轴值较远的点作为下一个轴值,直至得到所有的轴值。然后分别对leftRDD与rightRDD根据与各个轴值的远近划定分区。
2)然后为leftRDD的每个分区,根据rightRDD的分区与该分区的最小距离是否大于本分区内找到的KNN阈值找到匹配的数据分区集合。由于leftRDD与rightRDD由相同轴值、相同pivot_to_group数组进行分区,所以每个分区的数据具有相同的数据域和分区id。
val right_dup = right_partitioned.mapPartitions{iter => {
var ans = mutable.ListBuffer[(Int, ((Point, Int), InternalRow))]()
while (iter.hasNext) {
val now = iter.next()
for (left <- 0 until num_partitions)
if (now._2._1.minDist(bc_pivots.value(now._1)) >= lower_bounds(left)(now._1)) {
ans += ((left, ((now._2._1, now._1), now._2._2)))
}
}
ans.toArray.iterator
}}3)对leftRDD与rightRDD执行zipPartition算子,在同分区内进行计算,并汇总结果。
总结来说,个人认为VKJSpark相对于RKJSpark的劣势有两点:
轴值与分区数的选定存在很大随机性,且很大影响算法性能;
基于数据分区整体距离的分区过滤方法与RKJSpark相比存在劣势,且需要进行比较多的预计算。
ZKJSpark
ZKJSpark是一个非常有趣的KNN Join算法,最终是由李飞飞等人在2012年提出,发表在论文《Efficient Parallel kNN Joins for Large Data in MapReduce》中,simba中基于Spark实现了这个算法。之前介绍的几种算法面向的场景是准确计算KNN的结果,而ZKJSpark面向的场景则是快速给出KNN的一个估计结果,所给出的结果与真实结果差异较小。
ZKJSpark算法的主要思路是:1)将高维数据通过ZValue转化为一维数据;2)将一维数据排序分组,对于leftRDD每个分组的数据,根据ZValue结果二分查找到KNN范围对应的rightRDD分组集合;3)将每个leftRDD分区与找到的rightRDD分区集合的数据组合计算,并汇总KNN结果;4)将原始的leftRDD与rightRDD进行多次移位,防止ZValue的突变破坏数据聚集性,并将多次移位的结果汇总。下面结合代码详细介绍这种算法的逻辑:
1)首先生成一种随机移位矩阵,移位矩阵的维度为设定的移位次数参数;
private def genRandomShiftVectors(dimension : Int, shift : Int): Array[Array[Int]] = {
val r = new Random(System.currentTimeMillis)
val ans = Array.ofDim[Int](shift + 1, dimension)
for (i <- 0 to shift)
for (j <- 0 until dimension) {
if (i == 0) ans(i)(j) = 0
else ans(i)(j) = Math.abs(r.nextInt(100))
}
ans
}2)对数据进行移位,并对移位的数按上述算法逻辑计算KNN结果:
var joined_rdd = zKNNPerIter(left_rdd, right_rdd, k, shift_vec(0))
for (i <- 1 to num_shifts)
joined_rdd = joined_rdd.union(zKNNPerIter(left_rdd, right_rdd, k, shift_vec(i)))3)对多次移位得到的数据调用reduceByKey算子,去重以及取距离最近的前k个得到最终结果。
joined_rdd.reduceByKey((left, right) =>
(left ++ right).distinct.sortWith(_._2 < _._2).take(k), num_partition).flatMap(now => {
val ans = mutable.ListBuffer[InternalRow]()
now._2.foreach(x => ans += new JoinedRow(now._1, x._1))
ans
})接口层设计
Simba的接口层对Spark sql的SparkSession 和sessionState类进行了封装,所以在API层面能够完全复用spark DataSet的原生SQL查询的API。除此之外,simba还基于SQLDataSet重写了自己的DataSet类,使simba通过DataSet类调用spatial join算子。所以simba支持sql两种调用方式:
1)封装SQLDataSet而带来的Spark SQL原生的sql调用方式:
val res = simba.sql("SELECT * FROM b")
2)通过重写DataSet类而为spatial类算子增加的DataSet API类调用方式:
import simbaSession.implicits._
import simbaSession.simbaImplicits._
val ps = (0 until 10000).map(x => PointData(Point(Array(x.toDouble, x.toDouble)), x + 1)).toDS
ps.knn("p", Array(1.0, 1.0), 4).show()分析与总结
Simba的内容大体介绍完了,可以看出simba的几乎所有优化,包括分区器、索引、谓词下推等等,都是面向spatial join服务的,所以这里重点针对spatial join做个总结。
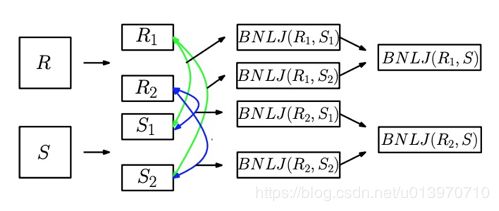
图4 spatial join类算法的分桶、组合计算、归并结合的通用做法示意
总结来说,simba在spatial operator的解决思路既与其他空间计算引擎有类似之处:对参与运算数据分区(分桶),将数据分区进行组合计算,合并结果。也通过自己的two-level机制,通过索引或者ZValue排序去除了一部分无需参与运算的分区组合(对比于基础方案直接将分区两两组合,对所有的分区组合计算),从而进一步减少了计算量。这种思想是值得借鉴的。
个人觉得Simba也有一些可能需要改进的地方:
1)有很多需要调节的参数,这些参数会极大地影响性能,然而又需要根据具体数据集仔细设定,比如:生成分区数,构建索引时的取样率,树索引构建时的单节点容量、分区器单分组内的最大数据量等。
2)simba在生成分区,构建索引的时候会做随机抽样,抽取数据的均匀性会影响索引的构建质量、分区的划分合理性等等。这就给结果带来了一定的随机性,可能多次查询之间的性能差异很大。
在设定了合适的参数,随机抽样的结果能很好地反应数据整体分布的情况下,理论上数据分布的越均匀,simba相对于常规算法(数据分桶,组合运算)的优势会越明显;数据热点问题越严重,simba的优势会越小一些。