R语言笔记1:数据类型(向量、数组、矩阵、 列表和数据框)
宏基因组按:科研中数据分析解读占用了我们太多时间,学习R语言是生物测序领域数据(reads count表)分析及可视化的首选。举个例子,扩增子分析从fastq到OTU表至多是de novo或reference两种套路(1-3天)。而对OTU表开始的组间比较、网络分析、机器学习等会有上百种方法和展示方式,每一篇优秀的文章,都是数据反复咀嚼上百次优化出来的结果(3个月-3年),而这一漫长的科研之路有R语言技能的相伴,可将统计分析可视化操作一网打尽,定能助你事半功倍。
前期公众号己分享了扩增子、宏基因组分析流程及可视化文章上百篇,但一直缺少基础入门的知识。今天起分享一位从18年3月1号刚要从wet转dry的学生零基础学习笔记,供初学者学习,虽然笔记会有不系统的地方,但也正是初学者需要经历和面对的,希望想入行的快上车,共同学习,一起成长。
学习R语言,需要先安装R语言,只需要从 https://www.r-project.org/ 下载适合你系统的最新版本软件安装即可。R语言有个优秀的环境叫Rstudio,具体安装可参考 《R语言学习 - 入门环境Rstudio》一文。
R语言中的数据类型(Data Types)
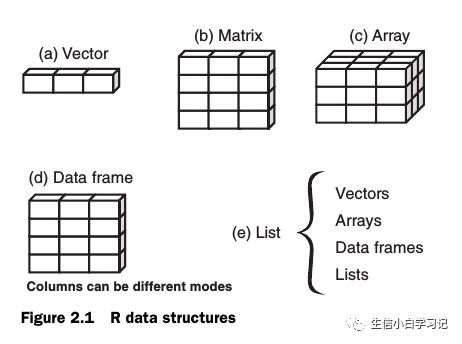
R语言的对象(Objects)主要包括向量、矩阵、数组、数据框和列表。
R语言的对象有五种最基本的类型,即,字符型(character)、数值型(numeric,包括小数)、整型(integer)、复数型(complex)以及逻辑型(logical,TRUE/FALSE)
属性是R语言对象的一部分。主要包括以下几种:名字(names,dimnames),维度(dimensions,包括矩阵等),类别(class,包括数字、整数等),长度(length),以及其他。可通过 attributes()函数查看对象的属性,不是所有对象都有属性,如果没有则返回NULL。
1. 向量
向量(vector)是R语言中最基本的数据类型,执行组合功能的函数 c()可用来创建向量。
各类向量如下例所示:
a <- c (1, 2, 7, -4, 5) ## numeric
b <- c ("Rice", "Wheat") ## character
c <- c (TRUE, TRUE, FALSE, TRUE) ## logical
d <- c (1+0i, 2+4i) ## complex
e <- c (9:17) ## integer
注意:单个向量中的数据必须拥有相同的类型(数值型、字符型或逻辑型)。
创建空向量可以使用 vector()函数。例如创建一个指定长度为10、类型为数值型的空向量:
> x <- vector("numeric", length = 10)
> x
[1] 0 0 0 0 0 0 0 0 0 0
另外,标量是只含一个元素的向量,它们用于保存常量。例如
f <- 3
g <- "US"
h <- TRUE
2. 矩阵
矩阵(matrice)是具有维度属性的向量,矩阵都是二维的,和向量类似,矩阵中也仅能包含一种数据类型。
主要有三种创建矩阵的方法:
(1)直接创建
例:数字1-20自动创建为一个5行4列的矩阵,自动填充第一列之后开始填充第二列
y <- matrix(1:20, nrow = 5, ncol = 4)
> y
[,1] [,2] [,3] [,4]
[1,] 1 6 11 16
[2,] 2 7 12 17
[3,] 3 8 13 18
[4,] 4 9 14 19
[5,] 5 10 15 20
> dim(y)
[1] 5 4 ##dim()看维度,5行4列
(2)矢量+维度向量
添加维度向量 dim()是将矢量转变为矩阵的方法
> m <- c(1:10)
> m
[1] 1 2 3 4 5 6 7 8 9 10
> dim(m) <- c(2,5) ##2行5列
> m
[,1] [,2] [,3] [,4] [,5]
[1,] 1 3 5 7 9
[2,] 2 4 6 8 10
(3)绑定行或列来创建矩阵
绑定行或绑定列可以通过 cbind()和 rbind()来实现
> x <- 1:3
> y <- 10:12
> cbind (x, y)
x y
[1,] 1 10
[2,] 2 11
[3,] 3 12
> rbind (x, y)
[,1] [,2] [,3]
x 1 2 3
y 10 11 12
3. 数组
数组(array)与矩阵类似,但是维度可以大于2。数组可通过array函数创建。
4. 列表
列表(list)是一种可包含多种不同类型对象的向量,是一些对象(或成分,component)的有序集合。
> x <- list(1, "a", TRUE, 1 + 4i)
> x
[[1]]
[1] 1
[[2]]
[1] "a"
[[3]]
[1] TRUE
[[4]]
[1] 1+4i
5.数据框
数据框(Data Frames)是一种特殊的列表,其中所用元素长度都相等,列表中的每个元素都可以看作一列,每个元素的长度可以看作行数。
创建显式数据框的方法是 data.frame()
> ID <- c(1,2,3,4)
> age <- c(25,26,55,43)
> diabetes <- c("Type1","Type2","Type3","Type1")
> status <- c("Poor", "Improved", "Excellent","Poor")
> data <- data.frame(ID, age, diabetes, status)
> data
ID age diabetes status
1 1 25 Type1 Poor
2 2 26 Type2 Improved
3 3 55 Type3 Excellent
4 4 43 Type1 Poor
参考资料:
https://bookdown.org/rdpeng/rprogdatascience/R Programming for Data Science
《R语言实战》 Robert I. Kabacoff
猜你喜欢
热文:1高分文章 2不可或缺的人 3图表规范
一文读懂:1微生物组 2寄生虫益处 3进化树
必备技能:1提问 2搜索 3Endnote
文献阅读 1热心肠 2SemanticScholar 3geenmedical
扩增子分析:1图表解读 2分析流程 3统计绘图 4功能预测
科研经验:1云笔记 2云协作 3公众号
系列教程:1Biostar 2微生物组 3宏基因组
生物科普 1肠道细菌 2人体上的生命 3生命大跃进 4细胞的暗战 5人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外120+ PI,1200+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习16S扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()