R语言聚类分析--cluster, factoextra
R语言聚类分析–cluster, factoextra
本文转载自“R语言中文社区”,己获授权,宏基因组公众号编辑对内容进行测试、修改及补充。
原文链接:https://mp.weixin.qq.com/s/M_rJqcHz6HUlGIHVeN4RkQ
对于有很多(成百上千)研究对象时,把对象分组是最常用的研究手段。而通过观察值进行聚类是非常有效的方法,可以按事物观察值有效的合理分组,再进一步分析各组的相同、与不同,可以很好的发现其中的规律。
本文将带你学习在R语言的Rstudio环境中,使用cluster、facteoextra包,以及kmeans进分析最优分组、评估及可视化。
准备包和数据
# 清空环境
rm(list=ls())
# 安装包并加载包
# 使用k-means聚类所需的包:factoextra和cluster
site="https://mirrors.tuna.tsinghua.edu.cn/CRAN"
package_list = c("factoextra","cluster")
for(p in package_list){
if(!suppressWarnings(suppressMessages(require(p, character.only = TRUE, quietly = TRUE, warn.conflicts = FALSE)))){
install.packages(p, repos=site)
suppressWarnings(suppressMessages(library(p, character.only = TRUE, quietly = TRUE, warn.conflicts = FALSE)))
}
}
# 数据准备
# 使用内置的R数据集USArrests
data("USArrests")
# remove any missing value (i.e, NA values for not available)
USArrests = na.omit(USArrests) #view the first 6 rows of the data
head(USArrests, n=6)
# 显示测试数据示例如下在此数据集中,列是变量,行是观测值。显示测试数据示例如下:
Murder Assault UrbanPop Rape
Alabama 13.2 236 58 21.2
Alaska 10.0 263 48 44.5
Arizona 8.1 294 80 31.0
Arkansas 8.8 190 50 19.5
California 9.0 276 91 40.6
Colorado 7.9 204 78 38.7数据基本统计
在聚类之前我们可以先进行一些必要的数据检查即数据描述性统计,如平均值、标准差等
# 在聚类之前我们可以先进行一些必要的数据检查即数据描述性统计,如平均值、标准差等
desc_stats = data.frame( Min=apply(USArrests, 2, min),#minimum
Med=apply(USArrests, 2, median),#median
Mean=apply(USArrests, 2, mean),#mean
SD=apply(USArrests, 2, sd),#Standard deviation
Max=apply(USArrests, 2, max)#maximum
)
desc_stats = round(desc_stats, 1)#保留小数点后一位head(desc_stats)
desc_stats统计结果如下:
Min Med Mean SD Max
Murder 0.8 7.2 7.8 4.4 17.4
Assault 45.0 159.0 170.8 83.3 337.0
UrbanPop 32.0 66.0 65.5 14.5 91.0
Rape 7.3 20.1 21.2 9.4 46.0数据标准化和评估
# 变量有很大的方差及均值时需进行标准化
df = scale(USArrests)
# 数据集群性评估,使用get_clust_tendency()计算Hopkins统计量
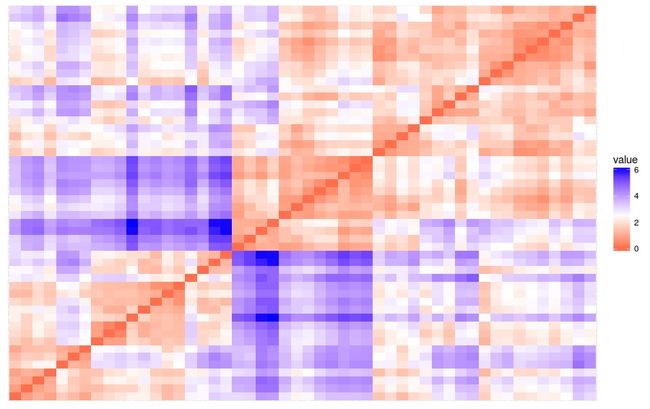
res = get_clust_tendency(df, 40, graph = TRUE)
res$hopkins_stat[1] 0.344087Hopkins统计量的值<0.5,表明数据是高度可聚合的。
res$plot另外,从图中也可以看出数据可聚合。
估计聚合簇数
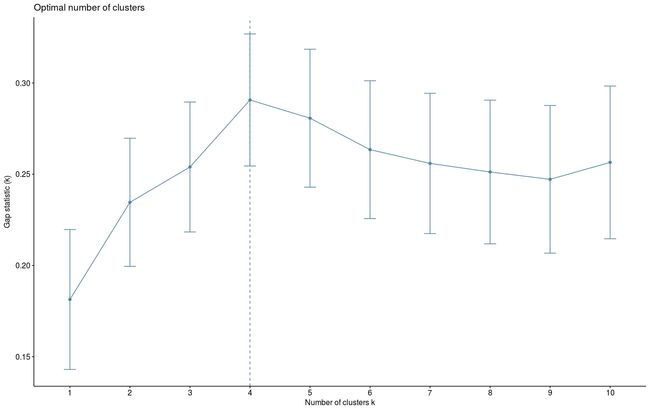
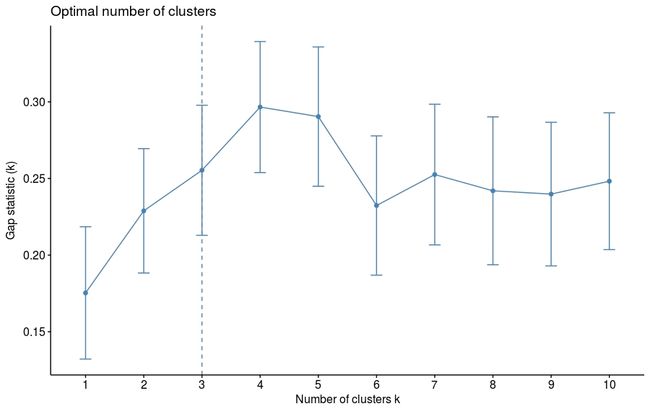
由于k均值聚类需要指定要生成的聚类数量,因此我们将使用函数clusGap()来计算用于估计最优聚类数。函数fviz_gap_stat()用于可视化。
set.seed(123)
## Compute the gap statistic
gap_stat = clusGap(df, FUN = kmeans, nstart = 25, K.max = 10, B = 500)
# Plot the result
fviz_gap_stat(gap_stat)图中显示最佳为聚成四类(k=4)
kmeans进行聚类
kmeans按四组进行聚类,选择25个随机集
km.res = kmeans(df, 4, nstart = 25)
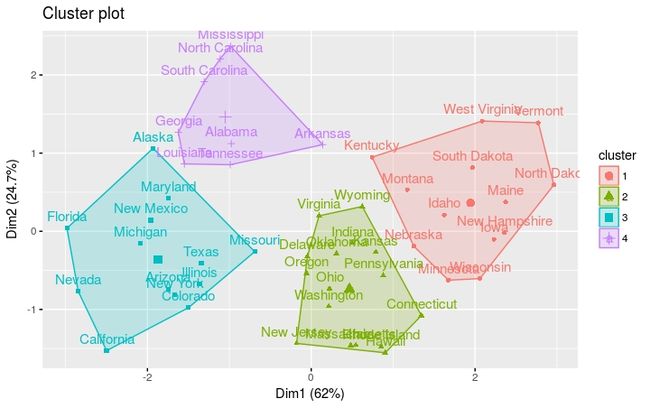
# Visualize clusters using factoextra
fviz_cluster(km.res, USArrests)提取聚类轮廓图
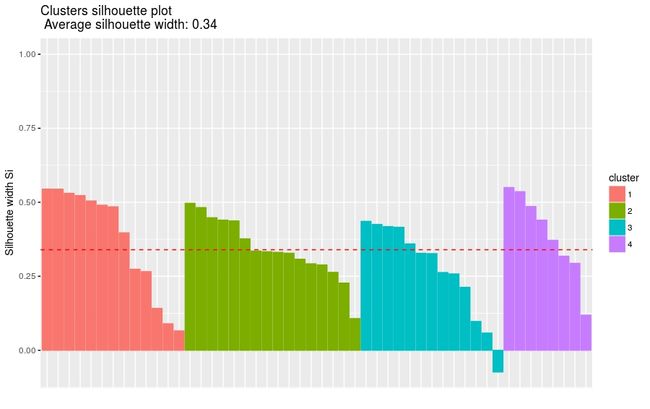
sil = silhouette(km.res$cluster, dist(df))
rownames(sil) = rownames(USArrests)
head(sil[, 1:3])四个cluster的基本信息
cluster size ave.sil.width
1 1 13 0.37
2 2 16 0.34
3 3 13 0.27
4 4 8 0.39可视化
# Visualize
fviz_silhouette(sil)图片尺寸宽900 dpi较适合微信手机端阅读
图中可以看出有负值,可以通过函数silhouette()确定是哪个观测值
neg_sil_index = which(sil[, "sil_width"] < 0)
sil[neg_sil_index, , drop = FALSE]显示为负的观测值
cluster neighbor sil_width
Missouri 3 2 -0.07318144eclust():增强的聚类分析
与其他聚类分析包相比,eclust()有以下优点:
简化了聚类分析的工作流程,可以用于计算层次聚类和分区聚类,eclust()自动计算最佳聚类簇数。
自动提供Silhouette plot,可以结合ggplot2绘制优美的图形,使用eclust()的K均值聚类
# Compute k-means
res.km = eclust(df, "kmeans")
# Gap statistic plot
fviz_gap_stat(res.km$gap_stat)使用eclust()的层次聚类
# Enhanced hierarchical clustering
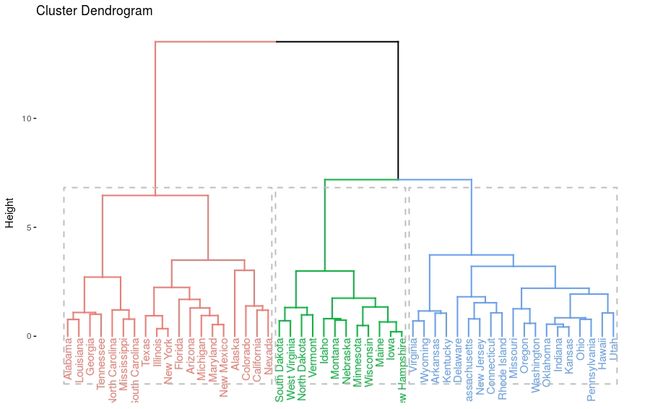
res.hc = eclust(df, "hclust") # compute hclust
fviz_dend(res.hc, rect = TRUE) # dendrogam层级聚类结果
下面的R代码生成Silhouette plot和分层聚类散点图。
fviz_silhouette(res.hc) # silhouette plot
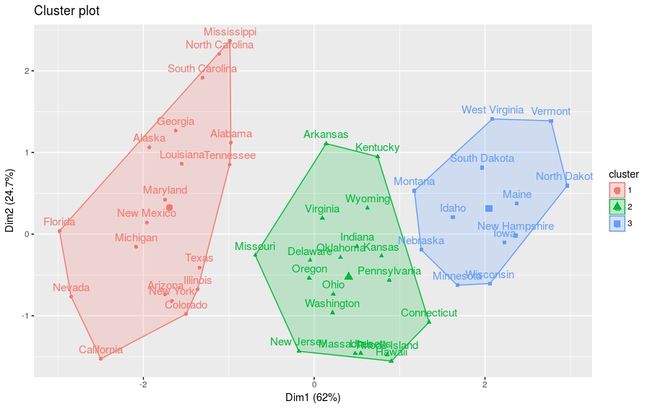
fviz_cluster(res.hc) # scatter plotR分析环境相关信息
sessionInfo()R version 3.4.1 (2017-06-30)
Platform: x86_64-pc-linux-gnu (64-bit)
Running under: Ubuntu 16.04.3 LTS
Matrix products: default
BLAS: /usr/lib/openblas-base/libblas.so.3
LAPACK: /usr/lib/libopenblasp-r0.2.18.so
locale:
[1] LC_CTYPE=en_US.UTF-8 LC_NUMERIC=C LC_TIME=en_US.UTF-8 LC_COLLATE=en_US.UTF-8 LC_MONETARY=en_US.UTF-8
[6] LC_MESSAGES=en_US.UTF-8 LC_PAPER=en_US.UTF-8 LC_NAME=C LC_ADDRESS=C LC_TELEPHONE=C
[11] LC_MEASUREMENT=en_US.UTF-8 LC_IDENTIFICATION=C
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] cluster_2.0.6 factoextra_1.0.5 ggplot2_2.2.1
loaded via a namespace (and not attached):
[1] Rcpp_0.12.15 DEoptimR_1.0-8 pillar_1.1.0 compiler_3.4.1 plyr_1.8.4 ggpubr_0.1.6.999 bindr_0.1
[8] viridis_0.4.1 class_7.3-14 prabclus_2.2-6 tools_3.4.1 dendextend_1.6.0 digest_0.6.14 mclust_5.4
[15] viridisLite_0.2.0 tibble_1.4.2 gtable_0.2.0 lattice_0.20-35 pkgconfig_2.0.1 rlang_0.1.6 ggrepel_0.7.0
[22] yaml_2.1.16 mvtnorm_1.0-6 bindrcpp_0.2 gridExtra_2.3 trimcluster_0.1-2 dplyr_0.7.4 stringr_1.2.0
[29] fpc_2.1-11 diptest_0.75-7 nnet_7.3-12 stats4_3.4.1 grid_3.4.1 robustbase_0.92-8 glue_1.2.0
[36] R6_2.2.2 flexmix_2.3-14 kernlab_0.9-25 reshape2_1.4.3 purrr_0.2.4 magrittr_1.5 whisker_0.3-2
[43] scales_0.5.0 modeltools_0.2-21 MASS_7.3-48 assertthat_0.2.0 colorspace_1.3-2 labeling_0.3 stringi_1.1.6
[50] lazyeval_0.2.1 munsell_0.4.3 猜你喜欢
- 热文:1高分文章 2不可或缺的人 3图表规范
- 一文读懂:1微生物组 2寄生虫益处 3进化树
- 必备技能:1提问 2搜索 3Endnote
- 文献阅读 1热心肠 2SemanticScholar 3geenmedical
- 扩增子分析:1图表解读 2分析流程 3统计绘图 4功能预测
- 科研经验:1云笔记 2云协作 3公众号
- 系列教程:1Biostar 2微生物组 3宏基因组
- 生物科普 1肠道细菌 2人体上的生命 3生命大跃进 4细胞的暗战 5人体奥秘
写在后面
为鼓励读者交流、快速解决科研困难,我们建立了“宏基因组”专业讨论群,目前己有国内外1200+ 一线科研人员加入。参与讨论,获得专业解答,欢迎分享此文至朋友圈,并扫码加主编好友带你入群,务必备注“姓名-单位-研究方向-职称/年级”。技术问题寻求帮助,首先阅读《如何优雅的提问》学习解决问题思路,仍末解决群内讨论,问题不私聊,帮助同行。
![]()
学习扩增子、宏基因组科研思路和分析实战,关注“宏基因组”
![]()
点击阅读原文,跳转最新文章目录阅读
https://mp.weixin.qq.com/s/5jQspEvH5_4Xmart22gjMA