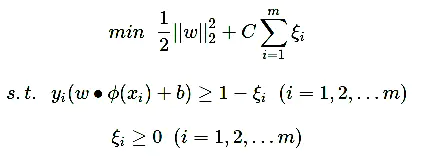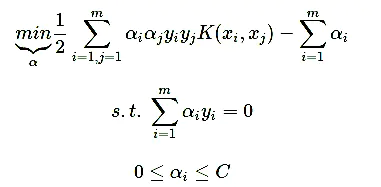- 鸿蒙仓颉开发语言实战教程:页面跳转和传参
湖北穷逼首席代表
harmonyos开发语言华为
合集-仓颉教程(25)1.详解鸿蒙仓颉开发语言中的日志打印问题05-212.鸿蒙仓颉开发语言实战教程:实现商城应用首页05-223.鸿蒙仓颉开发语言实战教程:页面跳转和传参05-254.鸿蒙仓颉语言开发教程:页面和组件的生命周期05-285.鸿蒙仓颉语言开发实战教程:购物车页面06-036.鸿蒙仓颉语言开发实战教程:商城登录页06-047.鸿蒙仓颉语言开发实战教程:商城搜索页06-058.鸿蒙仓颉
- 鸿蒙仓颉语言开发实战教程:购物车页面
王家那谁
harmonyos华为
合集-仓颉教程(25)1.详解鸿蒙仓颉开发语言中的日志打印问题05-212.鸿蒙仓颉开发语言实战教程:实现商城应用首页05-223.鸿蒙仓颉开发语言实战教程:页面跳转和传参05-254.鸿蒙仓颉语言开发教程:页面和组件的生命周期05-285.鸿蒙仓颉语言开发实战教程:购物车页面06-036.鸿蒙仓颉语言开发实战教程:商城登录页06-047.鸿蒙仓颉语言开发实战教程:商城搜索页06-058.鸿蒙仓颉
- MySQL事务深度解析:原理、优化及最佳实践
木木丰
mysqlmysql数据库javawindows
MySQL中的事务(Transaction)是数据库操作的基本单位,它代表着一组逻辑上相互关联的操作,要么全部成功,要么全部失败。这种“要么全做,要么全不做”的特性确保了数据库的完整性和一致性。事务在MySQL中扮演着至关重要的角色,特别是在处理复杂业务逻辑和并发访问时。下面将详细探讨MySQL事务的概念、使用方法、注意事项以及在实际应用中的最佳实践。一、事务的概念事务是一个不可分割的工作逻辑单元
- 深入详解:决策树算法的概念、原理、实现与应用场景
猿享天开
算法决策树机器学习
深入详解:决策树算法的概念、原理、实现与应用场景决策树(DecisionTree)是机器学习中一种直观且广泛应用的监督学习算法,适用于分类和回归任务。其树形结构易于理解,特别适合初学者。本文将从概念、原理、实现到应用场景,全面讲解决策树,并通过流程图和可视化示例增强理解,通俗易懂,帮助小白快速掌握决策树算法相关知识。1.决策树的概念1.1什么是决策树?决策树通过一系列条件判断(决策节点)将输入数据
- ArkTS与仓颉语言的深度解析(鸿蒙操作系统多设备)
爱学习的小齐哥哥
仓颉华为仓颉HarmonyOS5
一、引言随着物联网和智能设备的飞速发展,多设备协同开发成为当前软件开发领域的重要课题。鸿蒙操作系统作为面向全场景的分布式操作系统,为开发者提供了ArkTS和仓颉语言两种强大的开发工具,助力实现高效的多设备应用开发。本文将全面剖析这两种语言在鸿蒙多设备开发中的应用,探讨其优势、开发环境、实现一次开发多端部署的方法以及在不同设备上的性能表现和适配策略,并结合智能驾驶应用场景进行实例分析。二、ArkTS
- [学习] PID算法原理与实践(代码示例)
极客不孤独
学习算法c语言
PID算法原理与实践文章目录PID算法原理与实践一、PID算法原理1.1PID算法概述1.定义2.应用领域3.核心目标1.2基本原理1.3数学表达离散化实现(适用于数字控制)二、实践案例(C语言)1.电机转速控制2.温度控制系统3.时钟驯服系统三、常见问题与优化1.积分饱和(Windup)问题2.噪声干扰问题3.非线性系统适配问题四、扩展方向1.数字PID与模拟PID的差异2.变参数PID(如增益
- LeetCode--38.外观数列
dying_man
leetcode算法
前言:之前我不是说,我后续可能会讲一下递归吗,现在它来了,这道题会用到回溯的方法,并且比较纯粹哦解题思路:1.获取信息:(下面这些信息差不多是力扣上面的题目信息了,所以我这一环节在这次题解中的意义不大)外观数列是一个数位字符串序列,由递归公式定义:countAndSay(1)="1"countAndSay(n)是countAndSay(n-1)的行程长度编码。行程长度编码(RLE)是一种字符串压缩
- 代码随想录算法训练营第52天 | 101.孤岛的总面积 、102.沉没孤岛、103.水流问题、104.建造最大岛屿
Amor_Fati_Yu
算法java数据结构
101.孤岛的总面积importjava.util.*;publicclassMain{privatestaticintcount=0;privatestaticfinalint[][]dir={{0,1},{1,0},{-1,0},{0,-1}};//四个方向privatestaticvoidbfs(int[][]grid,intx,inty){Queueque=newLinkedList=gr
- python实现读取文件的指定某行内容
Fitz1318
Python3学习python
python实现读取文件的指定某行内容最近有一个需求就是读取一个文件中的指定某行的内容,现将方法记录如下importlinecache#这里填写你自己的文件位置和行号text=linecache.getline("../TestFile/test_C1.json",2)print(text)
- Golang Fiber框架最佳实践:如何构建企业级应用
Golang编程笔记
Golang编程笔记Golang开发实战golang开发语言后端ai
GolangFiber框架最佳实践:如何构建企业级应用关键词:Golang、Fiber框架、企业级应用、最佳实践、Web开发摘要:本文聚焦于GolangFiber框架在企业级应用构建中的最佳实践。详细介绍了Fiber框架的背景、核心概念、算法原理、数学模型等基础知识,通过具体的代码案例展示了如何搭建开发环境、实现和解读源代码。同时探讨了Fiber框架在实际应用场景中的应用,推荐了相关的学习资源、开
- 代码随想录算法训练营第52天| 101. 孤岛的总面积、102. 沉没孤岛、103. 水流问题、104.建造最大岛屿
扛过今天777
算法深度优先
101.孤岛的总面积卡码题目链接:101.孤岛的总面积学习链接:代码随想录题解:法一:count=0defdfs(grid,x,y):globalcountgrid[x][y]=0count+=1directions=[[1,0],[0,1],[-1,0],[0,-1]]fori,jindirections:next_x=x+inext_y=y+jifnext_x=len(grid)ornext_
- 深入研究 Golang 领域的 Fiber 框架架构
Golang编程笔记
golang架构网络ai
深入研究Golang领域的Fiber框架架构关键词:Golang、Fiber框架、架构、高性能、Web开发摘要:本文将深入探讨Golang领域的Fiber框架架构。我们会先介绍背景知识,包括目的、预期读者等。接着用通俗易懂的方式解释核心概念,如Fiber框架的各个组成部分,以及它们之间的关系。然后详细阐述核心算法原理、数学模型,通过实际代码案例展示其应用。还会介绍Fiber框架的实际应用场景、推荐
- Go语言--包(Package)
yunfan188
#Go语言学习笔记gogo语言golangpackage
1命名空间和作用域1.1命名空间命名空间(Namespace)在编程语言中常用来表示标识符(identifier)的可见范围。编程语言借助命名空间来解决标识符不能同名的问题,命名空间实际上相当于给标识符添加了标识前缀,使标识符变得全局唯一。另外,命名空间是程序组织更加模块化,降低了程序内部的耦合性。一个标识符可以在多个命名空间中定义,它在不同命名空间中的含义是不互相干的。新的命名空间中可定义任意的
- 如何在FastAPI中打造坚不可摧的Web安全防线?
url:/posts/9d6200ae7ce0a1a1a523591e3d65a82e/title:如何在FastAPI中打造坚不可摧的Web安全防线?date:2025-06-28T08:37:03+08:00lastmod:2025-06-28T08:37:03+08:00author:cmdragonsummary:Web安全三要素包括机密性、完整性和可用性。机密性通过加密算法保护数据传输和
- 算法竞赛备考冲刺必刷题(C++) | 洛谷 P8814 解密
热爱编程的通信人
算法c++开发语言
本文分享的必刷题目是从蓝桥云课、洛谷、AcWing等知名刷题平台精心挑选而来,并结合各平台提供的算法标签和难度等级进行了系统分类。题目涵盖了从基础到进阶的多种算法和数据结构,旨在为不同阶段的编程学习者提供一条清晰、平稳的学习提升路径。欢迎大家订阅我的专栏:算法题解:C++与Python实现!附上汇总贴:算法竞赛备考冲刺必刷题(C++)|汇总【题目来源】洛谷:P8814[CSP-J2022]解密-洛
- Java Fork/Join 框架详解
empti_
数据结构与算法java
JavaFork/Join框架详解Fork/Join框架是Java7引入的一个并行编程框架,专门设计用来高效地实现分治算法(Divide-and-Conquer)。它通过工作窃取(Work-Stealing)算法来最大化多核处理器的利用率。一、核心概念1.基本组成ForkJoinPool:特殊的线程池,管理工作线程ForkJoinTask:表示任务的抽象类,有两个重要子类:RecursiveAct
- Java注解的实现原理
empti_
Java基础java
Java注解的实现原理Java注解的实现涉及Java语言规范、编译器处理和JVM支持等多个层面。下面我将详细解释注解在Java中的实现机制。一、注解的本质注解本质上是一种特殊的接口,所有注解类型都隐式继承自java.lang.annotation.Annotation接口。当你定义一个注解时:public@interfaceMyAnnotation{Stringvalue();}编译器实际上会生成
- 程序化交易系统中如何精准获取MACD、KDJ、BOLL等基础指标的值?
股票程序化交易接口
量化交易股票API接口Python股票量化交易程序化交易系统macd指标kdj指标boll指标股票量化接口股票API接口
Python股票接口实现查询账户,提交订单,自动交易(1)Python股票程序交易接口查账,提交订单,自动交易(2)股票量化,Python炒股,CSDN交流社区>>>基础指标在程序化交易系统中的重要性基础指标对交易决策的指导意义MACD、KDJ、BOLL等基础指标在程序化交易系统中扮演着重要角色。MACD可以帮助判断市场的趋势和买卖信号,通过分析其快线和慢线的交叉情况,能为投资者提供入场和出场的参
- 【C++】atoi和std::stoi
bluebonnet27
编程语言#C++c++算法开发语言
两个将字符串转为int的方法atoi(C语言)atoi是C库中的一个函数,它定义在头文件里。其作用是把一个字符串转换为对应的整数。/*Convertastringtoaninteger.*/externintatoi(constchar*__nptr)__THROW__attribute_pure____nonnull((1))__wur;转换的原则如下:此函数接收一个以空字符'\0'结尾的字符串
- 人脸识别算法赋能园区无人超市安防升级
智驱力人工智能
算法人工智能边缘计算人脸识别智慧园区智慧工地智慧煤矿
人脸识别算法赋能园区无人超市安防升级正文在园区无人超市的运营管理中,传统安防手段依赖人工巡检或基础监控设备,存在响应滞后、误报率高、环境适应性差等问题。本文从技术背景、实现路径、功能优势及应用场景四个维度,阐述如何通过人脸识别检测、人员入侵算法及疲劳检测算法的协同应用,构建高效、精准的智能安防体系。一、技术背景:视觉分析算法的核心支撑人脸识别算法基于深度学习的卷积神经网络(CNN)模型,通过提取面
- 游戏寻路之A*算法(GUI演示)
jforgame
从零开始搭建游戏服务器框架javaA星自动寻路
一、A*算法介绍A*算法是一种路径搜索算法,用于在图形网络中找到最短路径。它结合了Dijkstra算法和启发式搜索的思想,通过综合利用已知的最短路径和估计的最短路径来优化搜索过程。在游戏自动寻路得到广泛应用。二、A*算法的基本思想在图形网络中选择一个起点和终点。维护两个列表:开放列表和关闭列表。开放列表用于存储待考虑的节点,关闭列表用于存储已考虑过的节点。将起点加入开放列表。循环以下工作当open
- Linux ss 指令
halugin
Linux指令linux运维
Linuxss指令ss(SocketStatistics)是Linux系统中用于显示网络套接字(socket)信息的现代命令行工具,是netstat的继任者,性能更高、输出更简洁。它提供详细的网络连接、监听端口和协议统计信息,广泛用于网络监控、故障排查和性能分析。相比传统的netstat,ss直接从内核获取数据显示更快,功能更强大,适合现代Linux系统。什么是ss指令?ss是Linux系统中的一
- 疲劳检测与行为分析:工厂智能化实践
智驱力人工智能
安全智慧城市行为识别人员属性识别疲劳检测抽烟检测徘徊检测
视觉分析算法赋能工厂疲劳与安全管理一、背景与需求在制造业中,疲劳作业是导致安全事故和效率下降的核心因素之一。传统人工巡检存在覆盖面不足、响应滞后等问题,而基于视觉分析的智能监控系统通过多算法协同,可实现全天候、高精度的疲劳检测与行为管理。本文围绕疲劳检测算法、人员计数算法、抽烟检测算法及徘徊检测算法,探讨其在工厂场景中的技术实现与应用价值。二、技术实现疲劳检测算法原理:基于PERCLOS(眼睑闭合
- 为什么90%企业的AI数据分析都失败了?奥威BI给出破局方案
qq_43696218
人工智能数据分析数据挖掘
一、引言:AI数据分析在数字化转型中的核心地位在当今企业全面数字化转型的背景下,AI数据分析已成为解锁业务增长潜力的关键钥匙。然而,市场上众多AI数据分析产品常陷入“伪需求场景”,看似前沿却难以真正落地。本文将深入探讨奥威BI如何通过其AI数据分析能力,突破伪需求,实现数据价值的最大化。二、AI数据分析:伪需求场景的挑战伪需求场景的定义与表现AI数据分析领域的伪需求场景,指的是那些表面创新实则难
- 010 【入门】链表入门题目-合并两个有序链表
要天天开心啊
算法专栏链表数据结构
合并两个有序链表|[算法]-[中级]-[链表]▶JDK8+|⏱️O(m+n)核心代码实现packageclass010;//将两个升序链表合并为一个新的升序链表并返回//新链表是通过拼接给定的两个链表的所有节点组成的//测试链接:https://leetcode.cn/problems/merge-two-sorted-lists/publicclassMergeTwoLists{//链表节点定义
- 008 【入门】算法和数据结构简介
要天天开心啊
算法专栏算法数据结构
算法与数据结构系统概览|[算法]-[基础]-[通用]一、算法分类与应用1.硬计算类算法|[算法]-[中级]-[通用]特点应用场景复杂度特征-精确求解问题-可能带来较高计算复杂度-大厂笔试/面试-ACM竞赛-所有程序员岗位必考⏱️通常为O(n)~O(n²)//[示例]快速排序算法-分治思想核心实现publicvoidquickSort(int[]arr,intleft,intright){if(le
- Python3 数字(Number)
froginwe11
开发语言
Python3数字(Number)引言在编程语言中,数字是构成程序的基础元素之一。Python3作为一种高级编程语言,提供了丰富的数字类型和操作方法。本文将详细介绍Python3中的数字类型,包括整数、浮点数、复数等,并探讨它们的特性和应用。整数(Integer)整数是Python3中最基本的数据类型之一,用于表示没有小数部分的数值。在Python3中,整数类型没有大小限制,可以表示任意大小的整数
- Ruby 字符串(String)
froginwe11
开发语言
Ruby字符串(String)引言在编程语言中,字符串是处理文本数据的基础。Ruby作为一种动态、面向对象的语言,提供了丰富的字符串处理功能。本文将详细介绍Ruby中的字符串(String)类型,包括其基本用法、操作方法以及高级特性。字符串的基本概念在Ruby中,字符串是由一系列字符组成的序列。这些字符可以是字母、数字、标点符号等。字符串是不可变的,这意味着一旦创建,其内容就不能被修改。创建字符串
- 009 【入门】单双链表及其反转-堆栈诠释
要天天开心啊
算法专栏算法链表
链表与堆栈系统详解|[数据结构]-[中级]-[通用]一、基础概念与内存模型1.按值传递vs按引用传递|[Java]-[基础]-[内存]//[典型错误示例]-Java中的引用传递陷阱voidmodify(Nodenode){node=node.next;//[警告]错误!仅修改局部引用的指向,不影响原始链表}//[正确做法]-通过引用修改对象内部状态voidrealModify(Nodenode){
- GitHub Actions 的深度解析与概念介绍
青草地溪水旁
linux环境配置开发管理githublinuxubuntudocker
GitHubActions核心定义GitActions是GitHub原生提供的自动化工作流引擎,允许开发者在代码仓库中直接创建、测试、部署代码。其本质是通过事件驱动(Event-Driven)的自动化管道,将软件开发中的重复任务抽象为可编排的流程。架构核心四要素工作流(Workflow)仓库中的自动化流程蓝图(.yml文件)存储在.github/workflows目录单仓库可包含多个独立工作流事件
- Spring4.1新特性——综述
jinnianshilongnian
spring 4.1
目录
Spring4.1新特性——综述
Spring4.1新特性——Spring核心部分及其他
Spring4.1新特性——Spring缓存框架增强
Spring4.1新特性——异步调用和事件机制的异常处理
Spring4.1新特性——数据库集成测试脚本初始化
Spring4.1新特性——Spring MVC增强
Spring4.1新特性——页面自动化测试框架Spring MVC T
- Schema与数据类型优化
annan211
数据结构mysql
目前商城的数据库设计真是一塌糊涂,表堆叠让人不忍直视,无脑的架构师,说了也不听。
在数据库设计之初,就应该仔细揣摩可能会有哪些查询,有没有更复杂的查询,而不是仅仅突出
很表面的业务需求,这样做会让你的数据库性能成倍提高,当然,丑陋的架构师是不会这样去考虑问题的。
选择优化的数据类型
1 更小的通常更好
更小的数据类型通常更快,因为他们占用更少的磁盘、内存和cpu缓存,
- 第一节 HTML概要学习
chenke
htmlWebcss
第一节 HTML概要学习
1. 什么是HTML
HTML是英文Hyper Text Mark-up Language(超文本标记语言)的缩写,它规定了自己的语法规则,用来表示比“文本”更丰富的意义,比如图片,表格,链接等。浏览器(IE,FireFox等)软件知道HTML语言的语法,可以用来查看HTML文档。目前互联网上的绝大部分网页都是使用HTML编写的。
打开记事本 输入一下内
- MyEclipse里部分习惯的更改
Array_06
eclipse
继续补充中----------------------
1.更改自己合适快捷键windows-->prefences-->java-->editor-->Content Assist-->
Activation triggers for java的右侧“.”就可以改变常用的快捷键
选中 Text
- 近一个月的面试总结
cugfy
面试
本文是在学习中的总结,欢迎转载但请注明出处:http://blog.csdn.net/pistolove/article/details/46753275
前言
打算换个工作,近一个月面试了不少的公司,下面将一些面试经验和思考分享给大家。另外校招也快要开始了,为在校的学生提供一些经验供参考,希望都能找到满意的工作。
- HTML5一个小迷宫游戏
357029540
html5
通过《HTML5游戏开发》摘抄了一个小迷宫游戏,感觉还不错,可以画画,写字,把摘抄的代码放上来分享下,喜欢的同学可以拿来玩玩!
<html>
<head>
<title>创建运行迷宫</title>
<script type="text/javascript"
- 10步教你上传githib数据
张亚雄
git
官方的教学还有其他博客里教的都是给懂的人说得,对已我们这样对我大菜鸟只能这么来锻炼,下面先不玩什么深奥的,先暂时用着10步干净利索。等玩顺溜了再用其他的方法。
操作过程(查看本目录下有哪些文件NO.1)ls
(跳转到子目录NO.2)cd+空格+目录
(继续NO.3)ls
(匹配到子目录NO.4)cd+ 目录首写字母+tab键+(首写字母“直到你所用文件根就不再按TAB键了”)
(查看文件
- MongoDB常用操作命令大全
adminjun
mongodb操作命令
成功启动MongoDB后,再打开一个命令行窗口输入mongo,就可以进行数据库的一些操作。输入help可以看到基本操作命令,只是MongoDB没有创建数据库的命令,但有类似的命令 如:如果你想创建一个“myTest”的数据库,先运行use myTest命令,之后就做一些操作(如:db.createCollection('user')),这样就可以创建一个名叫“myTest”的数据库。
一
- bat调用jar包并传入多个参数
aijuans
下面的主程序是通过eclipse写的:
1.在Main函数接收bat文件传递的参数(String[] args)
如: String ip =args[0]; String user=args[1]; &nbs
- Java中对类的主动引用和被动引用
ayaoxinchao
java主动引用对类的引用被动引用类初始化
在Java代码中,有些类看上去初始化了,但其实没有。例如定义一定长度某一类型的数组,看上去数组中所有的元素已经被初始化,实际上一个都没有。对于类的初始化,虚拟机规范严格规定了只有对该类进行主动引用时,才会触发。而除此之外的所有引用方式称之为对类的被动引用,不会触发类的初始化。虚拟机规范严格地规定了有且仅有四种情况是对类的主动引用,即必须立即对类进行初始化。四种情况如下:1.遇到ne
- 导出数据库 提示 outfile disabled
BigBird2012
mysql
在windows控制台下,登陆mysql,备份数据库:
mysql>mysqldump -u root -p test test > D:\test.sql
使用命令 mysqldump 格式如下: mysqldump -u root -p *** DBNAME > E:\\test.sql。
注意:执行该命令的时候不要进入mysql的控制台再使用,这样会报
- Javascript 中的 && 和 ||
bijian1013
JavaScript&&||
准备两个对象用于下面的讨论
var alice = {
name: "alice",
toString: function () {
return this.name;
}
}
var smith = {
name: "smith",
- [Zookeeper学习笔记之四]Zookeeper Client Library会话重建
bit1129
zookeeper
为了说明问题,先来看个简单的示例代码:
package com.tom.zookeeper.book;
import com.tom.Host;
import org.apache.zookeeper.WatchedEvent;
import org.apache.zookeeper.ZooKeeper;
import org.apache.zookeeper.Wat
- 【Scala十一】Scala核心五:case模式匹配
bit1129
scala
package spark.examples.scala.grammars.caseclasses
object CaseClass_Test00 {
def simpleMatch(arg: Any) = arg match {
case v: Int => "This is an Int"
case v: (Int, String)
- 运维的一些面试题
yuxianhua
linux
1、Linux挂载Winodws共享文件夹
mount -t cifs //1.1.1.254/ok /var/tmp/share/ -o username=administrator,password=yourpass
或
mount -t cifs -o username=xxx,password=xxxx //1.1.1.1/a /win
- Java lang包-Boolean
BrokenDreams
boolean
Boolean类是Java中基本类型boolean的包装类。这个类比较简单,直接看源代码吧。
public final class Boolean implements java.io.Serializable,
- 读《研磨设计模式》-代码笔记-命令模式-Command
bylijinnan
java设计模式
声明: 本文只为方便我个人查阅和理解,详细的分析以及源代码请移步 原作者的博客http://chjavach.iteye.com/
import java.util.ArrayList;
import java.util.Collection;
import java.util.List;
/**
* GOF 在《设计模式》一书中阐述命令模式的意图:“将一个请求封装
- matlab下GPU编程笔记
cherishLC
matlab
不多说,直接上代码
gpuDevice % 查看系统中的gpu,,其中的DeviceSupported会给出matlab支持的GPU个数。
g=gpuDevice(1); %会清空 GPU 1中的所有数据,,将GPU1 设为当前GPU
reset(g) %也可以清空GPU中数据。
a=1;
a=gpuArray(a); %将a从CPU移到GPU中
onGP
- SVN安装过程
crabdave
SVN
SVN安装过程
subversion-1.6.12
./configure --prefix=/usr/local/subversion --with-apxs=/usr/local/apache2/bin/apxs --with-apr=/usr/local/apr --with-apr-util=/usr/local/apr --with-openssl=/
- sql 行列转换
daizj
sql行列转换行转列列转行
行转列的思想是通过case when 来实现
列转行的思想是通过union all 来实现
下面具体例子:
假设有张学生成绩表(tb)如下:
Name Subject Result
张三 语文 74
张三 数学 83
张三 物理 93
李四 语文 74
李四 数学 84
李四 物理 94
*/
/*
想变成
姓名 &
- MySQL--主从配置
dcj3sjt126com
mysql
linux下的mysql主从配置: 说明:由于MySQL不同版本之间的(二进制日志)binlog格式可能会不一样,因此最好的搭配组合是Master的MySQL版本和Slave的版本相同或者更低, Master的版本肯定不能高于Slave版本。(版本向下兼容)
mysql1 : 192.168.100.1 //master mysq
- 关于yii 数据库添加新字段之后model类的修改
dcj3sjt126com
Model
rules:
array('新字段','safe','on'=>'search')
1、array('新字段', 'safe')//这个如果是要用户输入的话,要加一下,
2、array('新字段', 'numerical'),//如果是数字的话
3、array('新字段', 'length', 'max'=>100),//如果是文本
1、2、3适当的最少要加一条,新字段才会被
- sublime text3 中文乱码解决
dyy_gusi
Sublime Text
sublime text3中文乱码解决
原因:缺少转换为UTF-8的插件
目的:安装ConvertToUTF8插件包
第一步:安装能自动安装插件的插件,百度“Codecs33”,然后按照步骤可以得到以下一段代码:
import urllib.request,os,hashlib; h = 'eb2297e1a458f27d836c04bb0cbaf282' + 'd0e7a30980927
- 概念了解:CGI,FastCGI,PHP-CGI与PHP-FPM
geeksun
PHP
CGI
CGI全称是“公共网关接口”(Common Gateway Interface),HTTP服务器与你的或其它机器上的程序进行“交谈”的一种工具,其程序须运行在网络服务器上。
CGI可以用任何一种语言编写,只要这种语言具有标准输入、输出和环境变量。如php,perl,tcl等。 FastCGI
FastCGI像是一个常驻(long-live)型的CGI,它可以一直执行着,只要激活后,不
- Git push 报错 "error: failed to push some refs to " 解决
hongtoushizi
git
Git push 报错 "error: failed to push some refs to " .
此问题出现的原因是:由于远程仓库中代码版本与本地不一致冲突导致的。
由于我在第一次git pull --rebase 代码后,准备push的时候,有别人往线上又提交了代码。所以出现此问题。
解决方案:
1: git pull
2:
- 第四章 Lua模块开发
jinnianshilongnian
nginxlua
在实际开发中,不可能把所有代码写到一个大而全的lua文件中,需要进行分模块开发;而且模块化是高性能Lua应用的关键。使用require第一次导入模块后,所有Nginx 进程全局共享模块的数据和代码,每个Worker进程需要时会得到此模块的一个副本(Copy-On-Write),即模块可以认为是每Worker进程共享而不是每Nginx Server共享;另外注意之前我们使用init_by_lua中初
- java.lang.reflect.Proxy
liyonghui160com
1.简介
Proxy 提供用于创建动态代理类和实例的静态方法
(1)动态代理类的属性
代理类是公共的、最终的,而不是抽象的
未指定代理类的非限定名称。但是,以字符串 "$Proxy" 开头的类名空间应该为代理类保留
代理类扩展 java.lang.reflect.Proxy
代理类会按同一顺序准确地实现其创建时指定的接口
- Java中getResourceAsStream的用法
pda158
java
1.Java中的getResourceAsStream有以下几种: 1. Class.getResourceAsStream(String path) : path 不以’/'开头时默认是从此类所在的包下取资源,以’/'开头则是从ClassPath根下获取。其只是通过path构造一个绝对路径,最终还是由ClassLoader获取资源。 2. Class.getClassLoader.get
- spring 包官方下载地址(非maven)
sinnk
spring
SPRING官方网站改版后,建议都是通过 Maven和Gradle下载,对不使用Maven和Gradle开发项目的,下载就非常麻烦,下给出Spring Framework jar官方直接下载路径:
http://repo.springsource.org/libs-release-local/org/springframework/spring/
s
- Oracle学习笔记(7) 开发PLSQL子程序和包
vipbooks
oraclesql编程
哈哈,清明节放假回去了一下,真是太好了,回家的感觉真好啊!现在又开始出差之旅了,又好久没有来了,今天继续Oracle的学习!
这是第七章的学习笔记,学习完第六章的动态SQL之后,开始要学习子程序和包的使用了……,希望大家能多给俺一些支持啊!
编程时使用的工具是PLSQL