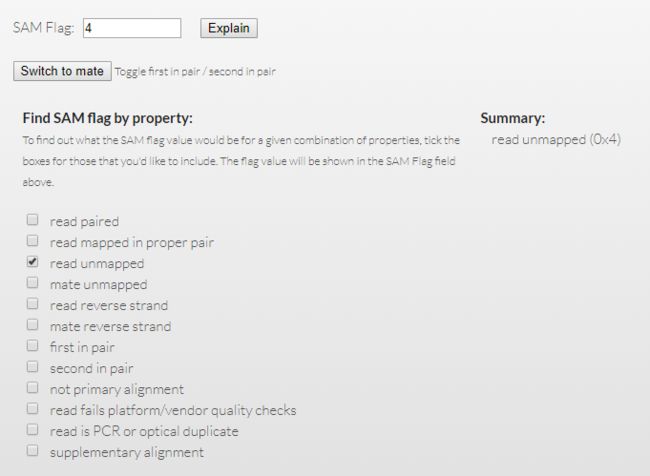
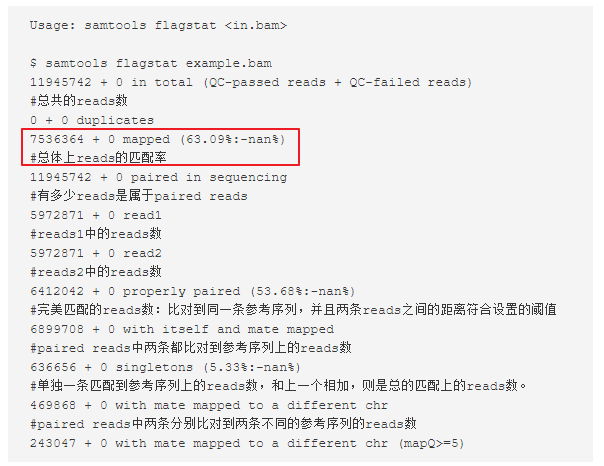
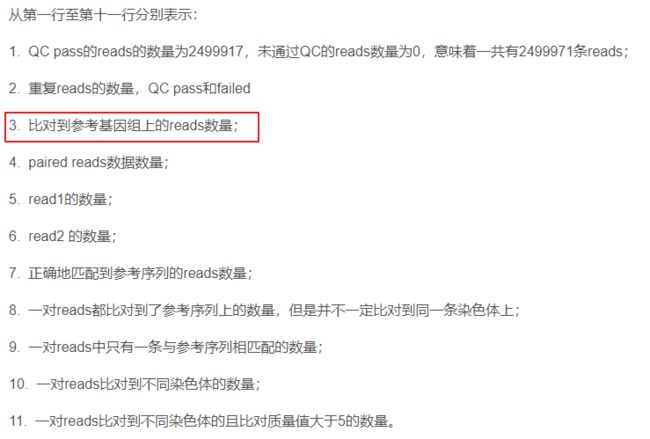
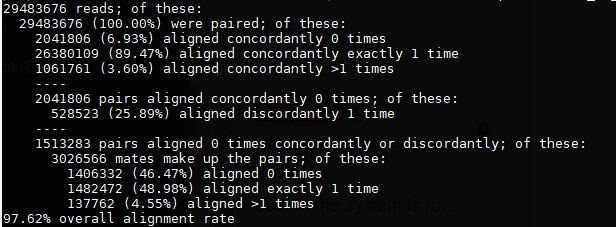
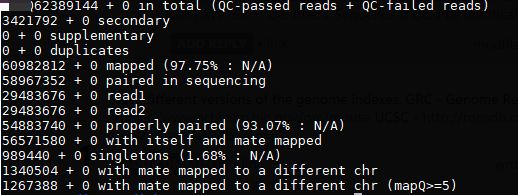
- c++ 的iostream 和 c++的stdio的区别和联系
黄卷青灯77
c++算法开发语言iostreamstdio
在C++中,iostream和C语言的stdio.h都是用于处理输入输出的库,但它们在设计、用法和功能上有许多不同。以下是两者的区别和联系:区别1.编程风格iostream(C++风格):C++标准库中的输入输出流类库,支持面向对象的输入输出操作。典型用法是cin(输入)和cout(输出),使用>操作符来处理数据。更加类型安全,支持用户自定义类型的输入输出。#includeintmain(){in
- Python中os.environ基本介绍及使用方法
鹤冲天Pro
#Pythonpython服务器开发语言
文章目录python中os.environos.environ简介os.environ进行环境变量的增删改查python中os.environ的使用详解1.简介2.key字段详解2.1常见key字段3.os.environ.get()用法4.环境变量的增删改查和判断是否存在4.1新增环境变量4.2更新环境变量4.3获取环境变量4.4删除环境变量4.5判断环境变量是否存在python中os.envi
- Python教程:一文了解使用Python处理XPath
旦莫
Python进阶python开发语言
目录1.环境准备1.1安装lxml1.2验证安装2.XPath基础2.1什么是XPath?2.2XPath语法2.3示例XML文档3.使用lxml解析XML3.1解析XML文档3.2查看解析结果4.XPath查询4.1基本路径查询4.2使用属性查询4.3查询多个节点5.XPath的高级用法5.1使用逻辑运算符5.2使用函数6.实战案例6.1从网页抓取数据6.1.1安装Requests库6.1.2代
- 使用Faiss进行高效相似度搜索
llzwxh888
faisspython
在现代AI应用中,快速和高效的相似度搜索是至关重要的。Faiss(FacebookAISimilaritySearch)是一个专门用于快速相似度搜索和聚类的库,特别适用于高维向量。本文将介绍如何使用Faiss来进行相似度搜索,并结合Python代码演示其基本用法。什么是Faiss?Faiss是一个由FacebookAIResearch团队开发的开源库,主要用于高维向量的相似性搜索和聚类。Faiss
- log4j配置
yy爱yy
#log4j.rootLogger配置的是大于等于当前级别的日志信息的输出#log4j.rootLogger用法:(注意appenderName可以是一个或多个)#log4j.rootLogger=日志级别,appenderName1,appenderName2,....#log4j.appender.appenderName2定义的是日志的输出方式,有两种:一种是命令行输出或者叫控制台输出,另一
- ExpRe[25] bash外的其它shell:zsh和fish
tritone
ExpRebashlinuxubuntushell
文章目录zsh基础配置实用特性插件`autojump`语法高亮自动补全fish优点缺点时效性本篇撰写时间为2021.12.15,由于计算机技术日新月异,博客中所有内容都有时效和版本限制,具体做法不一定总行得通,链接可能改动失效,各种软件的用法可能有修改。但是其中透露的思想往往是值得学习的。本篇前置:ExpRe[10]Ubuntu[2]准备神秘软件、备份恢复软件https://www.cnblogs
- Linux sh命令
fengyehongWorld
Linuxlinux
目录一.基本语法二.选项2.1-c字符串中读取内容,并执行2.1.1基本用法2.1.2获取当前目录下失效的超链接2.2-x每个命令执行之前,将其打印出来2.3结合Here文档使用一.基本语法⏹Linux和Unix系统中用于执行shell脚本或运行命令的命令。sh[选项][脚本文件][参数...]⏹选项-c:从字符串中读取内容,并执行。-x:在每个命令执行之前,将其打印出来。-s:从标准流中读取内容
- Linux查看服务器日志
TPBoreas
运维linux运维
一、tail这个是我最常用的一种查看方式用法如下:tail-n10test.log查询日志尾部最后10行的日志;tail-n+10test.log查询10行之后的所有日志;tail-fn10test.log循环实时查看最后1000行记录(最常用的)一般还会配合着grep用,(实时抓包)例如:tail-fn1000test.log|grep'关键字'(动态抓包)tail-fn1000test.log
- Vue中table合并单元格用法
weixin_30613343
javascriptViewUI
地名结果人名性别{{item.name}}已完成未完成{{item.groups[0].name}}{{item.groups[0].sex}}{{item.groups[son].name}}{{item.groups[son].sex}}exportdefault{data(){return{list:[{name:'地名1',result:'1',groups:[{name:'张三',sex
- python获取子进程返回值_Python对进程Multiprocessing子进程返回值
weixin_39752157
python获取子进程返回值
在实际使用多进程的时候,可能需要获取到子进程运行的返回值。如果只是用来存储,则可以将返回值保存到一个数据结构中;如果需要判断此返回值,从而决定是否继续执行所有子进程,则会相对比较复杂。另外在Multiprocessing中,可以利用Process与Pool创建子进程,这两种用法在获取子进程返回值上的写法上也不相同。这篇中,我们直接上代码,分析多进程中获取子进程返回值的不同用法,以及优缺点。初级用法
- Python编程 - 函数进阶
易辰君
Python核心编程python开发语言
目录前言一、函数参数的高级用法(一)缺省参数(二)命名参数(三)不定长参数二、拆包(一)函数返回值拆包(二)通过星号拆包(三)总结三、匿名函数(一)函数定义(二)使用匿名函数四、递归函数(一)简介(二)基本结构(三)简单示例(四)优缺点总结前言上篇文章主要了解了函数基础,如何定义函数,函数种类以及局部变量和全局变量的差异等,接下来就讲解python函数较为进阶的知识点,若有任何想法欢迎一起沟通讨论
- linux脚本sed替换变量,sed 命令中替换值为shell变量
诺坎普之约
linux脚本sed替换变量
文章目录sed命令中替换值为shell变量替换基本语法sed中替换使用shell变量总结参考文档sed命令中替换值为shell变量替换基本语法大家都是sed有很多用法,最多就应该是替换一些值了。让我们先回忆sed的替换语法。在sed进行替换的时候sed-i's/old/new/g'1.txtecho"hellooldfrank"|sed's/old/new/g'结果如下:hellonewfrank
- Dockerfile命令详解之 FROM
清风怎不知意
容器化java前端javascript
许多同学不知道Dockerfile应该如何写,不清楚Dockerfile中的指令分别有什么意义,能达到什么样的目的,接下来我将在容器化专栏中详细的为大家解释每一个指令的含义以及用法。专栏订阅传送门https://blog.csdn.net/qq_38220908/category_11989778.html指令不区分大小写。但是,按照惯例,它们应该是大写的,以便更容易地将它们与参数区分开来。(引用
- scanf占位符的一些用法
阿玉的屋檐
c语言初学者算法数据结构c语言青少年编程学习
1.限制输入数据的长度intmain(){inta=123456;scanf("%3d",&a);printf("%d",a);return0;}如果输入的值大于3位则最多读取输入的只读取前3位数据。2.匹配特定字符charss[6];scanf("%[abcd]",ss);%[abcd]表示只读取字符abcd,遇到其它的字符就读取结束,如果abcd字符在字符串的中间部分那么就不能正常读取字符。如
- 深入理解Python中的“_,”:一个实用的语法特性
小桥流水---人工智能
Python程序代码Python常见bug深度学习python开发语言
在Python编程中,你可能经常会看到一个特殊的标识符“_”。这个符号在Python中有多种用途,其具体含义依上下文而定。本文将探讨其中一种常见用法——作为一个临时性的占位符——并解释它在实际编程中的实用性和应用场景。1.“_”作为占位符在Python中,下划线(_)经常被用作一个临时或不重要的变量。当你在解包(unpacking)一个表达式但又不想使用其中某些值时,下划线可用作占位符。这样做的好
- k8s中Service暴露的种类以及用法
听说唐僧不吃肉
K8Skubernetes容器云原生
一、说明在Kubernetes中,有几种不同的方式可以将服务(Service)暴露给外部流量。这些方式通过定义服务的spec.type字段来确定。二、详解1.ClusterIP定义:默认类型,服务只能在集群内部访问。作用:通过集群内部IP地址暴露服务。示例:spec:type:ClusterIPports:-port:80targetPo
- python中zeros用法_Python中的numpy.zeros()用法
江平舟
python中zeros用法
numpy.zeros()函数是最重要的函数之一,广泛用于机器学习程序中。此函数用于生成包含零的数组。numpy.zeros()函数提供给定形状和类型的新数组,并用零填充。句法numpy.zeros(shape,dtype=float,order='C'参数形状:整数或整数元组此参数用于定义数组的尺寸。此参数用于我们要在其中创建数组的形状,例如(3,2)或2。dtype:数据类型(可选)此参数用于
- svg图片兼容性和用法优缺点
独行侠_ef93
svg图片的使用方法第一次来认认真真的研究了下svg图片,之前只是在网上见过,但都是一晃而过也没当回事,最近网站改版看到同事有用到svg格式的图片,想想自己干了几年的重构也没用过,这些细节的知识是应该好好研究研究了。暂时还没研究得完全透切,先记下目前为止所看到的吧不然又给忘了。svg可缩放矢量图形(ScalableVectorGraphics),顾名思义就是任意改变其大小也不会变形,是基于可扩展标
- Spring @Async 深度解读:默认线程池执行器的配置与优化
小码快撩
springjava前端
在Spring中,@Async注解用于异步执行方法。默认情况下,@Async注解的任务是由一个线程池执行的。然而,这个默认的线程池是如何初始化的呢?本文将深入探讨这一过程,帮助你理解Spring异步任务背后的线程池执行器的初始化原理。1.@Async的基本使用首先,让我们快速回顾一下@Async的基本用法。@Async通常用于标注在需要异步执行的方法上,比如:@Servicepublicclass
- Three.js AnimationUtils 和 AnimationObjectGroup
灵魂清零
three前端web3javascript
AnimationObjectGroup接收共享动画状态的一组对象。在使用手册的“下一步”章节中,“动画系统”一文对three.js动画系统中的不同元素作出了概述用法:将本来要作为根对象传入构造器或者动画混合器(AnimationMixer)的clipAction方法中的对象加入组中,并将这个组对象作为根对象传递。注意,这个类的实例作为混合器中的一个对象,因此,必须对组内的单个对象做缓存控制。限制
- 说说在 Vue.js 中如何实现组件间通信
deniro
1用法假设父组件的模板包含子组件,我们可以通过props来正向地把数据从父组件传递给子组件。props可以是字符串数组,也可以是对象。html:js:Vue.component('deniro-component',{props:['message'],template:'{{message}}'});varapp=newVue({el:'#app',data:{}});渲染结果:"嫦娥四号"成功
- Vector和Stack的用法
蟹道人
JavaSejava
/***作者:*日期:*功能:vector的用法*/packagecom.cg;importjava.util.*;publicclassDemo5{publicstaticvoidmain(String[]args){//Vector的使用Vectorvec=newVector();Empemp=newEmp("2011",25,"zhang");vec.add(emp);for(inti=0;
- Python round函数详解
寒秋丶
Python自动化测试性能测试python开发语言测试开发软件开发软件测试自动化测试性能测试
大家好,在Python编程中,经常需要对数字进行舍入操作。无论是在金融领域的货币计算,还是科学计算中的数据处理,都可能需要使用到四舍五入功能。为了满足这一需求,Python提供了一个内置函数round(),它能够方便地对数字进行舍入操作。在本文中,将深入探讨Python中round()函数的用法和特性。将从基本语法开始,逐步深入,讨论该函数在不同情况下的行为,以及如何在实际编程中灵活运用。无论您是
- malloc和new的区别及联系
月夜星辉雪
数据结构
一.区别1.用法上malloc是一个函数,而new是C++一个操作符malloc需要手动计算开辟的空间大小,new后面只需跟上空间的类型,如果有多个对象,加上[]给个数即可malloc申请的空间不能初始化,而new可以malloc返回void*,需要强制类型转换,而new返回对应类型的指针malloc失败会返回空指针,需要手动检查;new失败抛出异常,要用catch捕获2.底层原理上申请自定义类型
- 打开C语言常用内存函数的大门(一) —— memcpy()函数 (内含讲解用法和模拟实现)
埋头编程~
C语言c语言开发语言visualstudio算法
文章目录1.前言2.memcpy函数2.1memcpy函数的原型2.2memcpy函数的形参和返回值详解3.memcpy函数的演示4.memcpy函数的模拟实现5.总结1.前言在之前写的文章中,我介绍了几个比较常用的字符串函数strlen、strcmp、strcpy。它们作用的对象只能是形如字符串类型的数据。那这难免会引起我们心中一泡浓厚的求知欲——C语言有没有给我们提供一些类似于字符串函数的功能
- Java中四种常用的数组复制的方法copyOf(),arraycop(),clone()和copyOfRange()的使用与区别
方九九
java知识点总结java
所谓复制数组,是指将一个数组中的元素在另一个数组中进行复制。本文主要介绍关于Java里面的数组复制(拷贝)的几种方式和用法。在Java中实现数组复制分别有以下4种方法:1.Arrays类的copyOf()方法2.Arrays类的copyOfRange()方法3.System类的arraycopy()方法4.Object类的clone()方法下面来详细介绍这4种方法的使用。使用copyOf()方法和
- Day25_0.1基础学习MATLAB学习小技巧总结(25)——四维图形的可视化
非常规定义M
0.1基础学习MATLAB学习matlab开发语言SIMULINK数学建模
利用空闲时间把碎片化的MATLAB知识重新系统的学习一遍,为了在这个过程中加深印象,也为了能够有所足迹,我会把自己的学习总结发在专栏中,以便学习交流。参考书目:1、《MATLAB基础教程(第三版)(薛山)》2、《MATLABR2020a完全自学一本通》之前的章节都是基础的数据运算用法,对于功课来说更加重要的内容是建模、绘图、观察数据趋势,接下来我会结合自己的使用经验,来为大家分享绘图、建模使用的小
- 【十八】【QT开发应用】标签页QTabWidget的常见用法
妖精七七_
QT开发应用qt开发语言
#include"widget.h"//包含自定义的widget头文件#include//包含QHBoxLayout头文件,用于水平布局#include//包含QTabWidget头文件,用于创建标签页控件#include//包含QDebug头文件,用于调试输出widget::widget(QWidget*parent)//widget类的构造函数:QWidget(parent)//调用基类QWi
- 详解 Pandas 的 isin 用法
文刀小桂
Pandaspandaspython
Pandas的isin()方法可以判断数据值是否在某个数据集合中,若与集合中的某个值相等则返回True,反之返回False。importpandasaspddf=pd.DataFrame({"title":["one","two","three","four"],"type":["small","common","middle","large"],"num":[10,20,30,40]})#1.判
- Linux 中的 route 命令介绍以及使用
XMYX-0
Centos命令使用linux服务器运维route
文章目录路由的基本概念route命令基础用法查看路由表添加路由删除路由添加默认路由删除默认路由route命令示例route命令的替代工具:`iproute`总结在Linux系统中,route命令用于查看和操作路由表。路由表决定了数据包如何在网络中转发和发送。本文将介绍route命令的基本概念、使用方法,并提供一些示例,以帮助更好地理解和使用该命令。路由的基本概念在网络通信中,路由表用于决定网络数据
- 继之前的线程循环加到窗口中运行
3213213333332132
javathreadJFrameJPanel
之前写了有关java线程的循环执行和结束,因为想制作成exe文件,想把执行的效果加到窗口上,所以就结合了JFrame和JPanel写了这个程序,这里直接贴出代码,在窗口上运行的效果下面有附图。
package thread;
import java.awt.Graphics;
import java.text.SimpleDateFormat;
import java.util
- linux 常用命令
BlueSkator
linux命令
1.grep
相信这个命令可以说是大家最常用的命令之一了。尤其是查询生产环境的日志,这个命令绝对是必不可少的。
但之前总是习惯于使用 (grep -n 关键字 文件名 )查出关键字以及该关键字所在的行数,然后再用 (sed -n '100,200p' 文件名),去查出该关键字之后的日志内容。
但其实还有更简便的办法,就是用(grep -B n、-A n、-C n 关键
- php heredoc原文档和nowdoc语法
dcj3sjt126com
PHPheredocnowdoc
<!doctype html>
<html lang="en">
<head>
<meta charset="utf-8">
<title>Current To-Do List</title>
</head>
<body>
<?
- overflow的属性
周华华
JavaScript
<!DOCTYPE html PUBLIC "-//W3C//DTD XHTML 1.0 Transitional//EN" "http://www.w3.org/TR/xhtml1/DTD/xhtml1-transitional.dtd">
<html xmlns="http://www.w3.org/1999/xhtml&q
- 《我所了解的Java》——总体目录
g21121
java
准备用一年左右时间写一个系列的文章《我所了解的Java》,目录及内容会不断完善及调整。
在编写相关内容时难免出现笔误、代码无法执行、名词理解错误等,请大家及时指出,我会第一时间更正。
&n
- [简单]docx4j常用方法小结
53873039oycg
docx
本代码基于docx4j-3.2.0,在office word 2007上测试通过。代码如下:
import java.io.File;
import java.io.FileInputStream;
import ja
- Spring配置学习
云端月影
spring配置
首先来看一个标准的Spring配置文件 applicationContext.xml
<?xml version="1.0" encoding="UTF-8"?>
<beans xmlns="http://www.springframework.org/schema/beans"
xmlns:xsi=&q
- Java新手入门的30个基本概念三
aijuans
java新手java 入门
17.Java中的每一个类都是从Object类扩展而来的。 18.object类中的equal和toString方法。 equal用于测试一个对象是否同另一个对象相等。 toString返回一个代表该对象的字符串,几乎每一个类都会重载该方法,以便返回当前状态的正确表示.(toString 方法是一个很重要的方法) 19.通用编程:任何类类型的所有值都可以同object类性的变量来代替。
- 《2008 IBM Rational 软件开发高峰论坛会议》小记
antonyup_2006
软件测试敏捷开发项目管理IBM活动
我一直想写些总结,用于交流和备忘,然都没提笔,今以一篇参加活动的感受小记开个头,呵呵!
其实参加《2008 IBM Rational 软件开发高峰论坛会议》是9月4号,那天刚好调休.但接着项目颇为忙,所以今天在中秋佳节的假期里整理了下.
参加这次活动是一个朋友给的一个邀请书,才知道有这样的一个活动,虽然现在项目暂时没用到IBM的解决方案,但觉的参与这样一个活动可以拓宽下视野和相关知识.
- PL/SQL的过程编程,异常,声明变量,PL/SQL块
百合不是茶
PL/SQL的过程编程异常PL/SQL块声明变量
PL/SQL;
过程;
符号;
变量;
PL/SQL块;
输出;
异常;
PL/SQL 是过程语言(Procedural Language)与结构化查询语言(SQL)结合而成的编程语言PL/SQL 是对 SQL 的扩展,sql的执行时每次都要写操作
- Mockito(三)--完整功能介绍
bijian1013
持续集成mockito单元测试
mockito官网:http://code.google.com/p/mockito/,打开documentation可以看到官方最新的文档资料。
一.使用mockito验证行为
//首先要import Mockito
import static org.mockito.Mockito.*;
//mo
- 精通Oracle10编程SQL(8)使用复合数据类型
bijian1013
oracle数据库plsql
/*
*使用复合数据类型
*/
--PL/SQL记录
--定义PL/SQL记录
--自定义PL/SQL记录
DECLARE
TYPE emp_record_type IS RECORD(
name emp.ename%TYPE,
salary emp.sal%TYPE,
dno emp.deptno%TYPE
);
emp_
- 【Linux常用命令一】grep命令
bit1129
Linux常用命令
grep命令格式
grep [option] pattern [file-list]
grep命令用于在指定的文件(一个或者多个,file-list)中查找包含模式串(pattern)的行,[option]用于控制grep命令的查找方式。
pattern可以是普通字符串,也可以是正则表达式,当查找的字符串包含正则表达式字符或者特
- mybatis3入门学习笔记
白糖_
sqlibatisqqjdbc配置管理
MyBatis 的前身就是iBatis,是一个数据持久层(ORM)框架。 MyBatis 是支持普通 SQL 查询,存储过程和高级映射的优秀持久层框架。MyBatis对JDBC进行了一次很浅的封装。
以前也学过iBatis,因为MyBatis是iBatis的升级版本,最初以为改动应该不大,实际结果是MyBatis对配置文件进行了一些大的改动,使整个框架更加方便人性化。
- Linux 命令神器:lsof 入门
ronin47
lsof
lsof是系统管理/安全的尤伯工具。我大多数时候用它来从系统获得与网络连接相关的信息,但那只是这个强大而又鲜为人知的应用的第一步。将这个工具称之为lsof真实名副其实,因为它是指“列出打开文件(lists openfiles)”。而有一点要切记,在Unix中一切(包括网络套接口)都是文件。
有趣的是,lsof也是有着最多
- java实现两个大数相加,可能存在溢出。
bylijinnan
java实现
import java.math.BigInteger;
import java.util.regex.Matcher;
import java.util.regex.Pattern;
public class BigIntegerAddition {
/**
* 题目:java实现两个大数相加,可能存在溢出。
* 如123456789 + 987654321
- Kettle学习资料分享,附大神用Kettle的一套流程完成对整个数据库迁移方法
Kai_Ge
Kettle
Kettle学习资料分享
Kettle 3.2 使用说明书
目录
概述..........................................................................................................................................7
1.Kettle 资源库管
- [货币与金融]钢之炼金术士
comsci
金融
自古以来,都有一些人在从事炼金术的工作.........但是很少有成功的
那么随着人类在理论物理和工程物理上面取得的一些突破性进展......
炼金术这个古老
- Toast原来也可以多样化
dai_lm
androidtoast
Style 1: 默认
Toast def = Toast.makeText(this, "default", Toast.LENGTH_SHORT);
def.show();
Style 2: 顶部显示
Toast top = Toast.makeText(this, "top", Toast.LENGTH_SHORT);
t
- java数据计算的几种解决方法3
datamachine
javahadoopibatisr-languer
4、iBatis
简单敏捷因此强大的数据计算层。和Hibernate不同,它鼓励写SQL,所以学习成本最低。同时它用最小的代价实现了计算脚本和JAVA代码的解耦,只用20%的代价就实现了hibernate 80%的功能,没实现的20%是计算脚本和数据库的解耦。
复杂计算环境是它的弱项,比如:分布式计算、复杂计算、非数据
- 向网页中插入透明Flash的方法和技巧
dcj3sjt126com
htmlWebFlash
将
Flash 作品插入网页的时候,我们有时候会需要将它设为透明,有时候我们需要在Flash的背面插入一些漂亮的图片,搭配出漂亮的效果……下面我们介绍一些将Flash插入网页中的一些透明的设置技巧。
一、Swf透明、无坐标控制 首先教大家最简单的插入Flash的代码,透明,无坐标控制: 注意wmode="transparent"是控制Flash是否透明
- ios UICollectionView的使用
dcj3sjt126com
UICollectionView的使用有两种方法,一种是继承UICollectionViewController,这个Controller会自带一个UICollectionView;另外一种是作为一个视图放在普通的UIViewController里面。
个人更喜欢第二种。下面采用第二种方式简单介绍一下UICollectionView的使用。
1.UIViewController实现委托,代码如
- Eos平台java公共逻辑
蕃薯耀
Eos平台java公共逻辑Eos平台java公共逻辑
Eos平台java公共逻辑
>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>>
蕃薯耀 2015年6月1日 17:20:4
- SpringMVC4零配置--Web上下文配置【MvcConfig】
hanqunfeng
springmvc4
与SpringSecurity的配置类似,spring同样为我们提供了一个实现类WebMvcConfigurationSupport和一个注解@EnableWebMvc以帮助我们减少bean的声明。
applicationContext-MvcConfig.xml
<!-- 启用注解,并定义组件查找规则 ,mvc层只负责扫描@Controller -->
<
- 解决ie和其他浏览器poi下载excel文件名乱码
jackyrong
Excel
使用poi,做传统的excel导出,然后想在浏览器中,让用户选择另存为,保存用户下载的xls文件,这个时候,可能的是在ie下出现乱码(ie,9,10,11),但在firefox,chrome下没乱码,
因此必须综合判断,编写一个工具类:
/**
*
* @Title: pro
- 挥洒泪水的青春
lampcy
编程生活程序员
2015年2月28日,我辞职了,离开了相处一年的触控,转过身--挥洒掉泪水,毅然来到了兄弟连,背负着许多的不解、质疑——”你一个零基础、脑子又不聪明的人,还敢跨行业,选择Unity3D?“,”真是不自量力••••••“,”真是初生牛犊不怕虎•••••“,••••••我只是淡淡一笑,拎着行李----坐上了通向挥洒泪水的青春之地——兄弟连!
这就是我青春的分割线,不后悔,只会去用泪水浇灌——已经来到
- 稳增长之中国股市两点意见-----严控做空,建立涨跌停版停牌重组机制
nannan408
对于股市,我们国家的监管还是有点拼的,但始终拼不过飞流直下的恐慌,为什么呢?
笔者首先支持股市的监管。对于股市越管越荡的现象,笔者认为首先是做空力量超过了股市自身的升力,并且对于跌停停牌重组的快速反应还没建立好,上市公司对于股价下跌没有很好的利好支撑。
我们来看美国和香港是怎么应对股灾的。美国是靠禁止重要股票做空,在
- 动态设置iframe高度(iframe高度自适应)
Rainbow702
JavaScriptiframecontentDocument高度自适应局部刷新
如果需要对画面中的部分区域作局部刷新,大家可能都会想到使用ajax。
但有些情况下,须使用在页面中嵌入一个iframe来作局部刷新。
对于使用iframe的情况,发现有一个问题,就是iframe中的页面的高度可能会很高,但是外面页面并不会被iframe内部页面给撑开,如下面的结构:
<div id="content">
<div id=&quo
- 用Rapael做图表
tntxia
rap
function drawReport(paper,attr,data){
var width = attr.width;
var height = attr.height;
var max = 0;
&nbs
- HTML5 bootstrap2网页兼容(支持IE10以下)
xiaoluode
html5bootstrap
<!DOCTYPE html>
<html>
<head lang="zh-CN">
<meta charset="UTF-8">
<meta http-equiv="X-UA-Compatible" content="IE=edge">