09 拷贝数变异分析(GATK流程)
09 拷贝数变异分析(GATK流程)
我们已经分析了 Somatic mutations,并进行了注释和可视化,接下来我们进行拷贝数变异的分析。
这里我们还是先从 GATK 的 somatic cnv 的最佳实践开始
拷 贝数变异(copy number variations, CNVs)是属于基因组结构变异(structural variation, SV),是指 DNA 片 段长度在 1Kb-3Mb 的基因组结构变异。我们首先从 GATK 的 CNV 流程开始 CNV 的分析。
首先是流程图,先从 bam 文件,结合坐标文件计算每个外显子的 reads counts 数,然后 call segment,最后是画图:
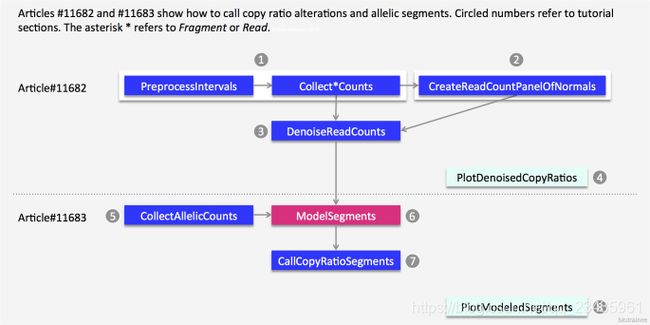
在下面这个链接中,给出了详细的教程:https://gatkforums.broadinstitute.org/gatk/discussion/11682#2
或者最近更新的教程,一样的:https://gatk.broadinstitute.org/hc/en-us/articles/360035531092
1. 外显子坐标的interval文件
interval 文件其实也是一个坐标文件,类似 bed,只不过 bed 文件的坐标是从 0 开始记录,而 interval 文件的坐标是从1开始记录。使用基因组 interval 文件可以定义软件分析的分辨率。如果是全基因组测序,interval 文件就用全基因组坐标的等间隔区间就好。对于外显子组的数据,我们使用捕获试剂盒的目标区域,理论上应该返回原文查找对应试剂盒,去搜索其捕获外显子的区域,一般是外显子侧翼上下游 250bp 以内。我懒得去查,就用软件的默认参数 250bp。
首先使用 BedToIntervalList 工具将 bed 转成 interval 格式(其实前面已经完成),然后用PreprocessIntervals工具获取 target 区间,即外显子侧翼上下游 250bp。
GATK=~/wes_cancer/biosoft/gatk-4.1.4.1/gatk
ref=~/wes_cancer/data/Homo_sapiens_assembly38.fasta
bed=~/wes_cancer/data/hg38.exon.bed
dict=~/wes_cancer/data/Homo_sapiens_assembly38.dict
ref=~/wes_cancer/data/Homo_sapiens_assembly38.fasta
## bed to intervals_list
$GATK BedToIntervalList -I ${bed} -O ~/wes_cancer/data/hg38.exon.interval_list -SD ${dict}
## Preprocess Intervals
$GATK PreprocessIntervals \
-L ~/wes_cancer/data/hg38.exon.interval_list \
--sequence-dictionary ${dict} \
--reference ${ref} \
--padding 250 \
--bin-length 0 \
--interval-merging-rule OVERLAPPING_ONLY \
--output ~/wes_cancer/data/targets.preprocessed.interval.list
2. 获取样本的read counts
这里分为两小步进行:
- 首先是获取所有样本的 read counts,用到了工具是
CollectReadCounts, 其根据所提供的 interval 文件,对 bam 文件进行 reads 计数,可以简单理解为把 bam 文件转换成 interval 区间的 reads 数。最后会生成一个 HDF5 格式的文件,需要用第三方软件 HDFView 来查看,这里不做展示。这个文件记录了每个基因组interval 文件的 CONTIG,START,END 和原始 COUNT 值,并制成表格。 - 然后构建正常样本的
CNV panel of normals,生成正常样本的cnvponM.pon.hdf5文件。对于外显子组捕获测序数据,捕获过程会引入一定的的噪音。因此后面需要降噪,该文件就是用于后面第 3 步DenoiseReadCounts。
实际使用到的脚本如下:
interval=~/wes_cancer/data/targets.preprocessed.interval.list
GATK=~/wes_cancer/biosoft/gatk-4.1.4.1/gatk
ref=~/wes_cancer/data/Homo_sapiens_assembly38.fasta
cat config3 | while read id
do
i=./5.gatk/${id}_bqsr.bam
echo ${i}
## step1 : CollectReadCounts
time $GATK --java-options "-Xmx20G -Djava.io.tmpdir=./" CollectReadCounts \
-I ${i} \
-L ${interval} \
-R ${ref} \
--format HDF5 \
--interval-merging-rule OVERLAPPING_ONLY \
--output ./8.cnv/gatk/counts/${id}.clean_counts.hdf5
## step2 : Generate a CNV panel of normals:cnvponM.pon.hdf5
$GATK --java-options "-Xmx20G -Djava.io.tmpdir=./" CreateReadCountPanelOfNormals \
--minimum-interval-median-percentile 5.0 \
--output ./8.cnv/gatk/cnvponM.pon.hdf5 \
--input ./8.cnv/gatk/counts/case1_germline.clean_counts.hdf5 \
--input ./8.cnv/gatk/counts/case2_germline.clean_counts.hdf5 \
--input ./8.cnv/gatk/counts/case3_germline.clean_counts.hdf5 \
--input ./8.cnv/gatk/counts/case4_germline.clean_counts.hdf5 \
--input ./8.cnv/gatk/counts/case5_germline.clean_counts.hdf5 \
--input ./8.cnv/gatk/counts/case6_germline.clean_counts.hdf5
done
这里的 input 重复了 6 次,为的是想让初学者容易理解。如果追求代码整洁,可以把这 6 句改成
$(for i in {
1..6} ;do echo "--input ./8.cnv/gatk/counts/case${i}_germline.clean_counts.hdf5" ;done)
3. 降噪DenoiseReadCounts
该步骤用到的工具是 DenoiseReadCounts,主要是做了一个标准化和降噪,会生成两个文件 ${id}.clean.standardizedCR.tsv 和 ${id}.clean.denoisedCR.tsv,该工具会根据 PoN 的 counts 中位数对输入文件 ${id}.clean_counts.hdf5 进行一个标准化,包括 log2 转换。然后使用 PoN 的主成分进行标准化后的 copy ratios 降噪。实际上这里只需要对 tumor 样本进行降噪的,为了比较,我就把 tumor 和 normal 样本都分析了一遍,后面可以做个对比。
GATK=~/wes_cancer/biosoft/gatk-4.1.4.1/gatk
cat config3 | while read id
do
i=./8.cnv/gatk/counts/${id}.clean_counts.hdf5
$GATK --java-options "-Xmx20g" DenoiseReadCounts \
-I ${i} \
--count-panel-of-normals ./8.cnv/gatk/cnvponM.pon.hdf5 \
--standardized-copy-ratios ./8.cnv/gatk/standardizedCR/${id}.clean.standardizedCR.tsv \
--denoised-copy-ratios ./8.cnv/gatk/denoisedCR/${id}.clean.denoisedCR.tsv
done
4. 可视化降噪后的copy ratios
我们使用 PlotDenoisedCopyRatios 可视化标准化和去噪的 read counts。这些图可以直观地评估去噪的效果。
GATK=~/wes_cancer/biosoft/gatk-4.1.4.1/gatk
dict=~/wes_cancer/data/Homo_sapiens_assembly38.dict
cat config3 | while read id
do
$GATK --java-options "-Xmx20g" PlotDenoisedCopyRatios \
--standardized-copy-ratios ./8.cnv/gatk/standardizedCR/${id}.clean.standardizedCR.tsv \
--denoised-copy-ratios ./8.cnv/gatk/denoisedCR/${id}.clean.denoisedCR.tsv \
--sequence-dictionary ${dict} \
--output ./8.cnv/gatk/cnv_plots \
--output-prefix ${id}
done
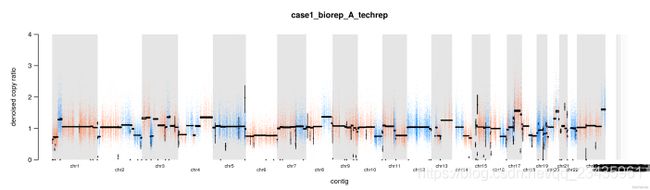
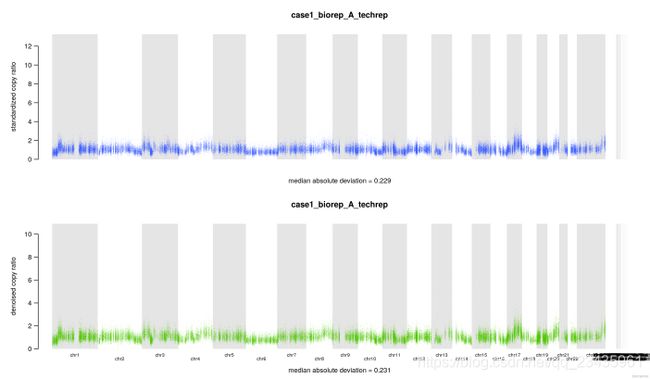
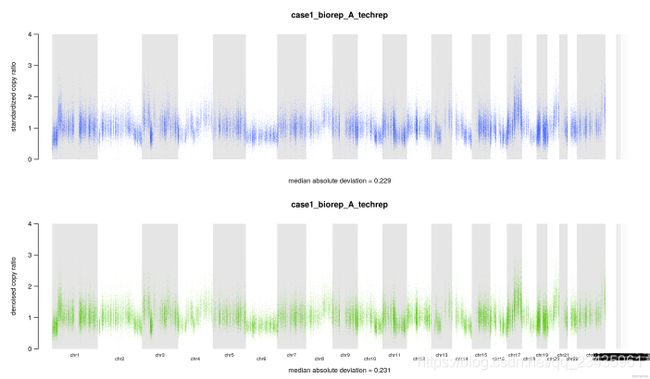
对于每一个样本,如 case1_biorep_A_techrep,将生成 6 个文件:
./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.denoised.png
./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.denoisedLimit4.png
./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.deltaMAD.txt
./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.scaledDeltaMAD.txt
./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.standardizedMAD.txt
./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.denoisedMAD.txt
其中4个文件是文本文件,里面就是一个数字,记录几个拷贝数变化比值 copy ratio 的中位数绝对偏差(median absolute deviation, MAD)
## 标准化后的 copy ratios 的 MAD
$ cat ./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.standardizedMAD.txt
0.229
## 降噪后的 copy ratios 的 MAD
$ cat ./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.denoisedMAD.txt
0.231
## 标准化后的 MAD 和降噪后的 MAD 的差
$ cat ./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.deltaMAD.txt
-0.002
## (降噪后的 MAD - 标准化后的 MAD ) / (标准化后的 MAD )
$ cat ./8.cnv/gatk/cnv_plots/case1_biorep_A_techrep.scaledDeltaMAD.txt
-0.01
另 外两个文件是图片,表达同一个意思,标准化和降噪后的 copy ratios,底下还有中位数绝对偏差(median absolute deviation, MAD),只不过一张图片把 y 轴设置为 0 到 4。假如 tumor 样本的拷贝数没有发生变化,copy ratio 应该稳定在 1 附近。当然,要是发生了 CNV 事件,那应该就在 1 附近波动。
5. 计算常见的germline mutation位点
这一步用到了 CollectAllelicCounts 工具,对输入的 bam 文件,根据指定的 interval 区间,进行 germline mutation 的检测(仅仅是 SNPs 位点,不包括 INDELs ),并计算该位点覆盖的 reads 数,即该位点的测序深度。值得注意的是,该工具一个默认参数是 MAPQ 值大于 20 的 reads 才会被纳入计数,最后生成一个 tsv 文件。
GENOME=~/wes_cancer/data/Homo_sapiens_assembly38.fasta
GATK=~/wes_cancer/biosoft/gatk-4.1.4.1/gatk
interval=~/wes_cancer/data/targets.preprocessed.interval.list
cat config3 | while read id
do
i=./5.gatk/${id}_bqsr.bam
echo ${i}
time $GATK --java-options "-Xmx20G -Djava.io.tmpdir=./" CollectAllelicCounts \
-I ${i} \
-L ${interval} \
-R ${GENOME} \
-O ./8.cnv/gatk/allelicCounts/${id}.allelicCounts.tsv
done
生成的 tsv 文件主要内容如下(这里我过滤掉了第六列为 N 的位点):
$ less ./8.cnv/gatk/allelicCounts/case1_biorep_A_techrep.allelicCounts.tsv | grep -v ^@ | awk '{if($6 != "N") print $0}' |less
CONTIG POSITION REF_COUNT ALT_COUNT REF_NUCLEOTIDE ALT_NUCLEOTIDE
chr1 925873 34 1 G T
chr1 925918 71 1 G T
chr1 925938 92 1 G T
chr1 925953 92 1 G T
chr1 925974 96 1 G T
chr1 925979 102 2 G A
6. ModelSegments
在第 3 步我们拿到了标准化和降噪后的两个 tsv 文件,记录了某个区间的 LOG2_COPY_RATIO 值,内容大致如下:
$ less ./8.cnv/gatk/denoisedCR/case1_biorep_A_techrep.clean.denoisedCR.tsv | grep -v ^@| less
CONTIG START END LOG2_COPY_RATIO
chr1 925692 926262 -1.178123
chr1 929905 930585 -0.569447
chr1 930789 931338 -0.686712
chr1 935522 936145 -0.404623
chr1 938790 939201 -0.727205
chr1 939202 939709 -0.882516
而第 5 步拿到的记录等位基因测序深度的 tsv 文件已经在上面展示过了。接下来第 6 步将利用这两个结果进行 call segment,需要注意的是输入文件要求 tumor match normal。不过好像也可以不输入第 5 步 CollectAllelicCounts 的结果,等有时间再比较一下两者的区别吧。这一步用到的工具是ModelSegments,它根据去噪后的第三步的 reads counts 值对 copy ratios 进行分割,并根据第五步的CollectAllelicCounts等位基因计数对分割片段进行分类。代码如下:
GATK=/~/wec_cancer/biosoft/gatk-4.1.4.1/gatk
cat config3 | while read id
do
germline=${id:0:5}_germline
## ModelSegments
$GATK --java-options "-Xmx20g" ModelSegments \
--denoised-copy-ratios ./8.cnv/gatk/denoisedCR/${id}.clean.denoisedCR.tsv \
--allelic-counts ./8.cnv/gatk/allelicCounts/${id}.allelicCounts.tsv \
--normal-allelic-counts ./8.cnv/gatk/allelicCounts/${germline}.allelicCounts.tsv \
--output ./8.cnv/gatk/segments \
--output-prefix ${id}
done
生成的文件有点多,每个样本生成 11 个文件。 param 文件包含用于 copy ratios(cr)和 allele fractions(af)的全局参数,而 seg 文件包含有关片段的数据。 具体说明可以查看这个链接:https://software.broadinstitute.org/gatk/documentation/tooldocs/4.1.4.0/org_broadinstitute_hellbender_tools_copynumber_ModelSegments.php
./8.cnv/gatk/segments/case1_biorep_A_techrep.af.igv.seg
./8.cnv/gatk/segments/case1_biorep_A_techrep.cr.igv.seg
./8.cnv/gatk/segments/case1_biorep_A_techrep.cr.seg
./8.cnv/gatk/segments/case1_biorep_A_techrep.modelBegin.af.param
./8.cnv/gatk/segments/case1_biorep_A_techrep.modelBegin.cr.param
./8.cnv/gatk/segments/case1_biorep_A_techrep.modelBegin.seg
./8.cnv/gatk/segments/case1_biorep_A_techrep.modelFinal.af.param
./8.cnv/gatk/segments/case1_biorep_A_techrep.modelFinal.cr.param
./8.cnv/gatk/segments/case1_biorep_A_techrep.modelFinal.seg
./8.cnv/gatk/segments/case1_biorep_A_techrep.hets.normal.tsv
./8.cnv/gatk/segments/case1_biorep_A_techrep.hets.tsv
其不过上面拿到的文件中的 ${id}*.igv.seg 文件,可以直接载入到 IGV 中进行可视化,如:
(不知道为什么,case4_biorep_B_techrep和case4_techrep_2的CNV事件是碎片化的,而 case5 病人的 X 染色体直接缺失)

7. CallCopyRatioSegments
这一步用来判断 copy ratio segments 是扩增、缺失、还是正常的可能性。对上一步拿到的${id}.cr.seg进行推断,得到${id}.clean.called.seg文件会增加一列 CALL,用 +、-、0分别表示扩增、缺失和正常。基本上MEAN_LOG2_COPY_RATIO大于 0.14 就是扩增,小于 -0.15 就是缺失,其他的为正常。
GATK=~/wes_cancer/biosoft/gatk-4.1.4.1/gatk
cat config3 | while read id
do
$GATK --java-options "-Xmx20g" CallCopyRatioSegments \
-I ./8.cnv/gatk/segments/${id}.cr.seg \
-O ./8.cnv/gatk/segments/${id}.clean.called.seg
done
8. 可视化CNV结果
通过上面的分析,我们拿到了最后建模的 copy ratios 和 allele fractions segment,接下来用一个工具进行可视化:PlotModeledSegments
dict=~/wes_cancer/data/Homo_sapiens_assembly38.dict
GATK=~/wes_cancer/biosoft/gatk-4.1.4.1/gatk
cat config3 | while read id
do
$GATK --java-options "-Xmx20g" PlotModeledSegments \
--denoised-copy-ratios ./8.cnv/gatk/denoisedCR/${id}.clean.denoisedCR.tsv \
--allelic-counts ./8.cnv/gatk/segments/${id}.hets.tsv \
--segments ./8.cnv/gatk/segments/${id}.modelFinal.seg \
--sequence-dictionary ${dict} \
--output ./8.cnv/gatk/cnv_plots \
--output-prefix ${id}.clean
done
整个gatk cnv流程
上面整个流程的代码,其实可以合并为一个脚本 gatk_cnv.sh:
GENOME=~/wes_cancer/data/Homo_sapiens_assembly38.fasta
dict=~/wes_cancer/data/Homo_sapiens_assembly38.dict
INDEX=~/wes_cancer/data/bwa_index/gatk_hg38
GATK=~/wes_cancer/biosoft/gatk-4.1.4.1/gatk
DBSNP=~/wes_cancer/data/dbsnp_146.hg38.vcf.gz
kgSNP=~/wes_cancer/data/1000G_phase1.snps.high_confidence.hg38.vcf.gz
kgINDEL=~/wes_cancer/data/Mills_and_1000G_gold_standard.indels.hg38.vcf.gz
interval=~/wes_cancer/data/targets.preprocessed.interval.list
cd ~/wes_cancer/project/8.cnv/gatk
###################################
#### 把bam文件转为外显子reads数 ######
###################################
cat ~/wes_cancer/project/config3 | while read id
do
i=~/wes_cancer/project/5.gatk/${id}_bqsr.bam
echo ${i}
## step1 : CollectReadCounts
time $GATK --java-options "-Xmx20G -Djava.io.tmpdir=./" CollectReadCounts \
-I ${i} \
-L ${interval} \
-R ${GENOME} \
--format HDF5 \
--interval-merging-rule OVERLAPPING_ONLY \
--output ${id}.clean_counts.hdf5
## step2 : CollectAllelicCounts
time $GATK --java-options "-Xmx20G -Djava.io.tmpdir=./" CollectAllelicCounts \
-I ${i} \
-L ${interval} \
-R ${GENOME} \
-O ${id}.allelicCounts.tsv
done
### 注意这个CollectAllelicCounts步骤非常耗时,而且占空间
mkdir allelicCounts
mv *.allelicCounts.tsv ./allelicCounts
mkdir counts
mv *.clean_counts.hdf5 ./counts
##################################################
# 接着合并所有的normal样本的数据创建 cnvponM.pon.hdf5 #
##################################################
$GATK --java-options "-Xmx20g" CreateReadCountPanelOfNormals \
--minimum-interval-median-percentile 5.0 \
--output cnvponM.pon.hdf5 \
--input counts/case1_germline.clean_counts.hdf5 \
--input counts/case2_germline.clean_counts.hdf5 \
--input counts/case3_germline.clean_counts.hdf5 \
--input counts/case4_germline.clean_counts.hdf5 \
--input counts/case5_germline.clean_counts.hdf5 \
--input counts/case6_germline.clean_counts.hdf5
############################################
############# 最后走真正的CNV流程 #############
############################################
cat config | while read id
do
i=./counts/${id}.clean_counts.hdf5
$GATK --java-options "-Xmx20g" DenoiseReadCounts \
-I $i \
--count-panel-of-normals cnvponM.pon.hdf5 \
--standardized-copy-ratios ${id}.clean.standardizedCR.tsv \
--denoised-copy-ratios ${id}.clean.denoisedCR.tsv
done
mkdir denoisedCR standardizedCR segments cnv_plots
mv *denoisedCR.tsv ./denoisedCR
mv *standardizedCR.tsv ./standardizedCR
cat config | while read id
do
i=./denoisedCR/${id}.clean.denoisedCR.tsv
## ModelSegments的时候有两个策略,是否利用CollectAllelicCounts的结果
$GATK --java-options "-Xmx20g" ModelSegments \
--denoised-copy-ratios $i \
--output segments \
--output-prefix ${id}
## 如果要利用CollectAllelicCounts的结果就需要增加两个参数,这里就不讲解了。
$GATK --java-options "-Xmx20g" CallCopyRatioSegments \
-I segments/${id}.cr.seg \
-O segments/${id}.clean.called.seg
## 这里面有两个绘图函数,PlotDenoisedCopyRatios 和 PlotModeledSegments ,可以选择性运行。
$GATK --java-options "-Xmx20g" PlotDenoisedCopyRatios \
--standardized-copy-ratios ./standardizedCR/${id}.clean.standardizedCR.tsv \
--denoised-copy-ratios $i \
--sequence-dictionary ${dict} \
--output cnv_plots \
--output-prefix ${id}
$GATK --java-options "-Xmx20g" PlotModeledSegments \
--denoised-copy-ratios $i \
--segments segments/${id}.modelFinal.seg \
--sequence-dictionary ${dict} \
--output cnv_plots \
--output-prefix ${id}.clean
done
对于每一个样本,就会拿到拷贝数变异的结果,如: