scikit-learn点滴
scikit-learn点滴
scikit-learn是非常漂亮的一个机器学习库,在某些时候,使用这些库能够大量的节省你的时间,至少,我们用python,应该是很难写出速度快如斯的代码的.
scikit-learn官方出了一些文档,但是个人觉得,它的文档很多东西都没有讲清楚,它说算法原理的时候,只是描述一下,除非你对这种算法已经烂熟于心,才会对它的描述会心一笑,它描述API的时候,很多时候只是讲了一些常见用法,一些比较高级的用法就语焉不详,虽然有很多人说,这玩意的文档写得不错,但是我觉得特坑.所以这篇博文,会记录一些我使用这个库的时候碰到的一些坑,以及如何跨过这些坑.慢慢来更新吧,当然,以后如果不用了,文章估计也不会更新了,当然,我也没有打算说,这篇文章有多少人能看.就这样吧.
聚类
坑1: 如何自定义距离函数?
虽然说scikit-learn这个库实现了很多的聚类函数,但是这些算法使用的距离大部分都是欧氏距离或者明科夫斯基距离,事实上,根据我们教材上的描述,所谓的距离,可不单单仅有这两种,为了不同的目的,我们可以用不同的距离来度量两个向量之间的距离,但是很遗憾,我并没有看见scikit-learn中提供自定义距离的选项,网上搜了一大圈也没有见到.
但是不用担心,我们可以间接实现这个东西.以DBSCAN算法为例,下面是类的一个构造函数:
class sklearn.cluster.DBSCAN(eps=0.5, min_samples=5, metric='euclidean', algorithm='auto', leaf_size=30, p=None, n_jobs=1)
# eps表示两个向量可以被视作为同一个类的最大的距离
# min_samples表示一个类中至少要包含的元素数量,如果小于这个数量,那么不构成一个类我们要特别注意一下metric这个选项,我们来看一下选项:
metric : string, or callable
The metric to use when calculating distance between instances in a feature array. If metric is a string or callable, it must be one of the options allowed by metrics.pairwise.calculate_distance for its metric parameter.
If metric is “precomputed”, X is assumed to be a distance matrix and must be square. X may be a sparse matrix, in which case only “nonzero” elements may be considered neighbors for DBSCAN.
New in version 0.17: metric precomputed to accept precomputed sparse matrix.这段描述其实透露了一个很重要的信息,那就是其实你可以自己提前计算各个向量的相似度,构成一个相似度的矩阵,只要你设置metric='precomputedd'就行,那么如何调用呢?
我们来看一下fit函数.
fit(X, y=None, sample_weight=None)
# X : array or sparse (CSR) matrix of shape (n_samples, n_features), or array of shape (n_samples, n_samples)
# A feature array, or array of distances between samples if metric='precomputed'.上面的注释是什么意思呢,我翻译一下,如果你将metric设置成了precomputed的话,那么传入的X参数应该为各个向量之间的相似度矩阵,然后fit函数会直接用你这个矩阵来进行计算.否则的话,你还是要乖乖地传入(n_samples, n_features)形式的向量.
这意味着什么,同志们.这意味着我们可以用我们自定义的距离事先计算好各个向量的相似度,然后调用这个函数来获得结果,是不是很爽.
具体怎么来编程,我给个例子,抛个砖.
import numpy as np
from sklearn.cluster import DBSCAN
if __name__ == '__main__':
Y = np.array([[0, 1, 2],
[1, 0, 3],
[2, 3, 0]]) # 相似度矩阵,距离越小代表两个向量距离越近
# N = Y.shape[0]
db = DBSCAN(eps=0.13, metric='precomputed', min_samples=3).fit(Y)
labels = db.labels_
# 然后来看一下分类的结果吧!
n_clusters_ = len(set(labels)) - (1 if -1 in labels else 0) # 类的数目
print('类的数目是:%d'%(n_clusters_))我们继续来看一下AP聚类,其实也很类似:
class sklearn.cluster.AffinityPropagation(damping=0.5, max_iter=200, convergence_iter=15, copy=True, preference=None, affinity='euclidean', verbose=False)关键在这个affinity参数上:
affinity : string, optional, default=``euclidean``
Which affinity to use. At the moment precomputed and euclidean are supported. euclidean uses the negative squared euclidean distance between points.这个东西也支持precomputed参数.再来看一下fit函数:
fit(X, y=None)
# Create affinity matrix from negative euclidean distances, then apply affinity propagation clustering.
# Parameters:
# X: array-like, shape (n_samples, n_features) or (n_samples, n_samples) :
# Data matrix or, if affinity is precomputed, matrix of similarities / affinities.这里的X和前面是类似的,如果你将metric设置成了precomputed的话,那么传入的X参数应该为各个向量之间的相似度矩阵,然后fit函数会直接用你这个矩阵来进行计算.否则的话,你还是要乖乖地传入(n_samples, n_features)形式的向量.
例子1
"""
目标:
~~~~~~~~~~~~~~~~
在这个文件里面,我最想测试一下的是,我前面的那些聚类算法是否是正确的.
首先要测试的是AP聚类.
"""
from sklearn.cluster import AffinityPropagation
from sklearn import metrics
from sklearn.datasets.samples_generator import make_blobs
from sklearn.metrics.pairwise import euclidean_distances
import matplotlib.pyplot as plt
from itertools import cycle
def draw_pic(n_clusters, cluster_centers_indices, labels, X):
''' 口说无凭,绘制一张图就一目了然. '''
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
for k, col in zip(range(n_clusters), colors):
class_members = labels == k
cluster_center = X[cluster_centers_indices[k]] # 得到聚类的中心
plt.plot(X[class_members, 0], X[class_members, 1], col + '.')
plt.plot(cluster_center[0], cluster_center[1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=14)
for x in X[class_members]:
plt.plot([cluster_center[0], x[0]], [cluster_center[1], x[1]], col)
plt.title('Estimated number of clusters: %d' % n_clusters)
plt.show()
if __name__ == '__main__':
centers = [[1, 1], [-1, -1], [1, -1]]
# 接下来要生成300个点,并且每个点属于哪一个中心都要标记下来,记录到labels_true中.
X, labels_true = make_blobs(n_samples=300, centers=centers,
cluster_std=0.5, random_state=0)
af = AffinityPropagation(preference=-50).fit(X) # 开始用AP聚类
cluster_centers_indices = af.cluster_centers_indices_ # 得到聚类的中心点
labels = af.labels_ # 得到label
n_clusters = len(cluster_centers_indices) # 类的数目
draw_pic(n_clusters, cluster_centers_indices, labels, X)
#===========接下来的话提前计算好距离=================#
distance_matrix = -euclidean_distances(X, squared=True) # 提前计算好欧几里德距离,需要注意的是,这里使用的是欧几里德距离的平方
af1 = AffinityPropagation(affinity='precomputed', preference=-50).fit(distance_matrix)
cluster_centers_indices1 = af1.cluster_centers_indices_ # 得到聚类的中心
labels1 = af1.labels_ # 得到label
n_clusters1 = len(cluster_centers_indices1) # 类的数目
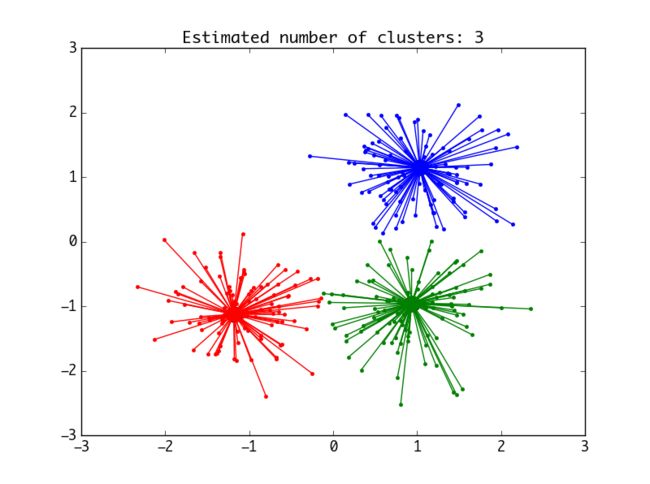
draw_pic(n_clusters1, cluster_centers_indices1, labels1, X)两种方法都将产生这样的图:
例子2
既然都到这里了,我们索性来测试一下DBSCAN算法好了.
"""
目标:
~~~~~~~~~~~~~~
前面已经测试过了ap聚类,接下来测试DBSACN.
"""
import numpy as np
from sklearn.cluster import DBSCAN
from sklearn import metrics
from sklearn.datasets.samples_generator import make_blobs
from sklearn.preprocessing import StandardScaler
import matplotlib.pyplot as plt
from sklearn.metrics.pairwise import euclidean_distances
def draw_pic(n_clusters, core_samples_mask, labels, X):
''' 开始绘制图片 '''
# Black removed and is used for noise instead.
unique_labels = set(labels)
colors = plt.cm.Spectral(np.linspace(0, 1, len(unique_labels)))
for k, col in zip(unique_labels, colors):
if k == -1:
# Black used for noise.
col = 'k'
class_member_mask = (labels == k)
xy = X[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=14)
xy = X[class_member_mask & ~core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=6)
plt.title('Estimated number of clusters: %d' % n_clusters)
plt.show()
if __name__ == '__main__':
#=========首先产生数据===========#
centers = [[1, 1], [-1, -1], [1, -1]]
X, labels_true = make_blobs(n_samples=750, centers=centers,
cluster_std=0.4, random_state=0)
X = StandardScaler().fit_transform(X)
#=========接下来开始聚类==========#
db = DBSCAN(eps=0.3, min_samples=10).fit(X)
labels = db.labels_ # 每个点的标签
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
core_samples_mask[db.core_sample_indices_] = True
n_clusters = len(set(labels)) - (1 if -1 in labels else 0) # 类的数目
draw_pic(n_clusters, core_samples_mask, labels, X)
#==========接下来我们提前计算好距离============#
distance_matrix = euclidean_distances(X)
db1 = DBSCAN(eps=0.3, min_samples=10, metric='precomputed').fit(distance_matrix)
labels1 = db1.labels_ # 每个点的标签
core_samples_mask1 = np.zeros_like(db1.labels_, dtype=bool)
core_samples_mask1[db1.core_sample_indices_] = True
n_clusters1 = len(set(labels1)) - (1 if -1 in labels1 else 0) # 类的数目
draw_pic(n_clusters1, core_samples_mask1, labels1, X)两种方法都将产生这样的图:
好吧,暂时介绍到这里吧,但是有意思的是,最简单的KMeans算法倒是不支持这样的干活.