作者|Kenichi Nakanishi
编译|VK
来源|Towards Data Science
我有一个爱买植物的未婚妻,还有一只爱啃植物的猫——我想,有什么比把一个能告诉我植物是否安全的分类器更好呢!
需要注意的一点是,这里所做的所有工作都是在google colabs上完成的,使用的notebook可以在我的Github上找到:https://github.com/kenichinak...
步骤1-获取数据
不幸的是,我找不到一个适合我在Kaggle上或使用Google的数据集搜索的预先制作的图像数据集。所以,我准备建立我自己的!
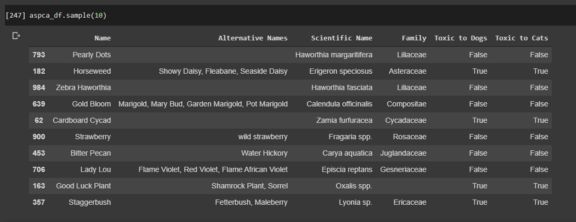
我决定使用ASPCA的《猫和狗的植物毒性清单》,我已经用了好几次了。这给了我们一个很好的核心工作。为了从网站上获取这些文本数据,我们可以求助于BeautifulSoup,这是一个Python库,用于从HTML和XML文件中提取数据。
from urllib.request import Request, urlopen
from bs4 import BeautifulSoup
def getHTMLContent(link):
html = urlopen(link)
soup = BeautifulSoup(html, 'html.parser')
return soup然而,当查看他们的网站时,该表并不是一个易于访问的html表,而是将数据存储为面板中的行。幸运的是,beauthulsoup为我们提供了一种简单的方法来搜索解析树,以找到我们想要的数据。例如:
req = Request('https://www.aspca.org/pet-care/animal-poison-control/cats-plant-list', headers={'User-Agent': 'Mozilla/5.0'})
webpage = urlopen(req).read()
# 爬取数据
soup = BeautifulSoup(webpage, 'lxml')
# 搜索解析树以从表中获得所有内容
content_list = soup.find_all('span')[7:-4]
# 将其放入一个dataframe中进行进一步处理
df_cats = pd.DataFrame(content_list) 在收集完原始数据后,我们需要将其分为多个列,并进行一些拆分:
# 清理字符串
df_cats[0] = df_cats[0].apply(lambda x: str(x).split('>')[1][:-3])
df_cats[4] = df_cats[4].apply(lambda x: str(x).split('>')[1][:-3])
df_cats[1] = df_cats[1].apply(lambda x: str(x).split('(')[1][0:-4])
# 删除无用的列并重命名列
df_cats = df_cats.drop(columns=[2,3,5,6]).rename(columns = {0:'Name',1:'Alternative Names',4:'Scientific Name',7:'Family'})
# 将有毒和无毒植物分开
df_cats['Toxic to Cats'] = True
first_nontoxic_cats = [index for index in df_cats[df_cats['Name'].str.startswith('A')].index if index>100][0]
df_cats.loc[first_nontoxic_cats:,'Toxic to Cats'] = False然后,我们可以对特定于狗的列表重复此过程,然后合并数据帧并清理nan:
# 合并数据框架到一个,用于保留只存在于一边的值
df_catsdogs = df_dogs.merge(df_cats, how='outer', on=['Name','Alternative Names','Scientific Name','Family'])
df_catsdogs = df_catsdogs.fillna('Unknown')
aspca_df = df_catsdogs.copy()
# 假设对猫和狗有相同的毒性
aspca_df['Toxic to Cats'] = aspca_df.apply(lambda x: x['Toxic to Dogs'] if (x['Toxic to Cats'] == 'Unknown') else x['Toxic to Cats'], axis=1)
aspca_df['Toxic to Dogs'] = aspca_df.apply(lambda x: x['Toxic to Cats'] if (x['Toxic to Dogs'] == 'Unknown') else x['Toxic to Dogs'], axis=1)步骤2-浅度清理
接下来,我们可以开始进行浅度清理,包括查看数据集,决定要使用哪些关键特征,并标准化它们的格式。
我们目前有名字,替代名称,学名,家族以及毒性列,所有这些都是从用BeautifulSoup在ASPCA网站上爬来的。
由于我们将使用谷歌图像搜索收集图像,因此我们决定根据每种植物的确切学名进行搜索,以获得尽可能具体的图像。像“珍珠点”、“大象耳朵”、“蓬松褶边”和“粉红珍珠”这样的名字会很快返回我们所寻找的植物之外的结果。
我们编写了几个快速函数来应用于该系列,以尝试将数据标准化以便进一步清理。
# 确保每个学名的标点符号正确
def normalize_capitalization(x):
first_word, rest = x.split()[0], x.split()[1:]
first_word = [first_word.capitalize()]
rest = [word.lower() for word in rest]
return ' '.join(first_word+rest)
# 清理那些名字不同的重复物种
def species_normalizer(word):
if word.split()[-1] in ['sp','species','spp','sp.','spp.']:
word = ''.join(word.split()[:-1])
return word
# 从名称中删除cv,因为这是一种过时的表示品种的方式
def cv_remover(word):
if 'cv' in word:
word = word.replace(' cv ',' ')
return word
# 从名称中删除var
def var_remover(word):
if 'var' in word:
word = word.replace(' var. ',' ')
return word
# 应用每个函数
aspca_df['Scientific Name'] = aspca_df['Scientific Name'].apply(normalize_capitalization)
aspca_df['Scientific Name'] = aspca_df['Scientific Name'].apply(species_normalizer)
aspca_df['Scientific Name'] = aspca_df['Scientific Name'].apply(cv_remover)
aspca_df['Scientific Name'] = aspca_df['Scientific Name'].apply(var_remover)
# 删除特殊字符
aspca_df['Scientific Name'] = aspca_df['Scientific Name'].apply(lambda x: ''.join([character for character in x if character.isalnum() or character.isspace()]))
# 进一步处理重置数据
aspca_df = aspca_df.sort_values('Scientific Name').drop_duplicates('Scientific Name')
aspca_df = aspca_df.reset_index(drop=True).sort_index()步骤3-通过交叉引用进行深度清理
仔细研究一下我们的数据里的学名(Scientific Name),我们发现很多名称是物种的过时同义词,或者拼写错误。这将在图像采集和以后的训练模型识别具有不同标签的相同图像时引起问题。
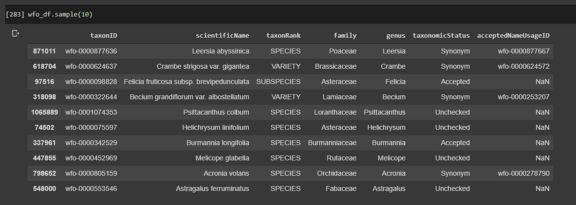
一个谷歌之后,我们发现了世界植物在线数据库,一个开放存取的,基于网络的世界植物物种简编(http://www.worldfloraonline.org/)。它们列出了同义词和公认的物种名称,并由“分类学专家网络”定期更新。非常适合交叉引用我们不可靠的学名。这个数据库以一个.txt文件提供了它们的数据,我们可以读入该文件并与从ASPCA植物毒性数据库中获取的数据库进行比较。
# 读取WFO数据,只保留有用的列
use_cols = ['scientificName','taxonRank','family','genus','taxonomicStatus','taxonID', 'acceptedNameUsageID']
wfo_df = pd.read_csv('/content/drive/My Drive/Houseplant Classifier/classification.txt', sep='\t', lineterminator='\n', usecols=use_cols)
wfo_df = wfo_df.sort_values('taxonomicStatus')作为第一步,我们将对来自ASPCA的数据进行左合并,保留我们的所有类,并添加与我们当前拥有的确切学名匹配的任何数据。我们的目标是将数据库中的所有植物更新为最新的可接受的学名。
# 不需要这个列,我们更信任WFO数据库
aspca_df.drop('Family', axis=1, inplace=True)
# 合并数据文件以获得可信信息
aspca_df = aspca_df.merge(wfo_df, how = 'left', left_on = ['Scientific Name'], right_on = ['scientificName'])
# 按taxonomicStatus进行排序,并删除重复项,保持优先级为被接受的名称
aspca_df = aspca_df.sort_values('taxonomicStatus').drop_duplicates('Scientific Name', keep='first').reset_index(drop=True)
# 用Unknown来填满NaN
aspca_df = aspca_df.fillna('Unknown')步骤3.1-用字符串匹配修复印刷错误
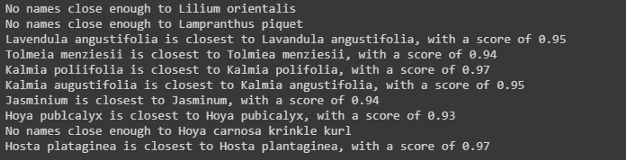
许多学名指的是同一物种,但由于在ASPCA数据库中的打字错误,有几个字母被删掉了。让我们使用difflib中的SequenceMatcher来量化字符串距离,通过比较WFO数据库中不匹配的条目来发现这些错误。
我们可以对数据帧进行排序,只与以同一字母开头的学名进行比较,以节省时间。如果名称足够相似,我们将保留它并最终返回最接近的匹配项。这里我们将阈值设置为0.9,以避免任何不正确的匹配。
def get_closest_name(unknown_name, name_df = wfo_df, name_col = 'scientificName', threshold=0.9, verbose=False):
""" 将'unknown_name'与'name_df'中接受的名称进行匹配。将返回超过接近的“threshold”的名字.
Parameters
----------
unknown_name: str
我们希望与该名称进行匹配.
name_df: DataFrame
包含名称的数据框.
name_col: str, name of name_df column
包含可接受名称的列
threshold: int
unknown_name需要在多大程度上与接受的名称匹配
如果超过这个阈值,名称将被添加到可能的名称字典中
verbose: bool
函数是否打印整个列表
Returns:
----------
str
与‘unknown_name’最接近的、高于给定‘阈值’的名称。
"""
import operator
from difflib import SequenceMatcher
def similar(a, b):
return SequenceMatcher(None, a, b).ratio()
poss_names = {}
# 为了节省时间,只看第一个字母相同的条目
for true_sciname in name_df[name_df[name_col].str.startswith(unknown_name[0])][name_col].values:
similar_score = similar(unknown_name, true_sciname)
if similar_score>threshold:
poss_names[true_sciname]=similar_score
# 如果dict为空
if verbose == True:
print(poss_names)
if not bool(poss_names):
print(f'No names close enough to {unknown_name}.')
return ''
else:
print(f'{unknown_name} is closest to {max(poss_names.items(), key=operator.itemgetter(1))[0]}, with a score of {max(poss_names.items(), key=operator.itemgetter(1))[1]:.2f}')
return max(poss_names.items(), key=operator.itemgetter(1))[0]我们还定义了一个函数来修复数据中的问题条目,它将把它们的学名、科、属和分类状态更新为WFO数据库中的(正确的)相应条目。
def fix_name(unknown_name, true_name):
""" 根据已接受的wfo_df条目修复aspca_df条目.
Parameters
----------
unknown_name: str
我们想要修复的名字.
true_name: DataFrame
修复的名称.
"""
#得到我们想要改变的列
unknown_data = aspca_df[aspca_df['Scientific Name'] == unknown_name]
# 根据ID查找从wfo数据库中获取已接受的数据
true_data = wfo_df[wfo_df['scientificName'] == true_name]
true_sciname = true_data.loc[:,'scientificName'].values[0]
true_family = true_data.loc[:,'family'].values[0]
true_genus = true_data.loc[:,'genus'].values[0]
true_taxonomicStatus = true_data.loc[:,'taxonomicStatus'].values[0]
# 更改学名、科、属和分类学地位为可接受的版本
aspca_df.iloc[unknown_data.index,2] = true_sciname
aspca_df.iloc[unknown_data.index,8] = true_family
aspca_df.iloc[unknown_data.index,9] = true_genus
aspca_df.iloc[unknown_data.index,10] = true_taxonomicStatus现在,我们可以遍历我们的数据,搜索匹配的名称并当场更正它们对应的数据帧条目。
unknown_idx = aspca_df[aspca_df.taxonomicStatus == 'Unknown'].index
print(f'{len(unknown_idx)} plants currently cannot be matched.')
from tqdm.notebook import tqdm
for i in tqdm(unknown_idx):
unknown_name = aspca_df.iloc[i,2]
closest_name = get_closest_name(unknown_name)
if closest_name == '':
continue
fix_name(unknown_name,closest_name)此过程有助于我们发现错误,否则需要进行深入的检查
步骤3.2-人工清理不明物种
不幸的是,许多未被确认的物种在数据库中没有一个足够接近的条目。因此,我们对剩余的未知项进行一些手动修复。谢天谢地,上面的代码将需要手动关注的样本数量减少到了50个左右,我们可以重新使用之前的fix_name函数,根据我们在Google上找到的正确条目来修复这些条目。
步骤3.3-匹配同义学名
既然学名已经全部更正,我们仍然需要对它们进行标准化,因为随着研究的更新,学名可能会随着时间的推移而改变(导致在“分类状态”列中出现同义词标签)。如果一个学名是一个公认的名字的同义词,我们希望在将来的谷歌图像搜索中使用这个被接受的名字。
# 更新剩下的已接受的学名的同义词学名
aspca_df = aspca_df.sort_values('taxonomicStatus').drop_duplicates('Scientific Name', keep='first').reset_index(drop=True)
synonym_idx = aspca_df[aspca_df['taxonomicStatus'].values == 'Synonym'].index
for i in synonym_idx:
# 得到我们想要改变的列
synonym_data = aspca_df.iloc[i,:]
synonym_name = synonym_data.loc['Scientific Name']
# 根据ID查找从wfo数据库中获取已接受的数据
true_data = wfo_df[wfo_df['taxonID'] == synonym_data.loc['acceptedNameUsageID']]
true_sciname = true_data.iloc[:,1].values[0]
fix_name(synonym_name,true_sciname)幸运的是,WFO数据库包含一个acceptedNameUsageID字段,该字段包含给定同义学名的可接受名称,我们可以利用该字段查找接受的学名并将其传递到fix_name函数中。
步骤3.4-结束
现在,我们已经纠正了拼写错误(自动和手动),并将发回的同义词与最新的已接受名称进行了匹配。剩下的就是清理图像下载的数据帧。
# 再次排序并删除
aspca_df = aspca_df.sort_values('taxonomicStatus').drop_duplicates('Scientific Name', keep='first')
aspca_df = aspca_df.sort_values('Scientific Name').reset_index(drop=True).sort_index()
# 设置一个单词名称的属作为名称,而不是NaN
aspca_df.loc[aspca_df.fillna('Unknown')['genus']=='Unknown', 'genus'] = aspca_df.loc[aspca_df.fillna('Unknown')['genus']=='Unknown', 'Scientific Name']
# 删除我们不再需要的行
aspca_df = aspca_df.drop(['taxonID', 'scientificName', 'taxonomicStatus', 'acceptedNameUsageID', 'taxonRank'], axis=1)
# 标准化列名
aspca_df.rename(columns = {'genus':'Genus', 'family':'Family'}, inplace=True)
# 重新排序
cols = ['Name', 'Scientific Name', 'Genus', 'Family', 'Alternative Names', 'Toxic to Dogs', 'Toxic to Cats']
aspca_df = aspca_df[cols]这个过程需要多次迭代才能使方法正确。然而,在我们建立图像数据库之前,确保我们有干净的数据可以工作,这在花费时间训练模型之前是至关重要的。
从最终的宠物植物毒性数据框架中得出一些有趣的结论:
- 110个植物家族中有33个并非完全有毒或无毒。
- 350个植物属中有7个并非完全有毒或无毒。
- 只有两种植物表现出物种特异性毒性,莉莉花对猫和核桃对狗!
步骤4-下载图像
下载图像的第一步是获取我们想要获取的每个图像的url。为此,我们根据fabianbosler的一篇文章,采用了一种基于Selenium的方法。
Selenium是一个用于测试web应用程序的可移植框架。Selenium webdriver充当我们的虚拟浏览器,可以通过python命令进行控制。
这里使用一个脚本来搜索Google图片,我们给它一个查询,只查找和下载缩略图的网址,因为我们要抓取很多图片。一个问题是,谷歌的许多图像缩略图存储为base64编码的图像。我们还想抓取这些图片,这样我们就不会错过任何具有高度相关性的图片,因为我们在搜索结果中走的越远,这些图片就越不适合用于训练目的。
# 如果运行在Colab
!pip install selenium -q
!apt-get update # to update ubuntu to correctly run apt install
!apt install chromium-chromedriver -q
!cp /usr/lib/chromium-browser/chromedriver /usr/bin
import sys
sys.path.insert(0,'/usr/lib/chromium-browser/chromedriver')
# 导入并设置Selenium webdriver
from selenium import webdriver
chrome_options = webdriver.ChromeOptions()
chrome_options.add_argument('--headless')
chrome_options.add_argument('--no-sandbox')
chrome_options.add_argument('--disable-dev-shm-usage')
wd = webdriver.Chrome('chromedriver',chrome_options=chrome_options)import requests
import time
def fetch_thumbnail_urls(query:str, max_links_to_fetch:int, wd:webdriver, sleep_between_interactions:int=1, non_commercial=False, shuffle=False):
""" 使用Selenium webdriver (wd)根据查询从谷歌图像中收集url
可以将sleep_between_interactions更改为适应较慢的计算机。
如果shuffle为真,则返回的url列表将被打乱为随机顺序
Parameters
----------
query: str
传递给谷歌图像。
max_links_to_fetch: int
要获取的url数目。
wd: Selenium webdriver
要使用的webdriver实例。
sleep_between_interactions: int
在webdriver交互之间等待的时间(秒)。
non_commercial: bool
标记仅为非商业用途。
shuffle: bool
返回的url顺序是否打乱。
Returns:
----------
List
url的列表。
"""
def scroll_to_end(wd):
wd.execute_script("window.scrollTo(0, document.body.scrollHeight);")
time.sleep(sleep_between_interactions)
# 构建谷歌查询
if non_commercial == True:
search_url = 'https://www.google.com/search?as_st=y&source=hp&safe=off&tbm=isch&as_epq={q}&gs_l=img&tbs=sur%3Af'
else:
search_url = "https://www.google.com/search?as_st=y&source=hp&safe=off&tbm=isch&as_epq={q}&gs_l=img"
# 加载页面
wd.get(search_url.format(q=query))
image_urls = []
image_count = 0
results_start = 0
while image_count < max_links_to_fetch:
scroll_to_end(wd)
# 获得所有图像缩略图结果
thumbnail_results = wd.find_elements_by_css_selector("img.Q4LuWd")
number_results = len(thumbnail_results)
for img in thumbnail_results:
# 提取图像url,如果它们是可用的地址
if img.get_attribute('src') and 'http' in img.get_attribute('src'):
image_urls.append(img.get_attribute('src'))
# 还获取了谷歌使用的编码图像
elif img.get_attribute('src') and 'data' in img.get_attribute('src'):
image_urls.append(img.get_attribute('src'))
image_count = len(image_urls)
# 如果我们达到指定的配额就中断
if len(image_urls) >= max_links_to_fetch:
break
# 如果我们需要更多的图片,点击加载更多图片按钮
else:
time.sleep(30)
load_more_button = wd.find_element_by_css_selector(".mye4qd")
if load_more_button:
wd.execute_script("document.querySelector('.mye4qd').click();")
# 移动指针
results_start = len(thumbnail_results)
if shuffle==True:
random.shuffle(image_urls)
return image_urls太好了!现在我们有了一种从谷歌图片中获取图片的方法!为了下载我们的图片,我们将利用fast.ai v2。然而,我们将深入研究源代码并对其进行一点升级,以便在图像进入时对其进行哈希处理,并忽略/删除任何重复项,以便最终得到一致的唯一图像集。我们还将允许它解码和下载编码的.jpg和.png图像,这是谷歌图像用来存储缩略图的格式。
# 每个会话运行一次
!pip install fastai==2.0.14 -q
from fastai.vision.all import *import io
from PIL import Image
import base64
import hashlib
def download_images(dest, url_file=None, urls=None, max_pics=150, n_workers=1, timeout=4):
"""
下载文本文件' url_file '中列出的图片到路径' dest ',最多下载' max_pics '个
下载图像后,在保存之前将哈希与其他图像哈希进行比较。
如果哈希已经存在,则尝试下一个url。
Parameters
----------
dest: Path or str
下载目标文件夹。
url_file:
URL文件,\n作为分隔符
urls:
url的列表。
max_pics: int
要下载的图像数量。
n_workers: int
要并行使用的内核数量。
Returns:
----------
从给定的url下载图像到dest目录。
"""
hash_keys = dict()
# 设置哈希以防止复制图像下载
if urls is None: urls = url_file.read().strip().split("\n")
dest = Path(dest)
dest.mkdir(exist_ok=True)
# n_workers必须是1,因为我们在下载过程中检查唯一的图像
parallel(partial(_download_image_inner, dest, timeout=timeout, max_pics=max_pics), list(enumerate(urls)), n_workers=1)
def _download_image_inner(dest, inp, timeout=4, max_pics=150):
# 输入是一个枚举对象
i,url = inp
suffix = re.findall(r'\.\w+?(?=(?:\?|$))', url)
suffix = suffix[0] if len(suffix)>0 else '.jpg'
# 如果我们有足够的图片,什么都不用做,直到url用完
if len(dest.ls()) >= max_pics:
return
# 函数处理base64编码的图像
# 如果抓取的url是已编码的jpg格式,将其解码并与其他格式一起保存
try:
if url[:15] == 'data:image/jpeg':
encoded_image = url[url.find('/9'):]
im = Image.open(io.BytesIO(base64.b64decode(encoded_image)))
filehash = hashlib.md5(im.tobytes()).hexdigest()
if filehash not in hash_keys:
hash_keys[filehash] = i
im.save(dest/f"{i:08d}{suffix}")
else:
pass
except:
pass
# 函数处理base64编码的图像
# 如果抓取的url是已编码的png,将其解码并将其与其余内容一起内联保存
try:
if url[:14] == 'data:image/png':
encoded_image = url[url.find('iVBOR'):]
im = Image.open(io.BytesIO(base64.standard_b64decode(encoded_image))).convert('RGB')
filehash = hashlib.md5(im.tobytes()).hexdigest()
if filehash not in hash_keys:
hash_keys[filehash] = i
im.save(dest/f"{i:08d}{suffix}")
else:
pass
except:
pass
# 如果抓取的url是一个http站点,下载它,并检查我们还没有得到相同的图像。
try:
download_url(url, dest/f"{i:08d}{suffix}", overwrite=True, show_progress=True, timeout=timeout)
im = Image.open(dest/f"{i:08d}{suffix}")
filehash = hashlib.md5(im.tobytes()).hexdigest()
if filehash not in hash_keys:
hash_keys[filehash] = i
else:
(dest/f"{i:08d}{suffix}").unlink()
except Exception as e: f"Couldn't download {url}."现在,我们可以遍历我们的每一个科学植物名称,收集它们的网址,然后下载这些图片,同时验证这些图片是否是唯一的。每一组图像都下载到Colabs上我的链接驱动器中自己的文件夹中。需要注意的一点是,由于google images上存在大量重复的图片,要抓取的url数量必须远远大于你最终想要的图片数量。
# 实例化webdriver
wd = webdriver.Chrome('chromedriver',options=options)
from tqdm.notebook import tqdm
import itertools
scientific_names = aspca_df['Scientific Name']
# 循环所有室内植物的名字,抓取url并下载到我的谷歌驱动器
for name in tqdm(scientific_names):
try:
path = Path('/content/drive/My Drive/Houseplant Classifier/plant_images_deepest');
folder = name
dest = path/folder
dest.mkdir(parents=True, exist_ok=True)
if len(dest.ls())<150:
print(f'{name} has {len(dest.ls())} images.')
url_science = fetch_thumbnail_urls(f'{name}', max_links_to_fetch = 600, wd=wd, non_commercial = False, shuffle = False)
dest = path/folder
# 强制刷新hash_key—在函数中作为全局变量存储,这里清空
hash_keys = dict()
download_images(path/folder, urls = url_science, max_pics=150)
print(f'Finished downloading images of {name} : {len(dest.ls())} images downloaded.')
else:
print(f'{name} already has sufficient images.')
except Exception as e:
print(f'Error with {name}. {e}')下载后,我们将采取步骤确保每个文件夹包含正确数量的唯一图像。
因此,在这个阶段,这些图片被整齐地分到各自的文件夹中,并直接放在我们的谷歌硬盘上。需要注意的是,如果你想用这些图片来训练CNN,如果你在使用它们之前把这些图片带到本地的Colab环境中,但这将在下一篇文章中进一步讨论。
最后
从零开始构建数据库图像分类项目对于简单的玩具示例来说很简单,参见fast.ai v2一个棕色/黑色/泰迪熊分类器的好例子(https://github.com/fastai/fas...。对于这个项目,我想扩展相同的方法,但将其应用到更大的类集合中。这个过程实际上可以分为几个步骤:
- 获取类列表
由于beauthulsoup,从web页面中获取表格或文本数据非常简单,通常只需要通过正则表达式或内置python方法进行更多处理。
清理和验证下载数据的准确性是这一步中最大的挑战。当我们有10个类和领域知识时,在继续之前很容易发现错误并修复它们。当我们有500个类,事情就变得更难了。一个独立的数据源是至关重要的,我们可以根据它来验证我们的数据。在这种情况下,我们信任ASPCA数据中的毒性信息,但不信任它们提供的学名,因此必须使用WFO数据库对其进行更正,后者提供了最新的分类信息。
- 获取每个类的图像url列表
我们可以执行搜索,找到缩略图并下载,甚至可以下载得到更大分辨率的图像。
- 将每个图像下载到带标签的文件夹中
用于下载图像的fastai函数运行良好,但是一个主要的绊脚石是下载重复的图像。如果你想要更多的图片(10-15张),并且你下载了谷歌图片搜索的所有结果,你很快就会得到大量的图片副本。此外,该函数无法处理base64编码的图像。值得庆幸的是,fastai提供了它们的源代码,可以对其进行修改,以解释编码的图像以及下载http链接,下载后对它们进行哈希处理,并且只保留唯一的图像。
原文链接:https://towardsdatascience.co...
欢迎关注磐创AI博客站:
http://panchuang.net/
sklearn机器学习中文官方文档:
http://sklearn123.com/
欢迎关注磐创博客资源汇总站:
http://docs.panchuang.net/