vbm 分析_手把手系列教程:使用FSL进行VBM分析
本文将介绍使用FMRIB Software Library(FSL)做VBM(voxel-based morphometry)分析。
操作系统:linux系统;
本教程使用的环境是ubuntu-18.04.1;虽然ubuntu目前的最新版本是18.10,但最好不要安装最新版本,相关的配套插件尚未完善,FSL运行要报错。
FSL版本号为FSL 6.0.0。
一、准备原始数据
(1)新建一个文件夹(假设命名为VBMtest)作为分析目录,并将所有被试的原始 T1 像放到该文件夹下。这里的文件是nii.gz格式,从磁共振机器拷贝出来的是dicom格式,需要将其压缩成nii.gz格式。具体压缩的软件有很多,熟悉windows系统的同学可以在windows系统使用相关软件完成压缩过程;在本教程中,是在linux环境下的dcm2niix程序完成压缩的。具体请参考:https://github.com/rordenlab/dcm2niix/releases
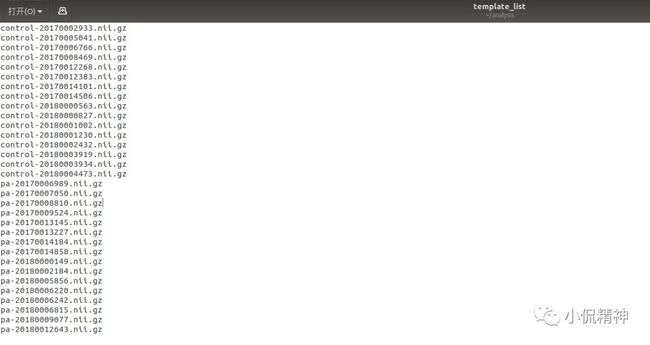
这里有两组被试,第一组健康对照组22 人, 命名为control*.nii.gz (*表示不同的subjects... ),第二组病人组16 人,命名为pa*.nii.gz (*表示不同的subjects... );见图1。
(2)构建组模板。将被试的名字,即 control*.nii.gz...,pa*.nii.gz...放到一个名为template_list 的文件里。如果两组被试数目不一样,就从被试数目较多的一组中随机选择部分被试,使得两组被试数据相等,这是为了保证构建的组模板不会偏向于某一组;具体操作:打开终端,cd到VBMtest目录(或者在VBMtest目录里点击右键,打开终端会自动到该目录),输入:gedit template_list,输入subjects的文件名,然后保存。见图2。
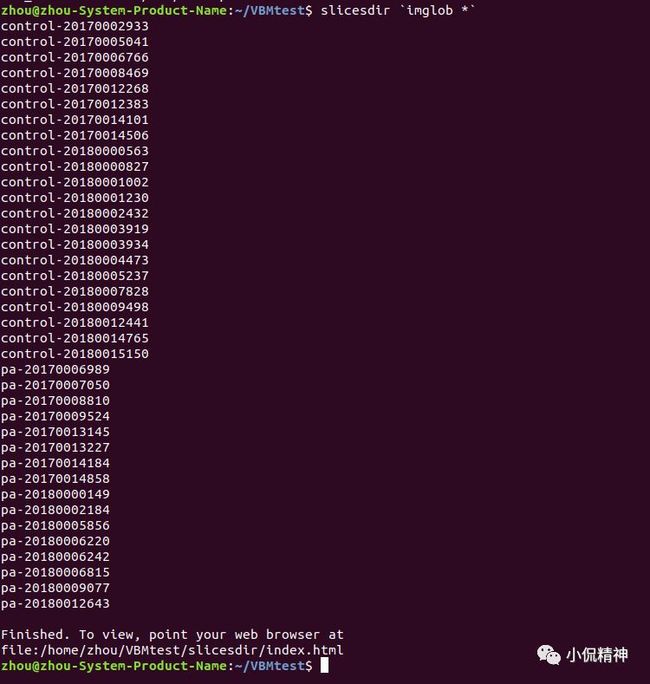
(3)[这部分并非VBM所必须]在命令行下运行slicesdir `imglob *` ,这里*表示所有的subjects. 即当前文件夹下所有被试的文件。见图3。
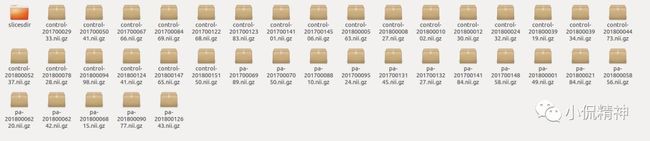
这行命令的作用是产生了一个slicesdir的文件夹,在里面截取每个被试的 T1 图像,并生成一个名为index.html的文件。见图4。
二、构建GLM模型,用于组间比较
(1)设计矩阵中被试的顺序应该与subjects的文件名一致。比如这里健康组的文件名命名为 control* ,而病人组名为pa*, control*在pa*前面(默认排序),所以设计矩阵中前22个应为健康对照组,后16个应为病人组;
(2)在终端中输入Glm(注意这里G一定是大写字母)会弹出两个窗口界面,见图5。
在 Glm Setup 中选择Higher-level/non-timeseries design, inputs设置为38(因为有38个被试),然后按回车;点击Wizard ,弹出 Model setup wizard 界面,选择 two groups, unparied ,设置第一组的被试个数为22(因为control*有22个subjects),点击 Process;见图6。
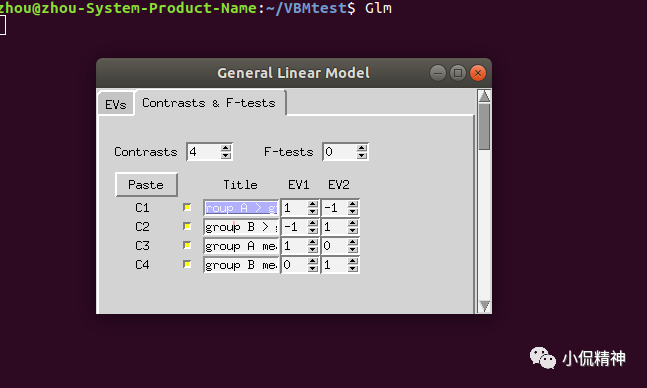
(3)在General Linear Model中设置 Contrasts,C1表示组A大于组B的cluster(1,-1),C2表示组A小于组B的cluster(-1,1),C3表示组A的均值;C4表示组B的均值;如图7所示:
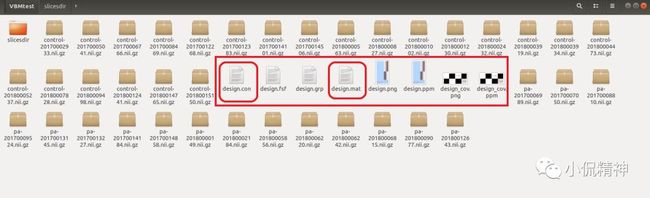
(4)在 Glm Setup 中点击 Save ,假设命名为design。这样在VBMtest 目录下就会出现design.mat和design.con 两个文件;见图8;
(5)想进一步了解其他的GLM设置方法,请参考https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/GLM;对于复杂的统计模型,可以选择手动构建;手动构建可在WPS或office表格中准备数据,将数据另存为txt文件,然后利用Text2Vest命令将数据改为设计矩阵与对比矩阵文件。届时需在终端敲入如下代码:
Text2Vest mat.txt design.mat
Text2Vest conv.txt design.con
Text2Vest ftest.txt design.fts(进行F检验时才需要)
三、颅骨剥离(脑提取)
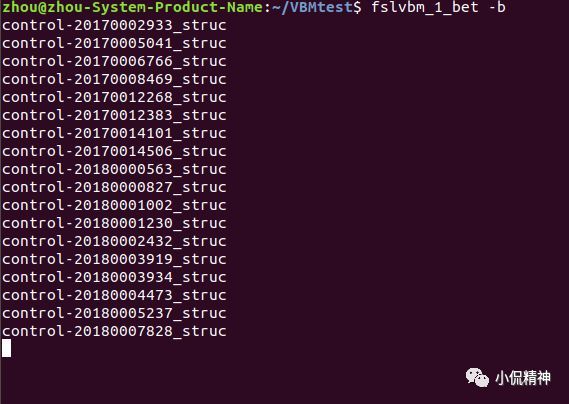
在命令行中运行fslvbm_1_bet -b 或者fslvbm_1_bet -N (如果你的图像包括很多颈部的话),这里选择使用 fslvbm_1_bet -b。见图9。
这行命令的作用是:
(1)在VBMtest里新建名为struc的文件夹,见图10。
将原始T1文件复制到该文件夹下,并添加_struc的后缀,比如control001.nii.gz改为 control001_struc.nii.gz ;
(2)使用Bet进行颅骨剥离,输出后缀名为_brain.nii.gz的文件,比如control001_struc.nii.gz 进行颅骨剥离后的文件名为control001_struc_brain.nii.gz;见图11;
(3)对颅骨剥离后的图像进行截图,并生成网页文件用于质量检查,index.html文件位于struc/slicesdir;打开后如图12所示(请原谅例子数据的质量)。
四、构建灰质组模板
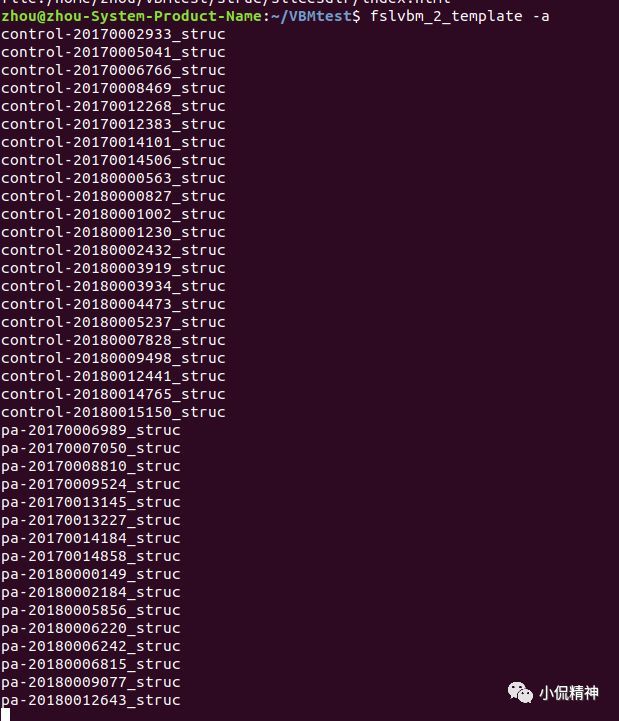
在命令行中运行 fslvbm_2_template -a(仿射配准)或者 fslvbm_2_template -n(非线性配准),为了节约时间,这里使用仿射配准方式生成组模板,所以使用fslvbm_2_template -a。如图13所示。
主要过程包括:
(1)将每个被试颅骨剥离后的图像分割成灰质、白质和脑脊液三部分;
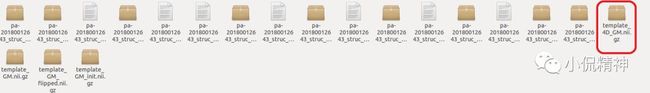
(2)将每个被试的灰质图像线性配准到 ICBM-152灰质模板上,平均后得到初步模板。再将每个被试的灰质图像线性配准到初步模板上,平均后得到最终的组模板;你可以选择用fsleyes打开查看template_GM_4D文件,如图14所示。
五、配准和平滑
在命令行运行 fslvbm_3_proc ,如图15所示。
这一步主要包括:
(1)在VBMtest文件夹里新建一个名为stats的文件夹;
(2)将每个被试的灰质配准到上一步构建的组模板上,并将配准后的图像进行不同程度的平滑;
(3)根据前面得到的统计模型进行组间比较,生成不同平滑度(Gaussian核;sigma = 2,3, 4mm)下的T值图,根据T值图的情况,选择该用哪一种平滑后的结果;本例中选择sigma=3,即文件名中包括s3的文件。
六、组间比较和多重比较校正
使用randomise命令进行置换检验,建议使用TFCE方法进行多重比较矫正,cd到stats文件夹,在终端输入命令如下:
randomise -i GM_mod_merg_s3 -m GM_mask -o fslvbm -d design.mat -t design.con -T -n 5000
运行完成后可见stats文件夹里增加了fslvbm_tfce_corrp_tstat1,fslvbm_tfce_corrp_tstat2之类的文件,这就是TFCE多重比较矫正之后的结果。
七:查看与输出结果
校正后的结果为 1-p ,所以小于 0.05 即为大于0.949。使用fsleyes查看组间比较结果是否显著,在终端敲入命令:
fsleyes -std fslvbm_tfce_corrp_tstat1 -cm hot -dr 0.949 1
以上命令发现结果为阴性,更改P值为0.5(为了方面演示结果)。在终端敲入命令(与上述命令基本相同,只是把0.949改为0.5):
fsleyes -std fslvbm_tfce_corrp_tstat1 -cm hot -dr 0.5 1
结果如图16所示。
除此之外,也可以用cluster命令查看显著的cluster,在终端敲入命令:
cluster -i fslvbm_tfce_corrp_tstat1 -t 0.949
结果同fsleyes为阴性,P换为0.5之后发现阳性cluster,如图17所示。
以上显示的是voxel的坐标,如要显示MNI标准空间的坐标,需在命令后加--mm,命令如下所示:
cluster -i fslvbm_tfce_corrp_tstat1 -t 0.5 --mm
结果见图18。
那么我们得到峰值坐标点在MNI空间的坐标为-40,-22,-26
进一步明确这个坐标在脑地图集的那个位置,可使用如下命令,如图19所示。
atlasquery -a "MNI Structural Atlas" -c -40,-22,-26
如要获取BA分区相关信息,可使用其他软件,如芒果(mango)软件,打开MNI模板,输入坐标,即可出现BA分区信息,具体在此不再详述。Mango软件在Windows系统中也可以使用。请参考:http://ric.uthscsa.edu/mango/mango.html
导出显著的cluster信息,可在终端敲入如下命令:
cluster-i fslvbm_tfce_corrp_tstat1 -t 0.5 -o cluster_index --osize=cluster_size >cluster_info.txt
以上命令导出三个文件,cluster_index,cluster_size, cluster_info.如图20所示。
进一步将显著的cluster制作mask,可在终端敲入如下命令:
fslmaths-dt int cluster_index -thr 1 -uthr 1 -bin cluster_mask1
可以看到多出了一个文件”cluster_mask1”,如图21所示。
提取每一个subjects的显著cluster的体积,可在终端敲入如下命令,见图22:
fslmeants-i GM_mod_merg_s3 -m cluster_mask1 -o Cluster1
可看到多出了一个文件“cluster1”,见图23。
打开之后如图24所示。按照文件中subjects的排序,每一行就是相对应subjects的cluster1的体积。
注意事项:fslvbm_2_template与fslvbm_3_proc命令需花费较长时间,在敲入命令前请做好思想准备。
参考资料:https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FSLVBM/UserGuide
更多精彩敬请关注: