KGNN:基于知识图谱的图神经网络预测药物与药物相互作用
点击蓝字
![]()
关注我们
AI TIME欢迎每一位AI爱好者的加入!
药物间相互作用(DDI)预测是药理学和临床应用中一个具有挑战性的问题,在临床试验期间,有效识别潜在的DDI对患者和社会至关重要。现有的大多数方法采用基于AI的计算模型,通常倾向于集成多个数据源并结合先进的图嵌入方法来实现。然而研究人员很少关注药物与其他实体(例如靶标和基因)之间存在的潜在关联。此外,最近的研究还采用知识图谱(KG)进行DDI预测。这一系列方法都是采取直接学习节点的潜在嵌入向量,但它们对于获得KG中每个实体的丰富邻域信息受到限制。
为解决上述局限性,林轩等人提出了一种端到端的框架,即基于知识图谱的图神经网络(KGNN),以解决DDI预测问题。该框架可通过在KG中挖掘相关联的关系,来有效地捕获药物及其潜在的邻域实体信息。为了提取KG中的高阶结构和语义关系,对KG中每个实体的邻域进行学习,作为它们的局部感知域,然后将邻域信息与来自当前实体表示的偏差进行整合。这样,感知域可以自然地扩展到多个跃点,以对高阶拓扑信息进行建模并获得潜在的药物长距离相关性特征。本次报告,我们有幸邀请到来自湖南大学的林轩博士为大家分享他们的这项研究工作!
林轩:湖南大学计算机科学与技术四年级博士生,导师为全哲副教授。于2019年10月前往伊利诺伊大学芝加哥分校计算机学院进行博士联合培养,指导老师是Philip S.Yu教授。主要研究方向为机器学习、图神经网络和药物重定位。目前已在IJCAI、AAAI、ECAI、Briefings in Bioinformatics等国际会议和期刊发表论文7篇,并担任IJCAI、AAAI、Briefings in Bioinformatics、Neurocomputing等会议和期刊审稿人。
![]()
一、背景和动机
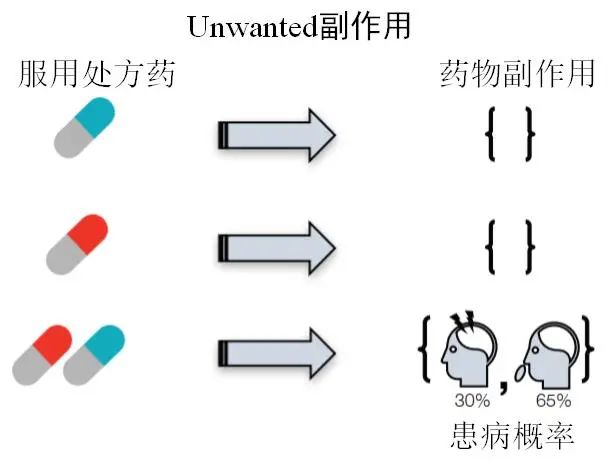
药物间的相互作用(DDI)是指同时或先后服用两种或两种以上药物时,药物之间所产生的相互作用,而该相互作用可能会导致意想不到的副作用。举个例子,在日常生活中,某人因睡眠不佳,服用了助眠药物,比如镇定剂。与此同时他又出现了过敏反应,需要服用治疗过敏的药物,比如抗组胺药。当两种药物混合服用,就可能会减缓大脑的反应。如果此人是从事车辆驾驶或者机械操作等需要注意力高度集中的工作,那么一旦出现紧急情况,就可能因无法及时做出反应,发生难以预料的危险。因此,如果能够提前预测DDI,就能有效避免类似情况的发生。
图1 药物间相互作用
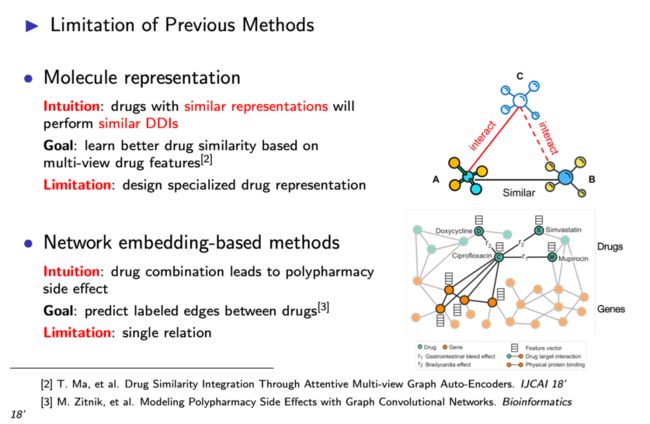
总结归纳现有DDI预测方法,大致可分为两大类。一类是分子表示,主要聚焦于药物分子的特征学习。这类方法都基于同样的假设:即具有相似嵌入表示的药物分子将会表现出相似的DDI。如图2右边所示,分子A和分子B有相似的分子结构,那它们所学到的特征向量也是相似的,如果分子A与分子C存在相互作用,那么可以推断分子B和分子C也有类似DDI存在。药物分子特征学习有很多方法,比如类似文本编码的一维SMILES序列,比如传统基于分子描述符或分子指纹ECFP的方法,或是基于3D坐标轴位置信息的方法。如文献2中提到了一种新颖的分子表示方法,即基于多视角药物特征学习更好的药物相似性,但这种方法仅限于对药物分子本身的表示学习,大多数情况下都依赖于领域知识。
另一类常用的DDI预测方法是基于网络嵌入的方法,通过构建各种与药物有关的生物网络,在这个网络中,将药物看作网络中的节点,通过学习节点的嵌入表示来预测潜在的边,即DDI的关系。构建映射关系网络也有多种方法,比如矩阵分解,把目标关系构建成一个矩阵进行求解;比如随机游走,在图中选择固定的路径进行游走以获取更多的节点特征。这类方法的目标在于预测药物之间的标签边,但它们只关注单一的DDI关系,并没有考虑与药物有关的其他联系。
图2 现有DDI预测方法
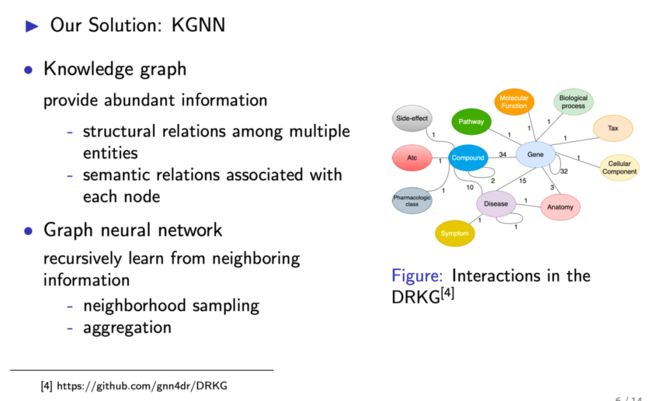
通过以上分析可以发现,这些方法的初衷是希望获得更多生物关联的信息。如果一个图或数据能够提供更多信息,那么就能有效辅助DDI预测,此时知识图谱就成为了一个上佳的选择。因为知识图谱蕴含了丰富的信息,包括多个实体之间的结构关系、与每个节点关联的语义关系等。在对过去DDI预测方法的梳理中,我们也找到了基于知识图谱的方法,但这个方法是采用知识图谱嵌入的方式,直接学习节点的嵌入表示,没有考虑每个实体丰富的邻域信息。为突破局限,林轩等人在知识图谱中引入图神经网络,借用图神经网络对每个节点进行邻域采样,通过聚合邻域信息获得实体的嵌入表示,这也就是基于知识图谱的图神经网络的动机来源。
图3 知识图谱和图神经网络
二、基于知识图谱的图神经网络
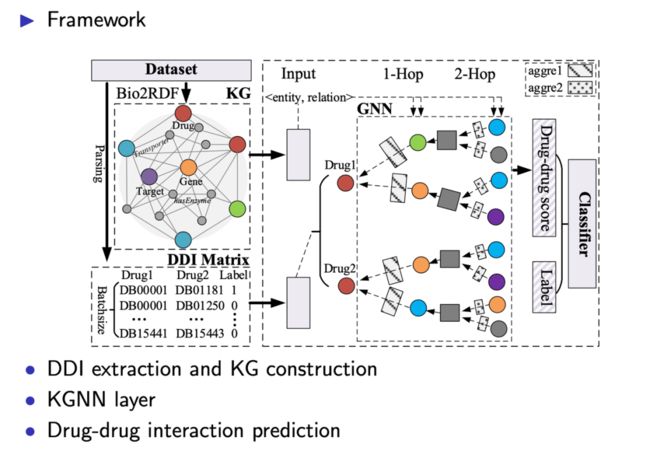
图4是基于知识图谱的图神经网络的整体框架,共包括三个模块:(1)DDI提取与KG构建;(2)KGNN层;(3)药物与药物相互作用预测。
图4 基于知识图谱的图神经网络框架
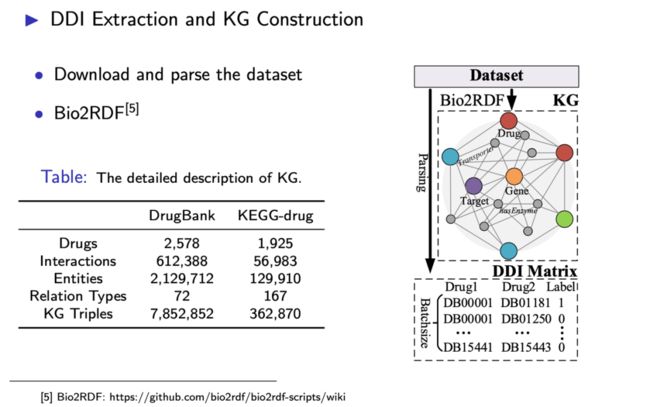
具体来说,第一步中DDI提取主要使用了DrugBank和KEGG-drug两个数据集。对数据集进行解析以提取药物对,这里的药物对其实是经过FDA认证的DDI,如图4中“DB00001-DB01181”所示。知识图谱构建,使用Bio2RDF工具构建链接的数据网络,基于传输定义从不同格式的数据源中获取数据,从而创建与RDF数据格式兼容的链接数据。
图5 DDI提取和KG构建
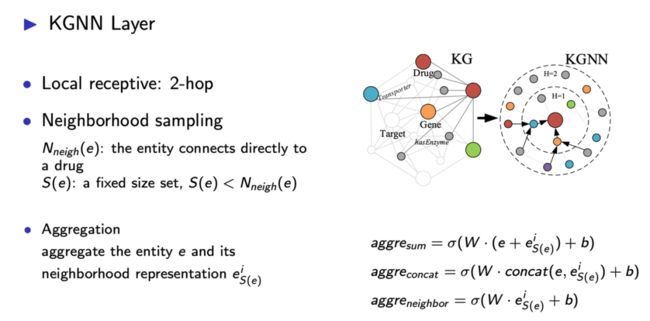
获得输入以后,需要对实体的邻域进行采样。每一个药物实体的邻域分布情况是不一样的,图6中红色的节点表示药物节点,考虑每个药物节点两跳的邻域范围。H参数可以理解为CNN中的感知域,H=1相当于只考虑与当前节点直接相连的邻居节点,H=2表示考虑二阶相连的节点情况,这样能够学习到更多的邻域实体信息,当然H可以取更大值。在这个框架中GNN是一种空间域的方法。在构建的知识图谱中,把和药物节点直接相连的节点定义为Nneigh(e)。因为每个药物节点邻域的分布是不同的,为了计算方便,借鉴GraphSAGE方法,采用固定大小的邻域范围S(e)。采样完成之后,通过三种聚合方法将实体自身的嵌入表示和邻域信息的嵌入表示聚合起来,最终得到当前实体的嵌入表示。其中,sum聚合方法是一种叠加操作,concat是一种拼接操作,neighbor只考虑邻域的信息,而忽略自身实体嵌入表示。
图6 KGNN层
图7总结了KGNN算法,回顾整个框架可分为三个步骤:(1)从数据集中提取药物对并构建相应的知识图谱;(2)将信息输入KGNN中,获得药物及其相关实体邻域的特征;(3)计算两个药物的相似度并反馈输出交互值。
图7 KGNN算法
三、实验结果
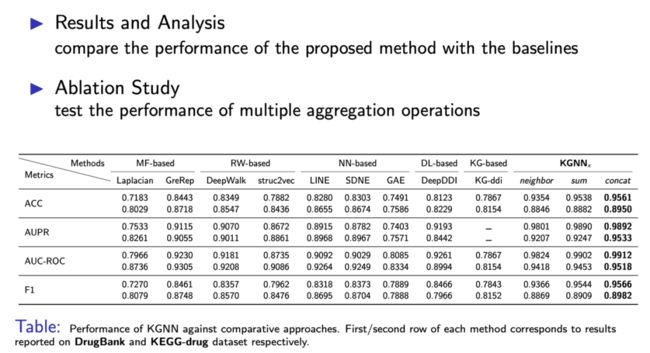
实验部分,通过DrugBank和KEGG-drug两个数据集来评估KGNN的性能。对于两个数据集,以8/1/1的比例将所有批准的DDI作为正样本随机分为训练、验证和测试集,并随机抽取正样本的补集作为负样本用于模型训练。使用多种指标评估预测性能,包括ACC、AUPR、AUC-ROC和F1分数。选取5类(MF、RW、NN、DL、KG)共9种方法作为实验基准,以更好对比实验结果。
图8 实验设定
图9是KGNN与基准方法的性能比较,每种方法的第一/第二行分别对应于DrugBank和KEGG-drug数据集上的实验结果。通过对比实验结果可以发现,KGNN在两个数据集上都取得了最优的效果。另外,在消融实验部分测试了三种聚合方法的性能,发现通过拼接聚合(concat)的方法效果是最好的。同时,这些变体的结果均优于基准对比方法,反映出了KGNN方法的稳定性。
图9 对比、消融实验结果
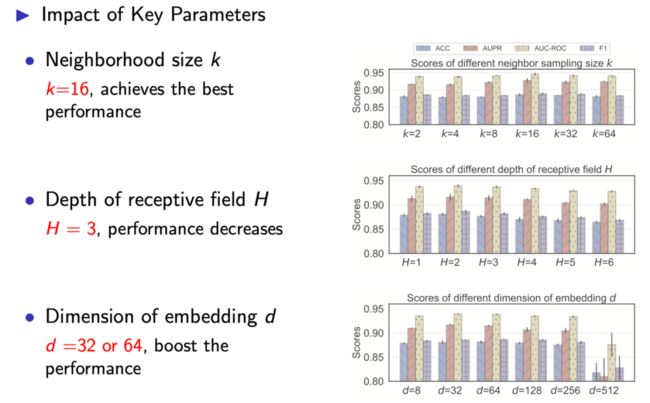
实验还研究了k、H、d三个关键参数对KGNN性能的影响。首先,改变邻域大小k发现,当k=16时,KGNN可获得最佳性能。这说明如果采样的邻居节点个数太少,邻域所能够包含的信息会不够。其次,通过设置为1到6(大于6时超出系统内存)来研究感知域深度H的影响。实验结果显示,当H=2时可以学到较多的特征,但模型所有指标的性能都会从H = 3开始降低。最后,检验嵌入维度大小d的影响如设置由8变化为512。结果表明,可通过设置适当的维度大小来提高其学习能力,值过大反而会带来过拟合的情况。
图10 不同参数的实验结果
四、总结和未来展望
总结来说,林轩等人的工作提出了一种新颖的框架来预测DDI任务。该框架将图神经网络应用到了知识图谱当中,同时考虑了药物实体在知识图谱中的拓扑结构信息以及自身附带的语义关联信息。对于未来的工作,主要有以下几点想法:(1)考虑更大规模的知识图谱;(2)设计有效的邻域采样方法;(3)拓展到多类型的DDI预测或其他相关任务,而不是仅限于二分类预测。
图11 总结和未来工作
Paper: https://www.ijcai.org/Proceedings/2020/0380.pdf
Code: https://github.com/jacklin18/KGNN
整理:何文莉
审稿:林 轩
排版:岳白雪
AI TIME欢迎AI领域学者投稿,期待大家剖析学科历史发展和前沿技术。针对热门话题,我们将邀请专家一起论道。同时,我们也长期招募优质的撰稿人,顶级的平台需要顶级的你!
请将简历等信息发至[email protected]!
微信联系:AITIME_HY
AI TIME是清华大学计算机系一群关注人工智能发展,并有思想情怀的青年学者们创办的圈子,旨在发扬科学思辨精神,邀请各界人士对人工智能理论、算法、场景、应用的本质问题进行探索,加强思想碰撞,打造一个知识分享的聚集地。
![]()
更多资讯请扫码关注
![]()
(直播回放:https://b23.tv/ifBA8L)
(点击“阅读原文”下载本次报告ppt)