pandas head 显示全部_Pandas与机器学习实例——肝炎数据集(1)
在学习了一段时间的pandas后,我们需要练习一些案例来巩固我们的实操能力。今天带来的是我在学习pandas和决策树时使用过的一个肝炎数据集,讲的内容主要涉及到:利用pandas处理缺失值、构建决策树等机器学习的相关知识。
首先和大家分享一下数据:
链接:
https://pan.baidu.com/s/15gdftmUHVbkh4dA79eNsjQpan.baidu.com提取码:jmx7
下面开始我们的实例学习之路~
一、了解数据集
(1)将文件导入我们的python中。(我用的是jupyter notebook )
import numpy as np
import pandas as pd
df = pd.read_excel(r'C:UsersAdministratorDesktopdata_feiyan.xlsx',encoding='utf-8')导入成功~
(2)查看数据的信息
data.head() #查看数据前五行
data.tail() #查看数据后几行
data.shape #查看数据的大小(几行几列),查看矩阵或数组的维数
http://data.info( ) #查看数据信息(数据类型。缺失值)
data.describe() #观察数据的描述性统计信息,观察是否有异常值
1、先用head()看看数据长什么样:
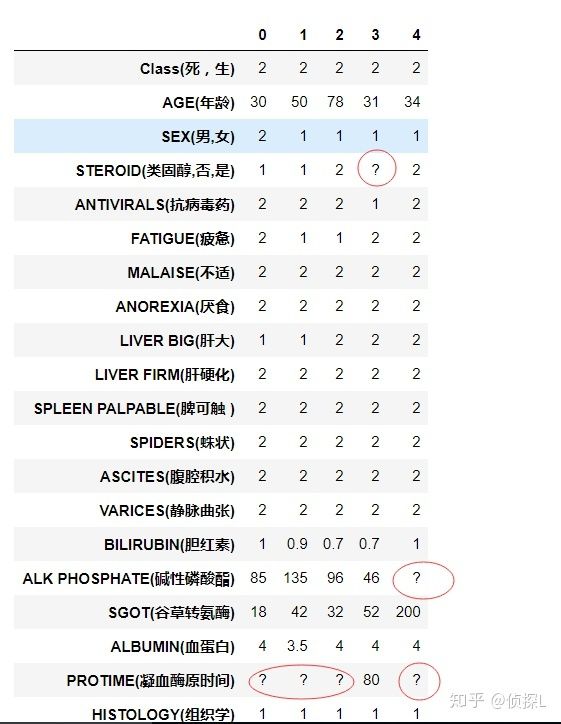
df.head().T注:由于列数较多,因此我们打印前5行的时候用(.T)转置了一下,从上面的结果可以看到,数据集中出现了较多的“?”。
出现“?”的情况,应该是由于数据缺失,然后在Excel表格中被默认写成“?”的形式,因此在进行实验之前,我们需要把“?”重新表现成缺失值的样子。
2、使用replace()函数将“?”转换成缺失值。
df.replace('?',np.nan,inplace=True)同时,我们也可以看到,特征名有一点点长:(看着总觉得有点别扭,强迫症呀。。)
3、处理一下数据集的特征名:
df.columns=['是否生还', '年龄', '性别', '类固醇', '抗病毒药','疲惫','不适','厌食','肝大','肝硬化','脾可触','蛛状','腹腔积水','静脉曲张','胆红素'
,'碱性磷酸酯','谷草转氨酶','血蛋白','凝血酶原时间','组织学']改完之后,我们再次用head()看看数据长什么样:
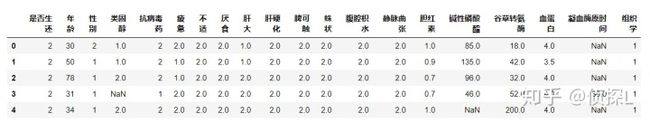
df.head()舒服多了~
4、查看数据的总体情况:
df.shape可以看到,数据总共有155行,20列。
df.info()看上去,好像缺失值有挺多的。(每一列本来应该都有155个数据的)
5、我们来看看具体每列有多少个缺失值:
df.isnull().sum()可以看到,各列缺失值的数量各有不同。
由于本次实例的目的是为了巩固pandas的使用技巧,因此我将针对不同列的缺失情况,采用不用的缺失值填补方法。
二、开始填补缺失值
在之前的文章中,我也介绍过关于缺失值处理的一些小技巧,忘记的小伙伴可以看看:
侦探L:如何处理Pandas里的缺失值(入门篇1)zhuanlan.zhihu.com(1)从上图可以看到,'类固醇','疲惫','不适','厌食','肝大','肝硬化','脾可触','蛛状','腹腔积水','静脉曲张'这几列数据类似逻辑变量,取值均在(1,2)之间,而已缺失值数量不多,因此,这四列的缺失值我们采用众数的方式填补。
众数函数表示:mode()[0]。
注:带了[0]是因为一列数据里可能有多个众数,为了避免发生多个众数冲突而出现缺失值填补不上的问题,所以指定第一个众数。
df_mode=['类固醇','疲惫','不适','厌食','肝大','肝硬化','脾可触','蛛状','腹腔积水','静脉曲张']
for i in df_mode:
df.loc[:,i]=df.loc[:,i].fillna(df.loc[:,i].mode()[0])当然我们也可以看看这几列的众数分别是什么:
df.loc[:,['类固醇','疲惫','不适','厌食','肝大','肝硬化','脾可触','蛛状','腹腔积水','静脉曲张']].mode()(2)'胆红素','碱性磷酸酯','谷草转氨酶','血蛋白'这几列数据属于数值型变量,我们采用“均值”的方式对其进行缺失值填充。
均值函数表示:mean( )
df_mean=['胆红素','碱性磷酸酯','谷草转氨酶','血蛋白']
for i in df_mean:
df.loc[:,i]=df.loc[:,i].fillna(df.loc[:,i].mean())查看一下这几列的均值:
df.loc[:,['胆红素','碱性磷酸酯','谷草转氨酶','血蛋白']].mean()(3)此时,缺失特征列只剩下‘凝血酶原时间’这一列,同时我们也可以看到,这一列的缺失数据是比较多的,有67个。此时如果我们仍采用均值、中位数等简单暴力的填充方式的话,填充效果可能会很差。因此,我们采用随机森林回归(RandomForestRegressor)的方法进行填充。
随机森林回归填充缺失值的理论说明:
侦探L:如何填补Pandas中的缺失值(机器学习入门篇)zhuanlan.zhihu.com首先,导入我们所需要使用的库:
from sklearn.ensemble import RandomForestRegressor接着,将我们的数据集分成两部分:
第一部分,不含缺失值的其他所有列:
df_full=df.drop(labels='凝血酶原时间',axis=1)
df_full.head()第二部分,含缺失值的那一列:
df_nan=df.loc[:,'凝血酶原时间']
df_nan.head()然后,区别测试集与训练集:
Ytrain = df_nan[df_nan.notnull()]
Ytest = df_nan[df_nan.isnull()]
Xtrain = df_full.iloc[Ytrain.index]
Xtest = df_full.iloc[Ytest.index]接着,实例化,然后用随机森林回归来填补缺失值:
#用随机森林回归来填补缺失值
rfc = RandomForestRegressor(n_estimators=100)
rfc = rfc.fit(Xtrain, Ytrain)
Ypredict = rfc.predict(Xtest)完成~
看看我们的预测结果(即代替原来缺失的结果):
Ypredict将结果填补到我们原来的数据表中:
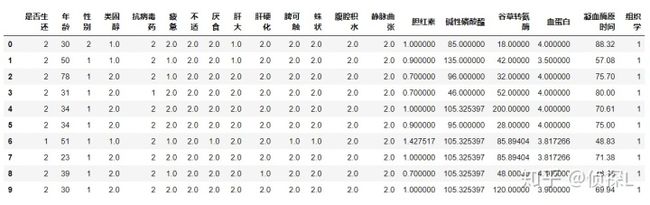
df_nan[df_nan.isnull()] = Ypredict此时,我们的填补过程已经全部结束,让我们再次看看我们数据集填补后的样子:
df.head(10)确认一下此时数据集里还有无缺失数据:
df.isnull().sum()很好~此时我们的数据集里的缺失值已全部填补完毕~
以上便是