文章来源:Multi-omic data integration and analysis using systems genomics approaches: methods and applications in animal production, health and welfare
阅读者:李冬艳
摘要
在过去的几年里,高通量组学技术(HTO)例如整个生物学各个方面的基因组学,表观基因组学,转录组学,蛋白质组学和代谢组学,取得了显著发展。该技术引领了系统生物学时代的进步,包括在动物生产和健康性状方面的应用。尽管有了这些新的HTO技术,在数据分析方面仍然面临着新的挑战。
一方面,根据自身优点判断不同的HTO技术适用于致病基因的鉴定,预防疾病的生物标志物和治疗疾病的药物靶点,以及对性能或疾病风险进行个性化的基因组预测。另一方面,复合多组数据和联合建模与分析,对于了解动物健康和可持续生产的系统生物学是非常有力和准确的。在介绍用于分析和整合多基因组学数据以改善动物生产,健康和增益的复合系统基因组学框架之前,我们将概述当前的新兴的HTO技术,并重点关注其在动物和兽医学中的应用。
我们的结论是,在多基因组学数据复合,建模和系统级分析方面,尤其是快速发展的HTO技术面临着巨大的挑战。我们着重强调现有的新兴的系统基因组学方法,并讨论它们如何有助于我们对复杂性状或疾病的生物学的理解以及生产性能,抗病性和增益的全面提升。
背景
寻找病因和调节基因变异,并使用复杂疾病和性状的预测性遗传标记或生物标记,构成了包括家畜基因组在内的所有基因组学的主要基准。全基因组关联研究以潜在的causal单核苷酸多态性,结构变异和candidate基因的形式,对复杂疾病和性状的遗传结构提供了有用的见解。然而,这些全基因组关联研究结果对如何提高我们对疾病和复杂性状的分子路径的理解的证据不充分,因此给全基因组关联研究后的分子数据表征带来困难。这种关联模式是从基因组之外推导到functome scale。
由于两种或多种高通量组学技术(基因组学,表观基因组学,转录组学,蛋白质组学,代谢组学,metagenomics等)可以潜在地应用于同一动物或来自同一动物的生物样本,重要的是评估如何将这些不同生物学水平的多样化数据复合起来,以充分利用此类信息的潜力,从而全面提升动物的生产性能,抗病性和。有趣的是,如何将这种HTO技术用于特定的组学分析。系统生物学已与现在的系统遗传学(在动物育种背景下适当地称为“系统遗传学”或“系统基因组学”)建立了有意义的联系。在此过程中,研究人员通过积极利用我们的知识成功地定义了从“omes”到后缀“logy”的术语(表1)。 “系统遗传学”或“系统基因组学”研究使用“omic”技术测量的复杂特征,例如基因组/外显子组阵列,基因表达阵列,质谱和新一代测序(NGS)。这些技术被用于研究基因组,转录组,表观基因组或甲基基因组,并将这些内部或“内表型”的变异与外部或“外表型”联系起来。系统遗传学讨论了系统生物学的一个分支,该分支集成了“omics”的规模数据,并使用它们来识别因果基因和网络,调节基因和网络以及复杂性状的预测标记。
最近,有几篇文章回顾了动物环境下的系统生物学和系统遗传学。从动物的组织层次到动物内部系统的各个组成部分,这些论文概述了启用“‘omics’-enabled”具有大量数据的组件。此外,在动物系统生物学研讨会上发表的一系列论文试图缩小这一差距,证明基因型和表型之间存在着复杂的调控关系,并着重强调了这些复合系统生物学方法的适用性。
由于在多个HTO平台(主要是NGS机器)上生成的数据量非常大,因此将数据复合到多组空间中既困难又乏味。在对原始数据集进行质量控制后生成的二次数据集将进一步通过生物信息学和统计方法进行分析,以创建三级数据集(质量控制的最终数据集),这反过来又构成了系统基因组分析输入数据的基础。有许多从基因组,转录组和表型资源中获得的,针对复杂性状的数据的综合资料库,这些资料可以解释变异体(单核苷酸多态性(SNP),插入,缺失,拷贝数变异体(CNV))的作用。研究已证实了一些旨在结合生物学发现和从各种资源获得的数据相结合的发展。
最近,以“基因组<==>表型高速公路(Phenome Superhighway)”(GPS)的形式概念化了一个完整的模型,该模型可以估计与疾病有关的候选基因和影响相互作用的基因。内部或内表型的遗传图谱(连锁或关联)(例如,基于基因表达或代谢产物或蛋白质水平或定量性状位点(QTL)图谱)已产生了大量数据,并产生了对这些数据进行分类,注释,存储和分析的需要,以了解它们在这些内表型遗传变异中所起的作用。更准确地说,包含某一特征数据的“omic”空间将促进我们对与生化途径以及大分子与小分子之间相互作用的功能的理解。但是,有必要仔细地对各种生成多组学数据集的HTO方法进行质量控制,以消除冗余和假阳性数据。由于数据冗余在数据错误使用方面构成了巨大的威胁,因此在公共信息空间中共享数据将减少冗余数据的数量和出错的可能性。这也将有助于更大程度地协调不同“组学”数据之间的标准化活动,这是考虑到来自这些不同来源的数据整合的一个关键问题。假设可以对每个HTO数据集进行质量控制,那么下一步的逻辑步骤是评估如何将代表不同生物学水平的这些不同的HTO数据集进行整合和联合分析,以充分发挥其在提升动物生产,健康和增益方面的潜力。通过前面对多基因组学数据复合与分析的介绍,本文的主要目的是概述当前和新兴的生成主要“omic”数据类型方法,并重点介绍在动物和兽医学中的某些应用。本文仅关注系统基因组学中的主要HTO数据类型(基因组学,转录组学,表观基因组学,代谢组学和蛋白质组学)。本文的最后一部分在新兴的多组学背景下重新介绍了“系统遗传学”或“系统基因组学”,并对如何将系统基因组学用于提升动物生产,健康和增益提供了展望。
讨论
动物基因组学
对于人类和各种动物物种,“全基因组”的研究可以追溯到20世纪90年代,当时的研究重点是使用GWAS方法鉴定遗传变异,该方法基于具有数以万计SNP的微阵列或芯片。在GWAS中,对每个SNP进行统计测试,以确定与特性/表型相关的显着性。
与多种现有的人类SNP芯片相比,动物SNP芯片上的SNP数量要少得多,例如,猪和鸡60K,绵羊50K,牛777K。在牲畜基因组学中,许多GWAS方法都着重于生产和健康特征。在2015年,报告了在牛,猪和鸡中进行的GWAS的广泛审查。例如,关于全球适应气候变化的肉牛的雌性繁殖性状的GWAS,猪饲料效率性状,肉鸡体重以及以猪为模型的肥胖和代谢性疾病进行了研究。与人相比,在牲畜物种中执行GWAS的优势在于可获得相关动物以及有关谱系的后续知识,这大大减少了检测与目标性状相关的遗传变异所需的个体数量。此外,在动物基因组中比在人类基因组中更为广泛的连锁不平衡(LD)取决于动物之间的相关性,它对所需样本量具有正面影响。
动物育种通过采用解释种群结构/系谱的混合模型来解决此类问题。一个重要的生物信息学任务是注释GWAS的变体,该变体解释了一定比例的表型变异和功能特性的预测,这些功能有助于基于多个数据库构建基于本体的功能网络。许多网络服务都是为了迎接挑战而创建的,但它们大多只是在人类医学研究的框架内工作。最近,启动了动物基因组功能注释(FAANG)国际项目联合会,目的是召集动物科学家一起,并持续关注该社区之间的合作。这个FAANG联合会的四个委员会中有三个委员会根据世界各地研究人员的贡献,处理动物基因组功能注释的关键问题,即动物、样本和化验委员会(ASA),生物信息学与数据分析委员会(B&DA)和元数据与数据共享(M&DS)委员会。此外,1000头牛基因组项目为牛研究界提供了大量关于牛变体的数据,这些数据将有助于GWAS和因果突变的识别。 这些举措为系统整合系统生物学和系统遗传学的发现并在网上提供这些发现铺平了道路。
动物育种的另一个非常重要的革命是基因组选择(GS),这是以一种标记辅助选择的形式,其中将整个基因组(SNP)与谱系结合使用,以预测特定种群中动物的育种价值。GS或基因组预测通常包括两个阶段。首先,使用可得到表型和基因组数据的训练群体来估计SNP的作用。其次,已知SNP的作用可用于预测可获得基因组数据的种群的育种值。基因组最佳线性无偏预(GBLUP)方法使用基因组关系矩阵(GRM),该基因组关系矩阵利用全基因组基因分型数据描述基因型个体之间的关系,并且在等位基因频率标准化矩阵后,其行为类似于分子关系矩阵A在常规的BLUP中。 GBLUP提供了动物的基因组估计育种值(GEBV)。该GBLUP版本已演变为单步方法(ssBLUP),也称HBLUP方法,通过对基因型和非基因型动物使用GRM和常规A矩阵来计算GEBV。
在几种GS方法中,由于GBLUP和ssBLUP它们的假设简单且易于计算,因此使用最为广泛。假设混合模型中建立QTL /候选基因的识别,通常采用贝叶斯方法,这些方法假定SNP效应具有不同的分布特性,而且有限比例的SNP具有非零效应。 GS在牲畜基因组学中具有巨大的优势,因为它大大缩短了生成间隔,从而增加了对选择的响应。此外,很难甚至不可能测量的性状(如公牛的产奶量或活体动物的胴体性状)现在可以从基因上预测,并用于提高育种水平。GS目前被广泛使用,例如在养猪业和养牛业中使用较多。
最近,序列辅助选择的潜在潜力被证明是不现实的,因为与高密度基因分型相比,基于序列的基因分型可以提高准确性。然而,对包括数量性状核苷酸(QTN)或数量性状基因位点(QTL)的认识的提高引起了人们的关注,因为目前的GS方法仍然忽略基因的功能或生物学相关性以及它们与QTN和QTL的关联,并且仅使用基因组数据来构建基因型和非基因型亲属之间的基因组关系矩(GRM)。人们试图评估单核苷酸多态性(SNP)组对性状的总遗传方差的相对贡献,例如内含子,外显子,基因间变异体,同义或非同义变体类别,总的来说,似乎没有显著差异。当一个性状明显受到主要数量性状基因位点(QTL)影响时,贝叶斯方法(如Bayes Cpi和Bayes R)往往比基于GBLUP的方法提供更准确的预测。系统基因组BLUP(sgBLUP)方法的概念是基于一个全基因组的预测和选择的混合模型。sgBLUP使用两种类型的SNP,即功能注释与所涉特定特性相关的SNP(例如,GWAS和后GWAS SNP注释软件)和称为“残基SNP”的SNP。它们通常用于构建GRM的SNP,可以跨所有性状使用。
基因组学和表观基因组学的新兴技术
基因组和表观基因组领域的技术迅速发展,并为研究基因组或表观基因组以及在动物育种方法中进一步实施的新方法提供了机会。随着NGS技术的强大功能和发展速度,现在已经在数千万个基因组位置的DNA序列水平捕获了全基因组的遗传变异。表观遗传变异还通过组蛋白修饰和基因水平的DNA甲基化而导致表型变异,这可能导致构成表型或疾病的基因表达发生变化或缺失。 NGS技术正在迅速发展,例如,与限制性位点相关的DNA测序(RAD-Seq),最近这一技术在鸡身上得到了应用,显示了NGS技术在动物育种问题上的有效性。这些由最新技术产生的数据将有助于识别(新颖的)因果或调控变异,精确的全基因组LD模式以及插入缺失(InDel)标记,这将对基于分子的动物育种计划很有帮助。测序基因型(GBS)是为植物育种而开发的另一种新颖的NGS技术,但由于其成本效益而具有动物育种的潜力。然而,其达到合理的预测准确性和最小偏差的潜力取决于测序深度和使用GBS进行测序的个体数量。
同样,使用NGS技术进行CNV分析对表型变异的研究也有重大影响。这种CNV研究已经发生了范式的转变,最初的全局特征扩展到了导致综合图的深入研究。虽然这种富集分析依赖于良好的模拟和生物信息学分析,但这些结果将产生大量的动物数据,这将有助于人类疾病的研究。为了确保适当的基因在动物中的表达,有必要确定半甲基化(CpG)和CGI在各种动物组织中存在的频率或位点,现在这可以通过全基因组DNA甲基化分析来实现。
通过组合用于生成NGS数据的不同技术方法,可以分析全基因组甲基化模式和符合单核苷酸分辨率的图谱。其中一种方法是减少亚硫酸氢盐测序(RRBS),因为它允许将限制酶和亚硫酸氢盐结合在一起,只对基因组的约1%进行测序,因此具有成本效益。
染色质免疫沉淀(ChIP)与高通量测序相结合,可用于整合并有效鉴定体内蛋白质-DNA结合位点。将ChIP-Seq数据与表达数据结合起来,将使我们能够解开全基因组模式并捕获调控子和调控网络。此外,近来用少量固定的动物组织代替培养的细胞进行ChIP-Seq成为可能,这将允许对蛋白质-蛋白质相互作用(PPI)进行系统分析。
此外,基于双重发光的免疫共沉淀(DULIP)使得检测PPI具有较高的特异性和灵敏性。从ChIP-Seq方法或其他测定方法(例如DULIP)获得了共免疫沉淀的荧光素酶标记,并进一步用于支持和理解蛋白质功能和复杂的生物学过程。随着这样的进步,整合突变依赖性结合模式(蛋白质或DNA)的全系统分析将变得可行。有趣的是,这些方法如何改进候选基因,药物靶标和生物标志物的识别,以及捕获完整的遗传和表观遗传变异以准确预测表型。目前,这些高通量技术提供了许多更好地理解复杂数量性状和基础(系统)生物学的机会。余下的挑战是克服在发现高复杂疾病和具有农业意义的特性的因果基因和变异、药物靶点、疫苗和生物标志物方面的困难。
动物转录
转录组学研究了所有基因转录本在特定细胞、特定时间和特定状态下的表达水平。基因上调与下调在不同水平的蛋白质和代谢物中诱导动物表型变化。因此,更好地理解基因的调控,有助于深入了解在疾病或生产性状中重要的基因的生物学功能和检测。分析表达数据的最常见方法是比较两种状态之间的表达水平,例如健康动物与疾病动物或高产动物与低产动物的比较,也称为差异表达分析。几项研究集中于检测不同物种中不同生产和健康性状的差异表达(DE)基因,例如肉鸡的肥胖,牛的肌肉发育,猪的骨骼肌发育和绵羊肠道寄生虫抵抗力。在受控条件下对动物进行实验,并对其进行解剖,以收集各种组织样本(例如脑组织),这在人类研究中几乎是不可能的,最近报道了一个牛的基因组学计划,该计划研究了一大群动物的脾脏,胎盘和脑组织的表达数据。在不同组织上的表达研究可以使人们更好地了解家畜健康和生产性状的病理生理。
除DE分析外,转录组学研究还使用网络方法来分析基因与基因的相互作用,该方法侧重于检测共同调控基因的簇。一种常用的方法是在R包中完成的加权基因共表达网络分析(WGCNA)。它利用Pearson的相关性来检测基因的共表达,并计算拓扑重叠度量(TOM),该度量表示跨基因对的共享相邻基因的数量。基于这种测量,对基因进行聚类,并进一步将这些聚类与表型数据联系起来,以揭示与所研究性状的生物学背景有关的重要途径。一些家畜转录组学研究已经使用这种方法阐明了健康和生产性状的遗传和生物学背景。在绵羊体内,检测到许多与肌肉和肠道寄生虫抗性有关的途径。在猪体内,发现了影响肌肉和肉品质的途径和基因,在韩牛体内,检测到了与肌内脂肪相关的基因。
转录学新兴技术
跟随人类研究的趋势,对家畜物种进行的转录组学研究正从微阵列表达数据向RNA测序(RNA-Seq)数据转变,并为检测新的转录本和遗传变异提供了新的机会。使用RNA-Seq,我们现在可以鉴定和定量:同工型,外显子特异性表达,等位基因特异性表达和单倍型特异性表达。 Malone和Oliver详细讨论了RNA-Seq数据与微阵列数据的比较及其优势。 Nookaew等人使用了来自两种类型平台的酿酒酵母中的真实表达数据进行了这样的比较,结果表明基于微阵列和RNA-Seq技术的发现是一致的。与微阵列研究一样,最常用的方法是检测DE基因,就像在牛,马和猪中所做的那样。如Lee等人所报道的,DE分析结果可用于系统生物学方法。其他几项研究通过使用RNA-Seq的基因共表达网络(GCN)方法,向前迈进了一步。在Nellore牛体内,第一项研究检测到了八个DE基因的饲料效率,而第二项研究应用WGCNA分析了饲料效率的遗传结构,并证明了共表达(CE)基因主要与胰岛素反应和脂质代谢相关。这些发现与来自肝脏组织病理学分析的数据相结合显示,低饲料效率的动物比高饲料效率的动物肝损伤更高。正在使用不同品种的牛(荷斯坦奶牛和泽西奶牛)进行类似的项目,这些牛种的饲料效率表型非常高。一种基因共表达网络(GCN)方法应用于猪身上,以检测共表达的基因簇,这些基因簇与背脂雄烯酮表型,沙门氏菌脱落和肥胖相关基因有关。
通过辅助生殖技术进行的动物育种,例如体外胚胎生成(IVEP)与基因组选择相结合,可快速改善遗传状况。与IVP,胚胎移植和随后的妊娠率相关的卵母细胞,胚胎性状的转录组学和系统生物学研究已经检测出成功IVP,胚胎移植和妊娠率的生物标志物。
在猪生产中,挑战之一是减少公猪异味,即在烹饪过程中散发出的令人讨厌的臭味,这使得消费者不满意。这种气味主要由两种化合物引起,即睾丸中产生的雄烯酮和后肠中产生的粪臭素。这两种化合物都主要在完整雄性的背部脂肪中蓄积,而减少公猪异味的唯一方法是通过手术阉割。但是,手术阉割引发了严重的猪的保护问题,导致了欧洲自愿禁止手术阉割以避免公猪异味。雄烯酮和粪臭素水平的遗传力是中等至高的,这表明雄性猪低异味育种的潜力很大,从长远来看可能解决(猪)保护问题,因为完整的雄猪可在食物链被食用。GWAS已检测到几个与雄烯酮和粪臭素水平相关的基因组区域。结果表明,公猪异味与生长性状和产仔数没有显著相关性,甚至与雄性生育率也没有显着相关性。对公猪异味重或轻的RNA-Seq转录组谱分析的最新研究表明,某些基因的表达谱存在关键差异。在此基础上,我们的研究现在集中在丹麦猪公猪异味的RNA-Seq转录组学和系统生物学上,以鉴定DE和CE基因,并构建GCN以提升丹麦的猪肉产量。为了更深入地了解特定性状或疾病的调控结构,可以结合或整合几种方法来阐明基因与基因的相互作用。
另一种相对新颖和有前途的方法是单细胞转录组分析,可对细胞的基因转录进行深度测序。主要在细胞发育研究具有潜力(例如干细胞研究)以及在采集足以用于RNA-Seq转录组学的活检样本的情况下(例如,在将胚胎移植到供体牛之前进行胚胎活检),这种更深刻的见解可以使人们更好地理解基因组研究与表型研究之间的联系。尽管可以利用RNA-Seq分析开发多种基因和转录本,但主要的挑战是鉴定真正的积极因素。如果不使用适当的过滤技术,基于作图和基因组组装的方法可能会错过一些候选基因。另一个挑战是量化G/C块,paralogons, 等容线,5'UTR区,剪接变异体特有的表达,外显子特异性和等位基因特异性表达。基于RNA-Seq的转录组学研究能够进行非编码RNA的研究,但是如何将这种非编码RNA的研究用于系统基因组学方法尚待探索。在过去的几年中,对包括miRNA在内的小RNA进行测序的工作越来越多。自RNA-Seq首次应用以来,量化小RNA,确定交替剪接事件,转录起始位点(TSS)和链特异性基因定位都得益于此类技术。目前许多研究正在为miRNA-Seq和其他小型RNA测量提供严格的方法,但这超出了本文的范围。
动物代谢组学和蛋白质组学
蛋白质组学旨在描述生物体中蛋白质的完整组成部分,而代谢组学则是对生命系统中整体代谢产物的研究。尽管代谢组学术语和代谢组学这两个术语的使用仍存有争议,但对代谢组进行的分析是一项艰巨的任务,因为它考虑了所有代谢物,无论其化学成分如何,即氨基酸,抗体,适体,小生物分子等,并以整合的方式提供连贯的基因表达数据。代谢组学不仅可以作为细胞内代谢物的定性数据,而且可以作为定量数据的来源,这些数据对于在体内运行的代谢网络进行基于模型的描述至关重要。近年来,一些关于家畜的研究对代谢组进行了探讨,现在代谢物谱研究已成为动物和兽医基因组学领域中一个迅速发展的领域。代谢组学工具旨在通过允许同时监测生命系统中的分子来填补基因型和表型之间的差距。此类代谢信息可应用于临床实践,发现与细胞完整性,由细胞损伤或死亡引起的细胞和组织动态平衡有关的生物标记物,以及在代谢工程中优化微生物的生物技术。
在奶牛体内,通过研究不同体液的代谢产物,发现了许多潜在的用于乳牛产奶量和品质生物标志物。同样,在鸡体内,通过对肝脏代谢组的研究,发现了几种可能的腹水综合征生物标志物。代谢组学的另一种潜力是预测具有经济意义的表型,如对猪的报道。
蛋白质组学和代谢组学的新兴技术
蛋白质组和代谢组学数据集提供了大量多维数据点,需要仔细地对其进行质量控制,分析和解释。在基因组学和转录组学中,有各种各样的公共数据库和工具可用于存储,查询,浏览,分析和可视化代谢组学网络。例如,PathCase代谢组学分析工作台在人工创建的通用哺乳动物代谢网络上运行。蛋白质-蛋白质相互作用(PPI)网络到表型和疾病途径的映射是理解各种生物学和病理生理过程的关键。这种相互作用研究可以与远距离的非编码RNA的保存及其在哺乳动物基因组中的潜在功能的研究结合起来。
总体而言,此类关联网络,集合体和交互作用研究揭示的生态系统受到各种环境条件的挑战。在这一过程中,出现了一个新的领域,称为“生态学”,该领域涉及生物群与其非生物和生物环境的相互作用,并受到以以生物信息学为中心的方法的元组学法的发展的推动。这可能会导致一个新的“组学”术语,即系统组学定义为分子系统生态学,这不仅有助于理解哺乳动物或动物的动力学,而且有助于理解依赖于系统级响应的微生物过程。
最近,发起了许多结构性蛋白质组学研究,以确定生物化学和细胞功能,并允许在分子水平上设计药物。使用的方法包括硬件设计,数据采集方法,样品制备和数据分析的进一步自动化方面的进步。40%-50%的识别基因与功能未知的蛋白质相对应,开发了一种核磁共振功能注释筛选技术(NMR)(FAST-NMR)来分配生物学功能。这些方法假定可以基于蛋白质之间结合区域的相似性来描述生物学功能,并且给定的配体与目标序列相互作用。最终的蛋白质结构和功能分配可以为药物的发现提供起点,也可以为调控性或非调控性区域提供功能线索。然而,功能蛋白质组学/代谢组学已发展成为必要的下一步,对于该步骤,由于现有的方法仅用于序列同源性检测,而不能推断出大量序列库的功能,因此核磁共振波普法被用于研究大量序列库的功能。此外,蛋白质/代谢物的三维结构对推断分子功能(物理和化学功能)有很大的帮助。我们可以预测,未来的系统基因组学将包含大规模蛋白质组学/代谢组学,作为提供与表型变异相关的附加层。
功能注释与路径分析
在前面的部分中,我们讨论了各个“组学”平台和数据集的现状和新兴趋势。无论使用哪种“组学”平台,基因本体(GO)注释都是使用标准化词汇分配功能信息的最重要和最有价值的方法。有几种计算方法和工具可用于所有物种的功能注释。基于基因的注释可以识别SNPs或CNV是否造成蛋白质编码变化。为此,使用了诸如RefSeq基因,UCSC基因,ENSEMBL基因,GENCODE基因的基因定义系统。
基于基因组区域的注释可识别特定基因组区域中的变体,例如,保守区域,基于NGS的DE/CE区域,转录因子结合位点,GWAS区域等。尽管基于相似性的GO注释被广泛应用,但它主要包括序列具有最佳匹配的数据,可以从大量的多基因组学数据中预测候选目标。但是,这些序列的一些同源词与GO术语没有关联,并且可以与保守域进行交叉验证,手动查看数据或通过湿式实验室实验确定,从而使功能分配具有生物学上的适当性。未加注释的区域以假设蛋白质或“已知未知数”的形式存在,即它们的存在被预测但其功能未知,代表着一个很大的问题。设计了几种方法来将不同的结构和功能结果与对应生物体GO关系的数据进行整合。
此外,在获得新信息时,许多物种的基因组组装会定期被细化和更新。提供的综合分析的发展有所增加。对基因组组件进行全面且有力的GO注释,为其他基因组的功能性查询提供了坚实的基础。绘制路径图并确定其独特性和新的信号改变了牛体内转化通路关联研究。此外,医学标题为人类和模型生物的研究提供了全面的生命科学词汇。多方面“组学”是由选择注释和丰富的分析以来解释GO-aided MeSH功能术语。总之,此类GO注释对应于特定物种的特定生物学条件或复杂性状。
路径分析可以描述为“一组利用路径先验知识的统计方法”。它构成了“组学”结果与正在研究的表型/疾病之间的联系,并为检测到的基因和变异提供了生物学意义。 此外,它减轻了多重测试的负担,因此,具有巨大的分析潜力。 Aslibekyan等人研究表明,尽管途径分析具有很大潜力,但仍有许多障碍需要克服(例如,由于缺乏黄金分析标准)。
路径分析和遗传网络方面的新兴技术
尽管多维HTO技术致力于理解表型变异,但关于从生物系统变异到表型变异的转化所基于的背景路径的推断仍然存在重大的科学瓶颈。最近,已经报道了一些解决功能模块之间相互作用的特征的复合模型。例如,路径网络分析方法(PANA)利用机器学习方法集成了高通量数据及其功能注释。最终用户可以检测分子系统内相关的功能模块以及疾病或表型中的转录连接。分子系统生物学以路径,相互作用和/或关联的形式整合网络。关联仅被推断为关系内的链接,而以生化实验为形式的物理关系则被推断是相互作用。然而,所有交互都是相关联的但并非所有关联都是交互的范例可以广泛应用于所有功能模块。如前所述,考虑到各种生化路径和反应,模型旨在分析和测量细胞内存在的分子数量。然而,所使用的模型取决于检测的类型,表观遗传修饰是如何从转录组数据中推导出来,是否考虑疾病风险以及与表型性状相关的遗传异质性类型。
许多研究表明了遗传相互作用的重要性,尤其是在确定复杂的多基因性状方面,这为网络遗传学提供了巨大潜力。通常,GWAS研究不同个体的基因组以检测与性状相关的变异,但是没有考虑基因座之间的相互作用。然而,关于特定性状的SNPs之间的全基因组相互作用的研究正在进行,例如,参考关于婆罗门牛胴体相关性状的论文。下一步是在包括上位性相互作用网络方法中,例如使用加权交互SNP集线器(WISH)网络方法,该方法已成功应用于猪资源种群中以检测与人类肥胖有关的基因和路径。
该方法(加权交互SNP集线器(WISH)网络方法)通过设置一个比标准GWAS低地多的显著性水平,基于其全基因组显着性水平预选SNPs,并用于在聚类方法中对所有SNP之间的上位性相互作用的后续计算。
另一种方法是关联权重矩阵方法(AWM),该方法通过根据SNP之间估计的累加效应的大小来查看SNP之间的相互作用,从而整合了来自多个GWAS的数据。这种方法已经成功地鉴定了牛生长的基因和路径,也成功地鉴定了热带黄牛品种青春期发育的基因和途径。
最新报告显示了lncRNA如何促进与其非编码对等体(如miRNA)的调控相互作用。 lncRNA-蛋白质网络是否抑制相互作用尚不清楚。 lncRNA和蛋白质之间的这种调节相互作用如何对有机体产生重大影响是关注的焦点。最近,我们的小组报告了一组与RNA-Seq数据构建的交互网络相一致的IncRNA-蛋白质结合的检测。这些研究将使我们了解这种关联网络如何促进各种细胞器中的转录调控。此外,将网络方法和路径分析应用于与多种疾病和表型相关的基因将使研究人员对病理生理学和生物学过程有更深入的了解。
从基因组到基因组的多组数据:系统基因组学的集成
在动物育种背景下,“系统遗传学”或“系统基因组学”一词最初是由Kadarmideen等人提出的。但有关该主题的最新评论在人类和动物中都有应用。正如这些文章中所详细讨论的那样,系统遗传学/系统基因组学专注于整合不同的“组学”水平。包括从单基因水平和路径分析水平上,将个体的“组学”水平与功能注释相关联到所有不同的multi-omic水平整合为表型的多种方法。典型的数据整合过程从基因组→表观基因组→转录组→代谢组→蛋白质组→表型或疾病变异。
利用网络调控对蛋白质丰度/mRNA表达的相关数据的整合进行了研究,探讨了与牛青春期有关的基因表达。基因组和转录组数据的整合,例如利用表达定量性状位点方法(eQTL),可检测基因组中与转录水平相关的区域。这些eQTL可以是顺式也可以是反式的:一个顺式eQTL位于编码该转录本的基因附近,而一个反式eQTL则相距甚远,甚至位于另一条染色体上。
一些在猪身上的研究已经采用这种方法来检测候选基因,例如肌肉特征和肥胖表型。一项在大鼠,小鼠和人类中对异质种群(HS)进行的多亲本群体的研究确定了靶向候选基因,并将其定位于疾病表型中。在这项研究中,作者采用了差异表达分析和eQTL分析,基于动物序列的混合模型分析,识别了在被检测区域内的变异。尽管这样的研究可以检测出致病基因中的变异,但是这些致病基因是否单独在这些复杂的表型中起作用仍然是一个挑战。仍然存在以下问题:
*这些eQTL对于构成表型变异的遗传网络的研究有多重要?
*这些转录分析产生的eQTL数据能否与蛋白质组学相关联?尽管转录数据和表型数据可能看起来不太相关,但是定位基因表达的遗传决定因素(eQTL)可以为理解大的表型效应并将遗传变异与疾病联系起来提供一个显著的框架。
与Cas9蛋白(CRISPR/Cas)系统结合在一起的,定期排列的短回文重复序列(简称CRISPR)引导RNA进入到细胞的基因组(核酸酶)中并在特定位置切割基因组(该技术通常称为基因组编辑)。自Cong等人首次提出以来,已经在各种组织,大型动物模型和种群中进行了多次定点诱变实验。与早期的转录激活因子效应核酸酶和zinc-finger核酸酶系统相比,这种定点基因组编辑技术大大提高了获得基因组修饰的精度。
迄今为止,它尚未在动物和兽医科学中得到广泛使用,但显然可以预见的是,利用CRISPR/Cas系统对动物基因组进行修饰将在提高抗病性或性状表现或创造“designer animals”如转基因动物方面发挥关键作用。另一个应用是一旦验证了因果基因/ QTL,则可以通过使用CRISPR-Cas系统对其进行特殊编辑。这类对生物体遗传结构的修改才刚刚开始,还未在动物身上进行充分研究。一项有关通过CRISPR /Cas9介导的人cDNA在合子水平上敲入猪白蛋白位点而产生的人白蛋白的最新研究说明了该技术在动物研究中的潜力。使用RNA-Seq转录组学研究推断CRISPR介导的系统在进一步推动该技术中发挥出关键作用。
动物系统基因组学远远没有充分利用NGS技术的功能。例如,RNAseq不仅可以提供基因表达水平的精确测量,还可以提供有关异构体,外显子特异性表达,等位基因特异性表达和单倍型特异性表达的数据。RNA-Seq在系统基因组学研究中的优势尚未得到充分利用,例如在eQTL研究中,Wang等人报道了通过利用RNA-Seq数据建立的Lyon hypertensive(LH)大鼠在eQTL定位中的应用。在eQTL研究中整合到RNA-Seq的这些额外优势在很大成熟上程度仍然是未知的,但是我们可以预知eQTL研究的出现,该研究将在整合系统基因组学中使用这类信息。
在过去的十年中,已经出现了检测代谢QTL的方法,这些方法基于利用大规模分析法对代谢物和小分子进行表征。此类方法使研究人员可以更好地了解跨代谢网络的生化路径。通过被称为“代谢型”的新陈代谢的表型状态来分析环境是否对新陈代谢产生影响。这种代谢组学/代谢型数量性状位点(mQTL)的定位和代谢组学全基因组关联研究(mGWAS)已广泛应用于获取遗传多态性研究中的信息,例如,参考心血管疾病的人类研究。然而,缺乏一个像R-package mQTL.NMR中开发的那样的用于了解高分辨率代谢组学的多组学融合的综合框架,该框架没有针对基因组和代谢组学特征的综合分析框架来表征混合系统。 mQTL或mGWAS的原理也可以应用于影响动物特定疾病表型的已知代谢物列表(例如由多达200种化合物组成的低通量代谢物谱)。例如,通过F2杂种猪的组合连锁不平衡连锁分析(LDLA),确定了几个影响肥胖和与肥胖相关表型的代谢产物的QTL,随后研究了与这些鉴定的mQTL相同的人类染色体区域。关于这项工作的进一步讨论超出了本文的范围,但是显然大规模代谢/代谢组表型数据的质量控制和分析对动物遗传学研究提出了巨大挑战。另一个新的研究领域是影响蛋白质丰度(pQTL)的遗传变异的定位,迄今为止,该变异已成功应用于F2小鼠种群并用于细胞对化学的反应分析。这样的发展为识别动物疾病和生产性状的生物标记物提供了巨大的机会。无论哪种类型的QTL或SNP检测到了动物性状(eQTL,mQTL,pQTL),都可以将它们整合到模型中,该模型旨在了解/检测基因组中因果关系和调控基因位点。
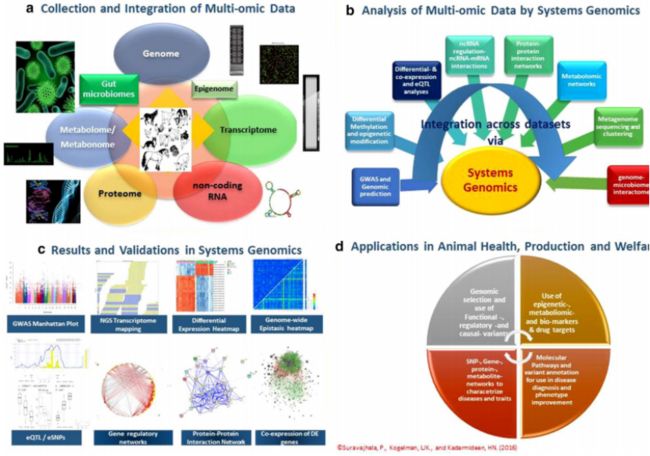
图1总结了前面有关多基因组学数据整合和系统基因组学的讨论。它说明了各种HTO平台如何生成和整合多基因组学数据的,从基因组水平到各种“组”再到动物表型或疾病变异数据库。它显示了在系统基因组学框架内的单个,单层“组”分析以及多层“组”分析。单层基因组分析或转录组分析的典型结果可以通过GWAS可视化-Manhattan plots,NGS数据的变异位点,RNA-Seq转录组数据的转录计数,全基因组上位位点和差异表达位点等。
对于来自“组学”平台之一的给定HTO数据集,在将给定HTO数据集与另一个HTO数据集合并之前执行单层特定分析。例如,在整合这些HTO数据集以识别eQTL或eSNP之前,应相互独立进行GWAS分析和差异表达分析或共表达分析(DE /CE)。这也适用于受QTL或SNP(mQTL或mSNP)影响的代谢物或代谢物谱(NMR peaks)。进行表观遗传学研究以确认差异甲基化区域,并将这些分析和结果与DE/CE分析的转录组分析相结合。两组或多组基因分析的典型结果包括eQTL图谱,eSNP效应,mQTL或mSNP效应,定向基因调控网络(使用SNP数据),以多组基因学数据作为证据构建的PPI网络,基因共表达网络被定位为顺式和反式作用的eQTL或差异表达的基因。这些多组基因学数据分析的应用包括基因组预测/选择,使用功能,调控和因果变异,为疾病预防和诊断或整体性状改进的高精度检测。表观遗传标记,代谢组学特征或图谱,生物标记物和药物靶点的利用非常适合于兽药的疾病预后/诊断/治疗和临床干预,因此有助于改善动物的健康和增益。变异的分子路径和注释可用于疾病诊断和表型改善。系统基因组学通常以几种类型的相互作用或关联网络为特征,包括SNP-SNP,基因-基因,蛋白质-蛋白质,代谢物-代谢物网络,这些网络表征了动物的特定疾病或健康状况或表现水平。这些网络提供了对组织内部和组织之间相互作用的深入了解,以及有关主要调控基因或变异,代谢产物或蛋白质(“集线器”)的信息,这些信息可用作疾病诊断和表型改善的预测标记物。
图1通过基于阵列或光谱技术或NGS技术和系统基因组学分析创建的具有各种“组学”平台/数据类型的集成基因组学的概述。
a.在受控实验条件下或野外实验中,农场或同伴动物中多种类型的“组学”数据集的集合。b.系统基因组学包括从GWAS,差异表达或甲基化分析到蛋白质组/代谢组学数据到eQTL / mQTL / pQTL和网络分析的单层(垂直箭头)和多层“组学”数据集(水平箭头)的分析。c.系统基因组学的典型结果包括来自GWAS Manhattan plots,全基因组上位位点,NGS数据中的变异检测或转录本计数,eQTL基因调控或共表达网络的基因表达位点的单层和多层分析,蛋白质-蛋白质相互作用(PPI)网络与差异连接基因的网络之间的关系。Esnp/eQTL框图取自Kogelman等人的研究。该报告中的其余图片来自作者自己未发表的材料。
d.这种方法的潜在应用包括鉴定因果基因或路径,生物标志物,药物靶点,特定性状水平或疾病状态的各种网络以及对表现或疾病风险的个性化基因组预测。
结论
本文综述了基因组学/表观基因组学,转录组学,代谢组学和蛋白质组学领域中的现有的和新兴的技术,并提供了一些牲畜物种的实例。在“序列空间”和多组基因学与对动物和兽医生物科学注重背景下,我们重新引入系统遗传学/基因组学。由于(例如排序)技术的巨大进步,数据生成变得越来越简便和容易,从而导致在不同的“组学”水平产生大量数据。在过去的几十年中,检测的数据以来阐明构成动物生产,健康和增益特性的生物学机制。
这使人们对(潜在的)生物标志物和疫苗的机制和检测有了更深入的了解,并改进了动物育种策略。我们简短地提到了现有的和新兴的不同“omic”技术及其在牲畜物种中的应用(包括基因组学,表观基因组学,转录组学,代谢组学和蛋白质组学)。剩下的挑战是通过良好的质量控制方法为数据集的每一层消除错误/干扰,有效地使用所有这些“omic”级数据集,根据定义的系统基因组学假设和统计模型进行适当的数据整合,应用高级统计生物信息学算法和有意义的结果解释。这些综合方法的明显优势是提高了检测真正的因果基因,调控网络和路径的能力,从而提升了动物的健康,增益和/或生产。因果基因或变异(QTN或QTL),调节基因,生物标志物和基因网络等结果应纳入基因组选择和育种计划中以产生更大的影响。随着基因组选择方法越来越倾向于将各种类型的QTL信息纳入基因组预测模型中,这些前景变得越来越可行。通过这种延伸,生物学和功能上有意义和准确的基因组选择方法,动物生产,健康和增益的提高将更快,更可持续。