机器学习算法/模型——有监督到无监督(降维):主成分分析(PCA)
主成分分析 PCA
- 0. 本质和概述
-
- 0.1 本质
- 0.2概念/术语
- 1. 数据降维(建立直觉)
-
- 1.1 降维
- 1.2 降维本质
- 2. 主成分分析(PCA)
-
- 2.1 两个目标
- 2.2 一个思路
- 2.3 两种方法
- 3. 求解主成分
-
- 3.1 分解协方差矩阵
- 3.2 奇异值分解
- 4. 代码
-
- 4.1
- 4.2
- 5. PCA小案例
PCA是一种无监督的学习算法,它是线性模型,不能直接用于分类和回归问题。
0. 本质和概述
0.1 本质
核心:向重构误差最小(方差最大)的方向做线性投影
PCA是一种数据降维和去除相关性的方法,它通过线性变换将向量投影到低维空间,而同时保证在低维空间中的投影能很好的近似表达原始向量,即重构误差最小化。
上图是主分量投影示意图,样本用红色的点表示,倾斜的直线是它们的主要变化方向。将数据投影到这条直线上即完成数据的降维,把数据从2维降为1维。
0.2概念/术语
- 维度灾难
- 特征空间
- 数据稀疏
- 超平面空间 = 原空间的子空间
向量空间都有对应的超平面,而超平面的维度低于其所在空间。(这也是降维的任务所在)
1. 数据降维(建立直觉)
1.1 降维
机器学习领域里讲的降维是指:采用某种映射方法,将原本高维空间中的数据样本映射到低维空间中。
降维算法多种多样,比较常用的有:PCA(Principal Component Analysis,主成分分析)、LDA(Linear Discriminant Analysis,线性判别分析)、LLE(Locally linear embedding,局部线性嵌入)、Laplacian Eigenmaps (拉普拉斯特征映射)等。
1.2 降维本质
降维的本质是学习一个映射函数 y=f(x)
,其中 x 表示原始的高维数据,y 表示映射后的低维数据。
下面介绍一个数据预处理常用的方法:数据主成分分析,简称PCA。
2. 主成分分析(PCA)
把一个空间中的样本点映射到它的超平面上,映射后的结果就只存在于超平面空间(也就是原空间的子空间)这样,我们就获得了降维的结果。

如上图,几个二维数据点可以投影到任意一个直线上面完成降维。(黄线比蓝线更接近主成分的方向)
(回归头来才发现,原来这么简单的道理:因为黄线的方向比蓝线方向更能代表原来的数据的走势!)
怎么能够保证超平面中的点不丧失对应原始点的“主成分”呢?看下面的几个点。
2.1 两个目标
- 特征减少(减少维度)
- 损失要小(抓住主要成分)
2.2 一个思路
① 利用变换,去除原始数据的相关性,将原特征转变为线性无关的新特征
② 舍弃不重要的特征,倾向于选方差大的特征作为主成分
为什么要去除相关性——相关性越大,冗余信息越多
为什么选方差作为判断标准——方差越大,信息量越大
2.3 两种方法
- 分解协方差矩阵来求取
- 绕开协方差矩阵对原始数据矩阵进行矩阵分解
3. 求解主成分
3.1 分解协方差矩阵
3.2 奇异值分解
注:除了可以用于 PCA 的实现,SVD 还可以直接用来降维。
4. 代码
4.1
- 模块导入
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns; sns.set()
- 准备数据
rng = np.random.RandomState(1)
X = np.dot(rng.rand(2, 2), rng.randn(2, 200)).T
plt.scatter(X[:, 0], X[: ,1])
plt.axis('equal')
- ① 数据主轴的可视化(数据分析)
设置 n_components=2
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
pca.fit(X)
print(pca.components_) # “成分”
print(pca.explained_variance_) # 可解释差异
# 可视化元数据的主轴
def draw_vector(v0, v1, ax=None):
ax = ax or plt.gca()
arrowprops=dict(arrowstyle='->',
linewidth=2,
shrinkA=0, shrinkB=0)
ax.annotate('', v1, v0, arrowprops=arrowprops)
# plot data
plt.scatter(X[:, 0], X[:, 1], alpha=0.2)
for length, vector in zip(pca.explained_variance_, pca.components_):
v = vector * 3 * np.sqrt(length)
draw_vector(pca.mean_, pca.mean_ + v)
plt.axis('equal');
- ② 进行降维
设置 n_components=1 (降低一个维度)
# 变换的数据被投影到一个单一维度。
pca = PCA(n_components=1)
pca.fit(X)
X_pca = pca.transform(X)
print("original shape: ", X.shape)
print("transformed shape:", X_pca.shape)
为了理解降维的效果,我们来进行数据降维的逆变换,并且与原始数据一起画出
X_new = pca.inverse_transform(X_pca)
plt.scatter(X[:, 0], X[:, 1], alpha=0.2)
plt.scatter(X_new[:, 0], X_new[:, 1], alpha=0.8)
plt.axis('equal');

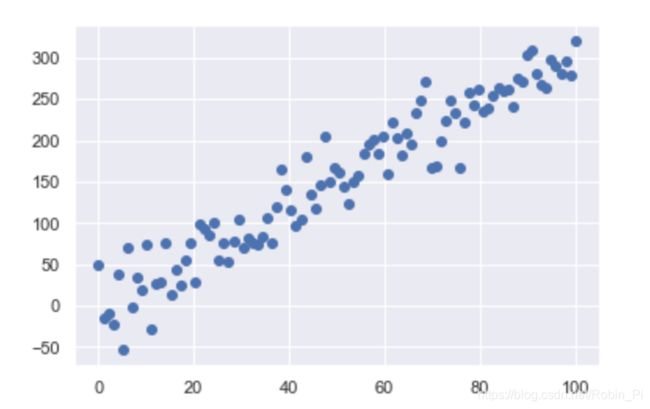
我们可以很清楚地看到 PCA 降维的含义:沿着最不重要的主轴的信息都被去除了,仅留下了含有最高方差值的数据成分。被去除的那一小部分方差值(与主轴上分布的点成比例,如图所示)基本可以看成是数据在降维后损失的“信息”量。
——被损失的是局有相关性(成比例)的那部分数据。(如下图标出的黑线部分:橙色点和蓝色点在不重要的主轴上基本成线性关系)

4.2
- 模块导入
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns; sns.set()
from sklearn.decomposition import PCA
- 数据准备
rng = np.random.RandomState(1)
x = np.linspace(0, 100, num=100)
y = 3 * x + 30 * rng.randn(100)
print(x.shape, y.shape)
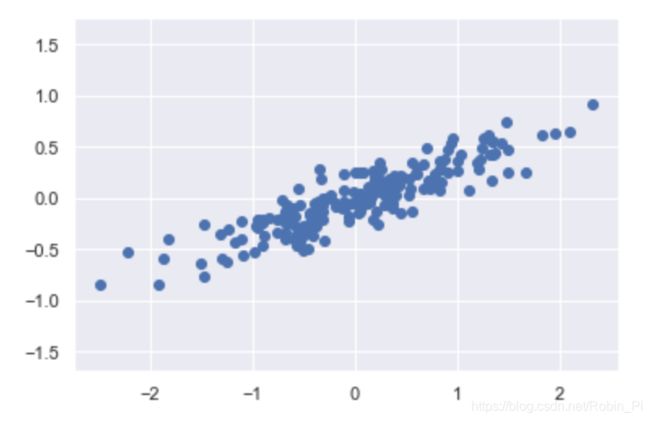
plt.scatter(x, y)
直接通过直线方程准备我们需要的数据
调整数据的 shape,并合并:
x = x.reshape(-1, 1)
y = y.reshape(-1, 1)
X = np.hstack((x, y))
print(X.shape)
- 拟合数据
pca_model = PCA(n_components=1)
pca_model.fit(X)
- 数据转换:降维
# 数据转换:二维数据投影到了一个单一维度
X_pca = pca_model.transform(X)
print(X_pca.shape)
- 逆向转换:进行可视化
X_new = pca_model.inverse_transform(X_pca)
print(X_new.shape)
plt.scatter(X[:, 0], X[:, 1])
plt.scatter(X_new[:, 0], X_new[:, 1])
5. PCA小案例
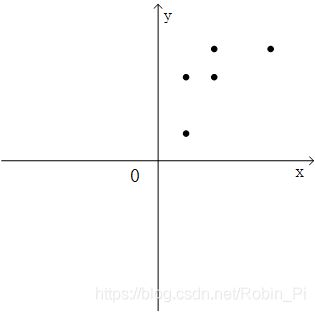
假设我们研究的对象有两个特征属性X和Y,对 5 个样本进行数据采样的结果如下:
| X | Y | |
|---|---|---|
| 样本1 | 2 | 2 |
| 样本2 | 2 | 6 |
| 样本3 | 4 | 6 |
| 样本4 | 8 | 8 |
| 样本5 | 4 | 8 |
我们的目标是对其降维,只用一维特征来表示每个样本,只用一维特征来表示每个样本。我们首先将其绘制在二维平面图中进行整体观察:
查看这两个变量的协方差矩阵,
import numpy as np
import matplotlib.pyplot as plt
x = [2,2,4,8,4]
y = [2,6,6,8,8]
S = np.vstack((x,y))
print(np.cov(S))
[[ 6. 4.]
[ 4. 6.]]
结合之前的二维散点图可以发现5个样本的特征 X 和特征 Y 呈现出正相关性,数据彼此之间存在着影响。
若直接粗暴地去掉一个特征,可行么?则会变成:

显然效果不理想:忽视了数据中的内在结构关系,并且带来了非常明显的信息损失。
(降维——>高维数据向低维进行投影)
一个解决思路便是:
① 去除原始特征的相关性,使用心新的一组特征来表示原始数据
② 然后从新的彼此无关的特征中舍弃不重要的特征,保留较少的特征,实现降维。
首先,第一点的目的是使用新的特征来对样本来进行描述,为了让这两个新特征满足彼此无关的要求,就需要让这两个新特征的协方差为0,构成的协方差矩阵是一个对角矩阵(原始特征X和Y的协方差不是0,只是一个普通的对称矩阵)
对变量分别进行0均值处理后,通过求解协方差矩阵的特征向量,就可以得到线性无关的特征矩阵(图中两个新的坐标方向)。
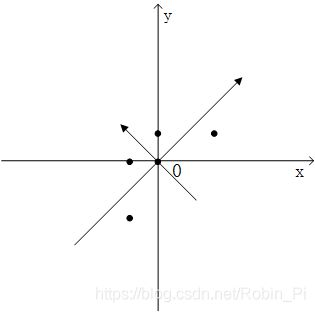
接下来的工作就是从这两个特征中选取一个作为原始数据的特征表达,其判断标准是方差,方差越大表示这个特征里的数据分布的离散程度就越大,特征所包含的信息量就越大。
参考
- 机器学习数据预处理:数据降维之PCA
- 数据预处理:PCA原理推导
- 数据降维处理:PCA之特征值分解法例子解析
- 数据降维处理:PCA之奇异值分解(SVD)介绍
- 主成分分析(上):利用特征值分解
- 主成分分析(中):奇异值分解的原理与通用性
- PCA——利用数学工具提取主要特征
- 矩阵特征值分解与主成分分析(Python 实现)