数学概念
方差:用来衡量随机变量与其数学期望(均值)之间的偏离程度。统计中的方差(样本方差)是各个数据分别与其平均数之差的平方的和的平均数。
协方差:度量两个随机变量关系的统计量,协方差为0的两个随机变量是不相关的。
协方差矩阵:在统计学与概率论中,协方差矩阵的每个元素是各个向量元素之间的协方差。特殊的,矩阵对角线上的元素分别是向量的方差。
PCA原理
主成分分析法(Principal Component Analysis)是一种基于变量协方差矩阵对数据进行压缩降维、去噪的有效方法,它借助正交变换将一组可能存在相关性的变量转换为一组线性不相关的变量,转换后的这组变量叫主成分(PC),主成分是旧特征的线性组合。
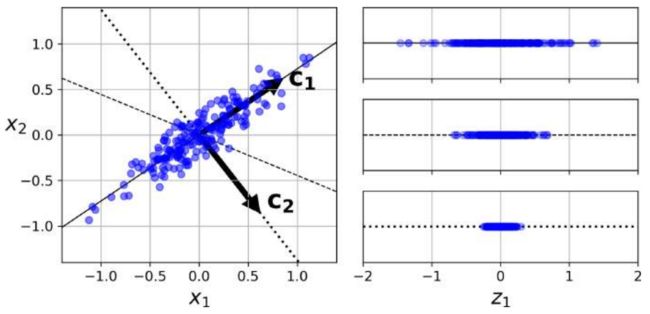
PCA的本质就是找一些投影方向,使得数据在这些投影方向上的方差最大,而且这些投影方向是相互正交的。这其实就是找新的正交基的过程,计算原始数据在这些正交基上投影的方差,方差越大,就说明在对应正交基上包含了更多的信息量。如下图,第一个 PC 为 c1 所在的轴,第二个PC 为 c2 所在的轴,第三个 PC 为与平面正交的轴。我们仅保留一定数量的主成分来解释原始数据集的方差,而忽略其余部分。
PCA常用于高维数据的降维、数据噪音消除、图像压缩、特征脸等等。
PCA公式推导
1.假设为 维随机变量,其均值为,协方差矩阵为。
考虑由维随机变量到维随机变量的线性变换
其中。
如果该线性变换满足以下条件,则称之为总体主成分:
(1);
(2);
(3)变量是的所有线性变换中方差最大的;是与不相关的的所有线性变换中方差最大的;一般地,是与都不相关的的所有线性变换中方差最大的;这时分别称为的第一主成分、第二主成分、…、第主成分。
假设是维随机变量,其协方差矩阵是,的特征值分别是,特征值对应的单位特征向量分别是,则的第2主成分可以写作
并且,的第主成分的方差是协方差矩阵的第个特征值,即
3.主成分有以下性质:
主成分的协方差矩阵是对角矩阵
主成分的方差之和等于随机变量的方差之和
其中是的方差,即协方差矩阵的对角线元素。
主成分与变量的相关系数称为因子负荷量(factor loading),它表示第个主成分与变量的相关关系,即对的贡献程度。
4.样本主成分分析就是基于样本协方差矩阵的主成分分析。
给定样本矩阵
其中是的第个独立观测样本,。
的样本协方差矩阵
给定样本数据矩阵,考虑向量到的线性变换
这里
如果该线性变换满足以下条件,则称之为样本主成分。样本第一主成分是在条件下,使得的样本方差最大的的线性变换;
样本第二主成分是在和与的样本协方差条件下,使得的样本方差最大的的线性变换;
一般地,样本第主成分是在和与的样本协方差条件下,使得的样本方差最大的的线性变换。
5.主成分分析方法主要有两种,可以通过相关矩阵的特征值分解或样本矩阵的奇异值分解进行。
(1)相关矩阵的特征值分解算法。针对样本矩阵,求样本相关矩阵
再求样本相关矩阵的个特征值和对应的单位特征向量,构造正交矩阵
的每一列对应一个主成分,得到样本主成分矩阵
(2)矩阵的奇异值分解算法。针对样本矩阵
对矩阵进行截断奇异值分解,保留个奇异值、奇异向量,得到
的每一列对应一个主成分,得到样本主成分矩阵
PCA算法流程
输入:n维样本集,要降维到的维数n'.
输出:降维后的样本集
1、 对所有的样本进行中心化:
2、 计算样本的协方差矩阵
3、 对矩阵进行特征值分解
4、 取出最大的n'个特征值对应的特征向量, 将所有的特征向量标准化后,组成特征向量矩阵W。
5、 对样本集中的每一个样本,转化为新的样本
6、 得到输出样本集
有时候,我们不指定降维后的n'的值,而是换种方式,指定一个降维到的主成分比重阈值t。这个阈值t在(0,1]之间。假如我们的n个特征值为,则n'可以通过下式得到:
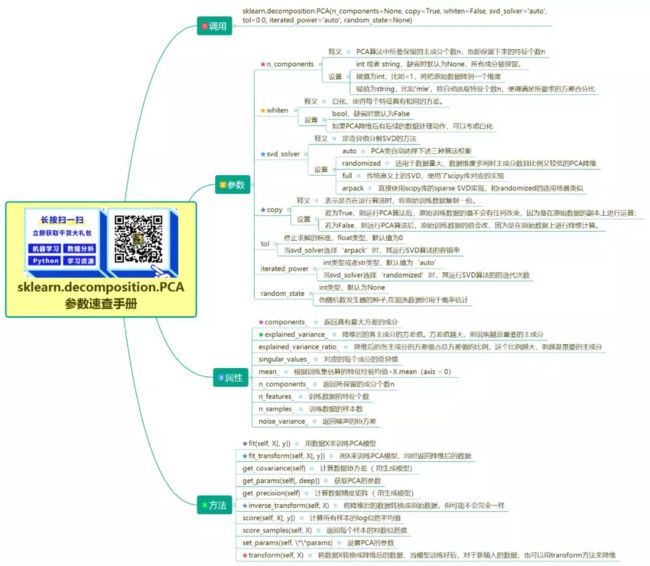
scikit-learn中PCA的使用方法
调用
sklearn.decomposition.PCA(n_components=None, copy=True, whiten=False, svd_solver='auto', tol=0.0, iterated_power='auto', random_state=None)
sklearn.decomposition.PCA 参数速查手册文字版
PCA 的使用简单的离谱,一般指定n_components即可,如果设置为整数,说明要保留的主成分数量。如果将其设置为小数,则说明降维后的数据能保留的信息。
上面提到主成分分析方法主要有两种,可以通过相关矩阵的特征值分解或样本矩阵的奇异值分解进行。scikit-learn库的PCA使用的就是奇异值分解方法,通过svd_solver参数指定:
randomized:适用于数据量大,数据维度多同时主成分数目比例又较低的 PCA 降维
full:传统意义上的 SVD,使用了 scipy 库对应的实现
arpack:直接使用 scipy 库的 sparse SVD 实现,和 randomized 的适用场景类似
import numpy as np
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
from sklearn.decomposition import PCA
from sklearn.datasets.samples_generator import make_blobs
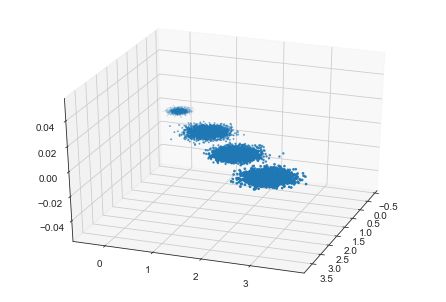
X, y = make_blobs(n_samples=10000, n_features=3, centers=[[3, 3, 3], [0, 0, 0], [1, 1, 1], [2, 2, 2]],
cluster_std=[0.2, 0.1, 0.2, 0.2], random_state=9)
fig = plt.figure()
plt.scatter(X_new[:, 0], X_new[:, 1], marker='o')
plt.show()
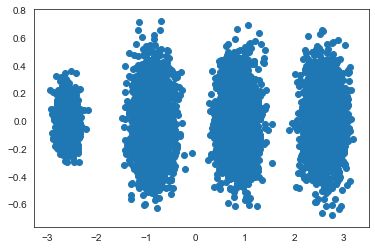
# 降维到二维
pca = PCA(n_components=2)
pca.fit(X)
# 输出特征值
print(pca.explained_variance_)
输出特征向量
print(pca.components_)
# 降维后的数据
X_new = pca.transform(X)
print(X_new)
fig = plt.figure()
plt.scatter(X_new[:, 0], X_new[:, 1], marker='o')
plt.show()
这样我们就实现了三维到二维的转换,可以把结果可视化:
PCA算法优缺点
PCA算法优点
1,仅仅需要以方差衡量信息量,不受数据集以外的因素影响
2,各主成分之间正交,可消除原始数据成分间的相互影响的因素
3,计算方法简单,主要运算时特征值分解,易于实现
4,它是无监督学习,完全无参数限制的。
PCA算法缺点
1,主成分各个特征维度的含义具有一定的模糊性,不如原始样本特征的解释性强
2,方差小的非主成分也可能含有对样本差异的重要信息,因降维丢弃可能对后续数据处理有影响。
参考
https://github.com/fengdu78/lihang-code
https://finthon.com/python-pca/
https://www.cnblogs.com/pinard/p/6239403.html