circRNA数据库
circRNA是什么
circRNAs(Circular RNAs,环形RNA分子)是一类不具有5’ 末端帽子和3’ 末端poly(A)尾巴,并以共价键形成环形结构的非编码RNA分子。circRNA是由非经典剪接方式进行反向剪接而形成。
环状的RNA主要特征
- 不具有5’末端耗子和3’末端ployA尾巴,并以共价键形成环状结构;
- 大部分属于ncRNA;
- 主要由外显子衍生而来,且由一个或多个外显子构成;
- 同一基因位点或许可通过选择性环化衬衫多种circRNAs;
- 环化过程与pre-mRNA剪接彼此竞争;
- 其形成过程收到剪接因子的调控;
- 比线性RNA稳定,不易被核酸外切酶RNase R降解;
- 多数具有序列保守性;
- 表达水平差异大,具有时空特异性及组织特异性;
- 大多数定位于细胞质中;
- 可作为竞争性内源RNA调控基因的表达;
- 在转录或转录后水平发挥调控作用。
环状RNA的作用机制
- circRNA充当miRNA分子“分子海绵”来调节基因表达。比如,天然环状RNA小脑变性相关蛋白Ⅰ反义转录物(CDR1as)可作为miR-7a/b的海绵发挥作用。当CDR1as过表达时,能大量结合miR-7而导致miR-7靶基因的表达水平上升;而当抑制CDR1as表达时,miR-7靶基因的表达水平会降低。
- circRNA调控基因的转录。比如,细胞核中的一些环状RNA可以与U1 snRNP进行RNA-RNA相互作用,形成复合物并在母系基因启动子区与RNA聚合酶Ⅱ转录复合物结合,顺式调控其母系基因的表达,这也为相应疾病的治疗提供了新思路。
- circRNA与蛋白质相互作用。circRNA也有蛋白质吸附功能,比如,CDR1as和SRY circRNAs 可与miRNA效益因子AGO相结合。
- circRNA参与蛋白质翻译。大多数circRNA由外显子构成且主要存在于细胞质中,暗示其可能可以被装载到核糖体中翻译成多肽。研究发现,丁型肝炎病毒(HDV)的核心circRNA分子编码相关蛋白HDV抗原,其与疾病发展密切相关。
数据库
下面简要介绍一些常用的数据库~欢迎大家跟进提供更新信息!
环状RNA(circRNA)常用在线数据库汇总
一 http://www.circbase.org
简介:circBase探索公共circRNA数据集和下载,发现在自己的(RIBOMINUS)RNA测序数据circRNAs需要自定义Python脚本。
二 http://starbase.sysu.edu.cn/mirCircRNA.php
简介:circRNABase是专为从数以千计circRNAs和108 CLIP-SEQ(HITS-CLIP,PAR-CLIP,iCLIP,CLASH)数据集进行解码的miRNA-circRNA相互作用网络。
三 http://regrna.mbc.nctu.edu.tw/php/prediction.php
四 http://gyanxet-beta.com/circdb/
简介:Circ2Traits收集可能与疾病和性状相关环状RNA的综合数据库,该数据库通过预测miRNAs和人类的蛋白质编码基因、长链非编码基因及环状RNA间的相互作用关系,构建了相互作用网络, 并对miRNAs-circRNA相互作用组中的蛋白编码基因进行了GO富集分析;此外,将与疾病相关的SNPs位点定位到circRNA基因座上,并鉴定了环状RNAs上的Ago相互作用位点。
五 http://circinteractome.nia.nih.gov/
简介:该数据库预测了已知的109个RNA结合蛋白数据集与circbase中的circRNA的结合位点,并利用Targetscan软件预测了miRNAs与circRNA的潜在结合位点。
六 http://circnet.mbc.nctu.edu.tw/
简介:利用464个RNA-seq测序数据,进行新circRNA预测及基因组注释,并计算已知的及新预测的circRNA表达情况,构建circRNA-miRNA-genet调控网络,以上信息均可从该数据库获得。
七 deepBase v2.0 http://deepbase.sysu.edu.cn/
简介:该数据平台收集了大约15万多的circRNA基因(人、鼠、果蝇、线虫等),并构建了最全面的circRNA的表达图谱。
八 http://reprod.njmu.edu.cn/circrnadb
首个汇总可编码蛋白的环状RNA数据库,共收集了32914条人类外显子环状RNA记录,每条记录都包括基因组位置信息,RNA编辑情况,所对应的基因组序列,IRES序列元件,预测的ORF以及相关的参考文献。作者发现了有16328条环状RNA包含了编码超过100个氨基酸的ORF,其中7170种环状RNA存在IRES序列元件,基本符合翻译蛋白的特征 ⌈详细介绍⌋
九 http://www.picb.ac.cn/rnomics/circpedia/
中科院上海生科院计算生物学研究所杨力研究组利用建立的高效环形RNA分析流程CIRCexplorer2,对多种(目前主要是人和小鼠)组织和细胞系样品中的环形RNA进行计算生物学预测分析,并进一步构建了CIRCpedia数据库,对环形RNA分子中的可变反向剪接(可变环化)和可变剪接进行归类,以便相关研究人员使用。⌈详细介绍⌋
十 http://gb.whu.edu.cn/CSCD (cancer-specific circRNA database)
CSCD数据库从ENCODE中收集了19种肿瘤类型的87种细胞系及141种正常细胞的circRNA数据,构建了该数据库。总共汇总得到了272152种肿瘤特异性的circRNAs,950962种正常细胞特异的circRNAs,还有170909种为肿瘤和正常样本共有的。
CSCD数据库可以预测circRNA亚细胞定位、MRE、RBP、ORF、相关基因可变剪切分析,CSCD还提供了到UCSC浏览器及circBase的链接,并且给出了每个样本和每个算法中预测得到的junction reads数以及相应的均一化表达量SRPTM,可以方便地进行不同样本的环状RNA丰度比较。共收集了1394023种circRNA分子,其中外显子来源759039种,内含子来源436668种,基因间序列来源122806种。来自mRNA的1153542种,来lncRNA的42165种。预测到了MRE 76439955个,RNA结合蛋白结合位点103927037个,ORF 3462097个。
网址:http://gb.whu.edu.cn/CSCD
十一 http://app.cgu.edu.tw/circlnc/
整合lncRNA和circRNA的功能网络的在线分析数据库
circlncRNAnet 在线数据库由我国台湾科学家创建的关于lncRNA和circRNA功能研究的数据库,本人试用后发现该数据库非常实用,对科研课题的数据挖掘可以起到事半功倍的作用,是近年来ncRNA领域不可多得的在线友好数据分析工具。
总结起来该数据库主要有如下三大优点:
1、 用户可提交分析自己的lncRNA和circRNA芯片或测序的表达数据;
2、 整合了TCGA数据库20多个肿瘤类型数据,无需编程实现在线可视化分析;
3、 分析模块非常丰富,包括lncRNA组织表达热图、箱线图、共表达散点图、circos图,基因功能富集分析GO和KEGG图,还有RBP-RNA结合蛋白网络图,miRNA网络图等;
十二、http://cgga.org.cn:9091/circRNADisease/
Cell Death and Disease以Letters形式发表了北京天坛医院神经外科江涛主任为通讯作者的文章,发布了一个收集人类疾病相关circRNA的数据库:circRNA disease [1]
circRNA Disease数据库的检索方式可以通过circRNA ID/name、circRNA host gene和疾病名称。 具体的circRNA记录包括circRNA基础信息项,相关疾病信息,PubMed链接。circRNA的ID号也做了circBase的链接,可以直接前往circBase数据库。
十三、CircAtlas:http://circatlas.biols.ac.cn/
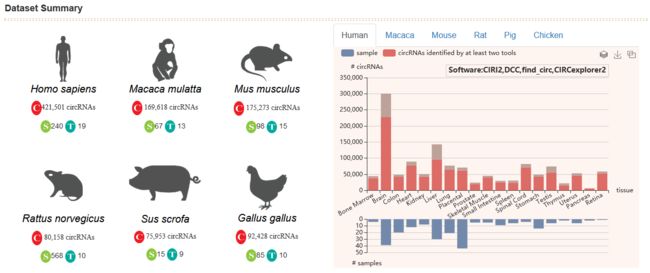
环状RNA(circRNA)相关表达谱数据已成为转录组学的重要组成部分。近日赵方庆教授团队在genome biology杂志发表了circAtlas数据库的文章。circAtlas数据库基于1070个RNA-seq样本数据(从六个脊椎动物物种的19个正常组织中收集)整合出环状RNA相关信息。该数据库包含1,007,087个高度可靠的circRNA,其中81.3%以上已被组装成全长序列。circAtlas网站全面开放提供了这些circRNA的表达模式,保守性和功能注释等信息,是circRNA研究最重要的在线资源之一。
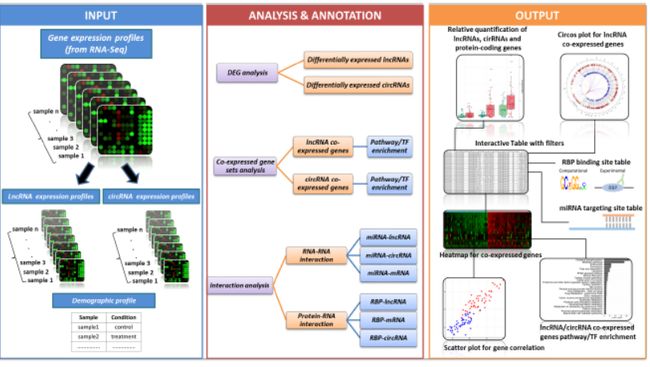
circAtlas包括从六种脊椎动物(人类,猕猴,小鼠,大鼠,猪和鸡)收集的19种正常组织,并使用了最新的生物信息学分析方法进行circRNA分析。circAtlas的内容和构建如上图所示。每种物种中的circRNA使用四种可靠的检测算法进行了识别,包括CIRI2,find_circ,CIRCexplorer2和DCC。使用CIRI-full / CIRI-vis pipline重建鉴定出的circRNA的全长序列。随后,在全长circRNA中搜索内部核糖体进入位点(IRESs)和ORF,以预测其编码潜力。使用多重保守评分(MCS)方案对circRNA的保守性进行了表征,该方案可估计circRNA在物种,组织和个体三个水平上的保守性。接下来,将有关共表达网络,circRNA-miRNA和RBP结合位点的信息结合起来,对circRNA进行了全面注释。使用GO和KEGG数据库来预测这些circRNA的潜在功能。同时,将circad,circR2Disease和circRNADisease数据库数据整合到circAtlas中,以评估circRNA与各种疾病的相关性。
用户在circAtlas网站界面可输入circRNA名称或序列进行检索。如果circAtlas中已经包含查询circRNA,则用户可以浏览并立即下载其序列,表达谱和注释等相关信息。否则,服务器将查询跨物种的新型circRNA的直系同源序列,执行功能注释,并基于综合注释对候选对象进行优先级排序。circAtlas的全部内容可免费获得,并可从网站下载。
相比于现有的circRNA数据库,circAtlas收集的脊椎动物circRNA信息最全面。不仅物种数量多,且在多个物种的正常样本和个体中检测到circRNA数量更多。在鉴定circRNA过程中,综合运用了CIRI2,DCC,find_circ和CIRCexplorer2等主流算法,显著提高了鉴定circRNA的可靠性。另一方面circAtlas中的样品多样性为不同维度分析circRNA提供了可能性,比如物种或组织间circRNA表达模式研究,circRNA进化保守性分析。circAtlas利用MSC方法全面评估了circRNA的保守性,发现绝大多数circRNA(平均61.7%)只在一种物种检测到,所有物种均保守的circRNA有797个。此外,circAtlas还在整合circRNA调控网络的基础上进行了深入的功能性注释,包括共表达、microRNA和RNA结合蛋白等信息,为circRNA的深入研究提供了重要的数据资源。
参考文献:
Wu, W., Ji, P. & Zhao, F. CircAtlas: an integrated resource of one million highly accurate circular RNAs from 1070 vertebrate transcriptomes. Genome Biol 21, 101 (2020). https://doi.org/10.1186/s13059-020-02018-y
1. Glazar, P., P. Papavasileiou, andN. Rajewsky, circBase: a database forcircular RNAs. RNA, 2014. 20(11):p. 1666-70.
2. Li, J.H., etal., starBase v2.0: decoding miRNA-ceRNA,miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seqdata. Nucleic Acids Res, 2014. 42(Databaseissue): p. D92-7.
3. Ghosal, S., etal., Circ2Traits: a comprehensivedatabase for circular RNA potentially associated with disease and traits.Front Genet, 2013. 4: p. 283.
4. Liu, Y.C., etal., CircNet: a database of circular RNAsderived from tranome sequencing data. Nucleic Acids Res, 2016. 44(D1): p. D209-15.
5. Zheng, L.L., etal., deepBase v2.0: identification,expression, evolution and function of small RNAs, LncRNAs and circular RNAsfrom deep-sequencing data. Nucleic Acids Res, 2016. 44(D1): p. D196-202.
6. Dudekula, D.B.,et al., CircInteractome: A web tool forexploring circular RNAs and their interacting proteins and microRNAs. RNABiol, 2016. 13(1): p. 34-42.
参考circRNA数据库 | 环状RNA社区——CircRNA Community,略有更新。由于网络限制某些数据库可能不能访问。