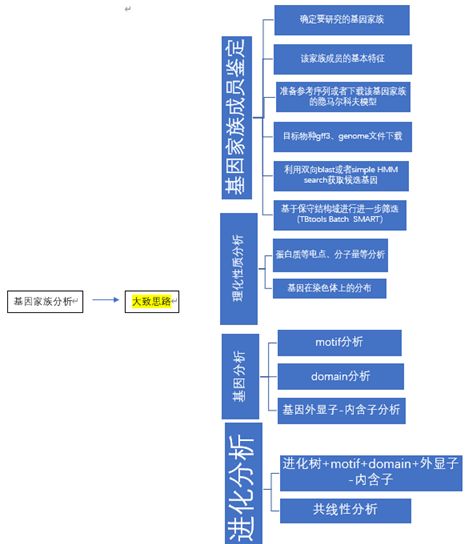
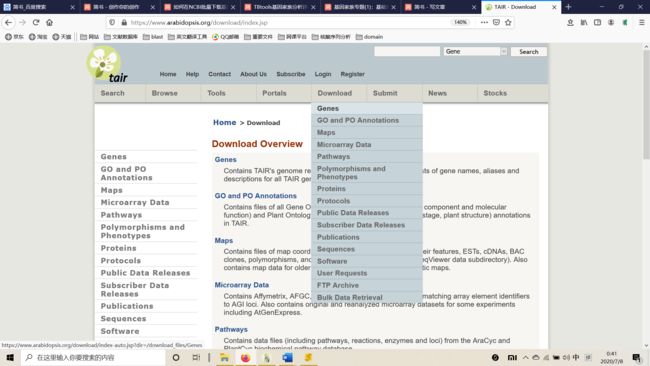
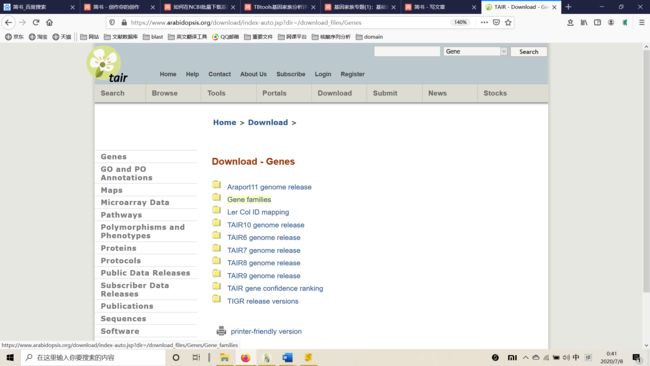
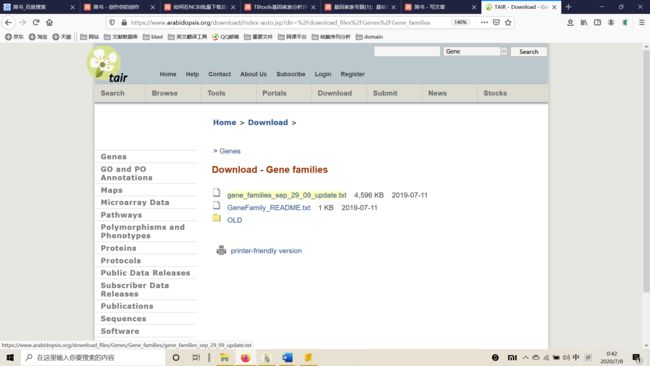
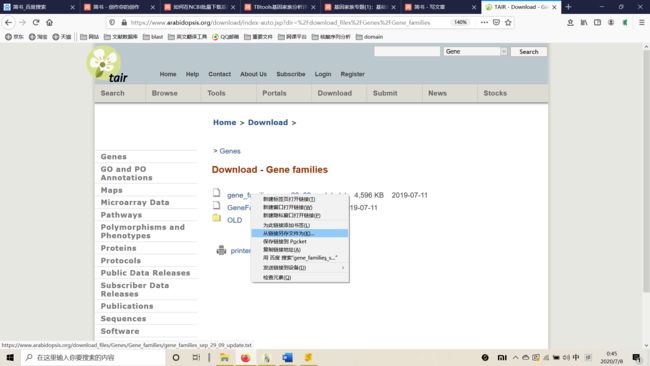
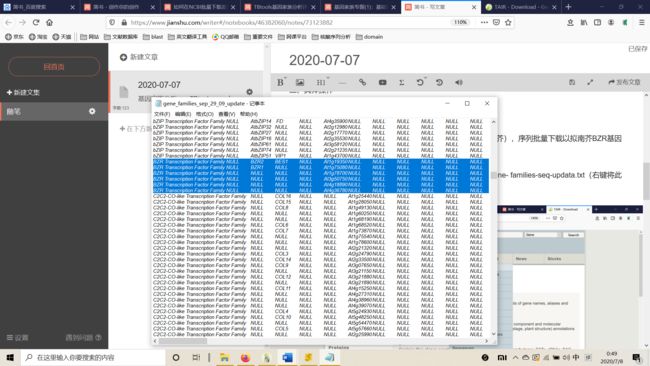
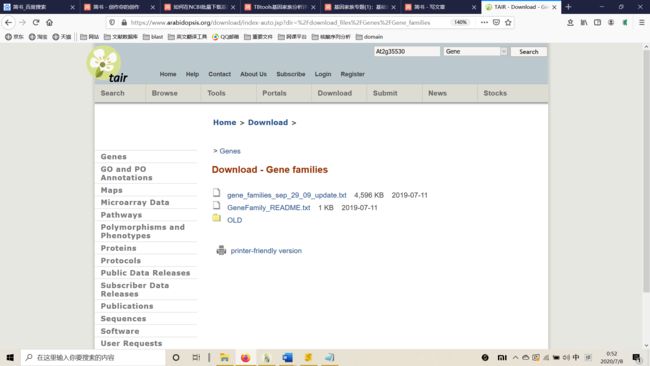
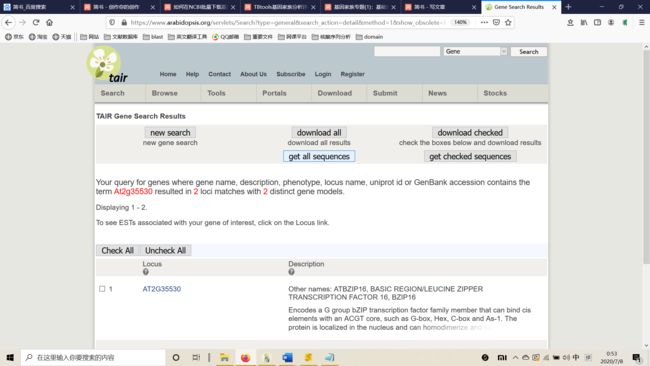
- ZYNQ MPSOC PL端DDR4读写--介绍(1)
LEEE@FPGA
FPGA高速接口开发fpga开发DDR4
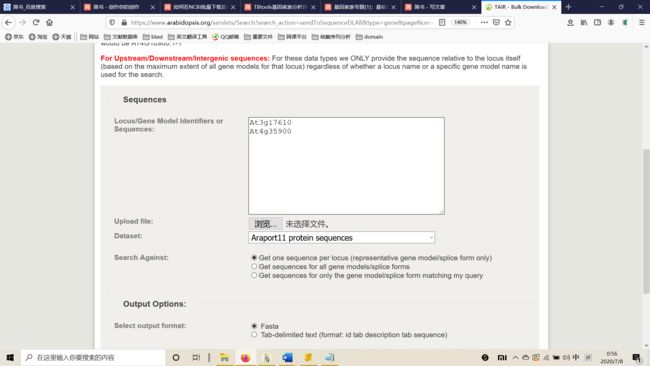
1DDR4介绍DDR4SDRAM(Double-Data-RateFourthGenerationSynchronousDynamicRandomAccessMemory,简称为DDR4SDRAM),是一种高速动态随机存取存储器,它属于SDRAM家族的存储器产品,提供了相较于DDR3SDRAM更高的运行性能与更低的电压,并被广泛的应用于计算机的运行缓存。DDR4主要特点1)更高频率与带宽起步频率为
- Codeforces Round 1034 (Div. 3) G解题思路
拉长时间线
数据结构与算法算法数据结构c++
链接Problem-G-Codeforces题目大意给定n,m,q分别为数组大小,数组的每个数非负且小于m,要进行q次操作操作分为两种:1.令a[i]=x(永久性)2.输入一个k,对于每个a[i]都可进行任意次操作a[i]=(a[i]+k)%m,对数组进行操作,判断能否增厚变成一个非严格递增数组题目思路对样例进行分析可以发现对于每个a[i]可以分为g=gcd(m,k)类,可以为每一类标号,号码为a
- 合成生物学奇点:AI驱动CRISPR超进化工厂2025投产纪实
HeartException
人工智能
前言前些天发现了一个巨牛的人工智能免费学习网站,通俗易懂,风趣幽默,忍不住分享一下给大家。点击跳转到网站《合成生物学奇点:AI驱动CRISPR超进化工厂2025投产纪实》副标题:全球首座AI-BioFab落地深圳,蛋白质设计周期从3年压缩至11天,生物制造成本暴跌90%一、生物制造范式的历史性颠覆▶︎传统生物工程的三大世纪困局graphTDA[缓慢的试错循环]-->B[单基因改造耗时≥6个月]C[
- 在股市中寻找志同道合的朋友
niuniu15816888
财经社交大数据
作为一名普通的股票投资者,我在这条路上已经走了五年。从最初的懵懂无知到现在能独立分析行情,我深深体会到:**投资不是一个人的战斗,找到志同道合的伙伴能让这条路走得更远**。今天,我想和大家分享一些我在投资社交中的真实感悟。一、为什么我们需要投资社交?记得刚开始炒股时,我总是一个人盯着K线图发呆。直到有一天,我在一个股票论坛上认识了几位同样喜欢研究新能源板块的朋友,我们组建了一个小群组。每周五晚上,
- [AI笔记]-LLM中的3种架构:Encoder-Only、Decoder-Only、Encoder-Decoder
Micheal超
AI笔记人工智能笔记架构
一、概述架构描述特点案例Encoder-Only仅包含编码器部分这类模型主要专注输入数据中提取特征或上下文信息,通常不需要生成新内容、只需要理解输入的任务,如:分类(文本分类、情感分析等)、信息抽取、序列标注等。在这种架构中,所有的注意力机制和网络层都集中在编码输入数据上,其输出通常是关于输入的复杂语义表示。谷歌的BERT、智谱AI发布的第四代基座大语言模型GLM4Decoder-Only也被称为
- H264码流介绍 及 FFmpeg解封装得到H264码流方法
冰冰的coco
音视频ffmpeg
参考文章音视频H264编解码协议-知乎视频H264编码详解(上)-知乎H.264媒体流AnnexB和AVCC格式分析-CSDNH264之NALU解析-知乎H264帧,SPS,PPS概念-知乎H.264流媒体协议格式中的AnnexB格式和AVCC格式深度解析-CSDNH264简介H.264从1999年开始,到2003年形成草案,最后在2007年定稿有待核实。在ITU的标准⾥称为H.264,在MPEG
- MyBatis SQL 执行过程原理分析(附源码) 代理层:Mapper 接口动态代理 路由层:MapperMethod 分发 核心引擎:SqlSession 执行
夜雨hiyeyu.com
mybatissql数据库数据库架构javaspringbootdb
MyBatisSQL执行过程原理分析(附源码)1.代理层:Mapper接口动态代理2.路由层:MapperMethod分发3.核心引擎:SqlSession执行4.执行器:Executor调度5.处理器层:StatementHandler执行6.结果映射:ResultSetHandler转换核心执行流程图关键设计亮点性能优化建议MyBatis的SQL执行过程可以分为6个核心阶段,我们将通过源码逐层
- 成像光谱遥感技术中的AI革命:ChatGPT在遥感领域中的应用
科研的力量
人工智能ChatGPTchatgpt人工智能
课程将最新的人工智能技术与实际的遥感应用相结合,提供不仅是理论上的,而且是适用和可靠的工具和方法。无论你是经验丰富的研究人员还是好奇的从业者,本课程都将为分析和解释遥感数据开辟新的、有效的方法,使你的工作更具影响力和前沿性。遥感技术主要通过卫星和飞机从远处观察和测量我们的环境,是理解和监测地球物理、化学和生物系统的基石。ChatGPT是由OpenAI开发的最先进的语言模型,在理解和生成人类语言方面
- 【Django开发】前后端分离django美多商城项目第3篇:用户注册业务实现,用户注册前端逻辑【附代码文档】
教程总体简介:欢迎来到美多商城!项目需求分析1.项目主要页面介绍2.归纳项目主要模块3.知识要点项目架构设计1.项目开发模式2.项目运行机制项目介绍创建工程1.准备项目代码仓库3.创建美多商城工程配置开发环境1.新建配置文件2.指定开发环境配置文件配置Jinja2模板引擎1.安装Jinja2扩展包配置MySQL数据库3.安装PyMySQL扩展包配置Redis数据库1.安装django-redis扩
- Python 开发规范:pdb & cProfile:调试 & 性能分析
写文章的大米
Python核心技术python
↑↑↑欢迎点赞、关注、收藏!!!,10年IT行业老鸟,持续分享更多IT干货文章目录pdb&cProfile:调试&性能分析核心内容1、调试和性能分析的必要性2、pdb调试工具3、cProfile性能分析工具pdb&cProfile:调试&性能分析核心内容1、调试和性能分析的必要性在实际生产环境中,代码调试(找问题根因、修复bug)和性能分析(优化效率、减少latency)是开发关键环节。尤其,面对
- 论文学习_SoK: An Essential Guide For Using Malware Sandboxes In Security Applications: Challenges, Pitfa
kitsch0x97
学习
0.文章概述恶意软件沙箱尽管在安全应用程序中带来许多优势,但其复杂的选择、配置和使用过程常让新用户不知所措,甚至可能导致错误的部署,进而对安全分析结果产生负面影响。目前,缺乏系统化的指导来帮助用户正确选择和应用沙箱工具,这种知识空白阻碍了沙箱在不同研究领域中的有效应用。为了填补这一知识空白,研究团队系统分析了84篇关于x86/64恶意软件沙箱的学术论文,并提出了一种新颖的框架,以简化沙箱组件和操作
- 数据结构:递归:汉诺塔问题(Tower of Hanoi)
95号闪电麦坤
数据结构数据结构
目录问题描述第一性原理分析代码实现第一步:明确函数要干什么第二步:写好递归的“结束条件”第三步:写递归步骤递归调用树问题描述有三个柱子(A,B,C),上面有n个大小不等的圆盘,最开始所有圆盘按从大到小顺序堆在柱子A上。目标:将所有圆盘移动到柱子C,移动时要满足:一次只能移动一个盘子;任何时刻小盘子不能压在大盘子上。❓核心问题:如何将n个盘子从A移动到C,同时只用B做辅助,且不违反约束?第一性原理分
- 【分析学】从有限开覆盖定理出发 -- 实数系完备性
BlackPercy
分析学数学高等数学
目录有限开覆盖定理实数系完备性定理确界定理单调有界定理闭区间套定理聚点定理柯西收敛定理有限开覆盖定理开覆盖定义:设{Ui}i∈I\{U_i\}_{i\inI}{Ui}i∈I是一个开覆盖,即[a,b]⊆⋃i∈IUi[a,b]\subseteq\bigcup_{i\inI}U_i[a,b]⊆⋃i∈IUi,称⋃i∈IUi\bigcup_{i\inI}U_i⋃i∈IUi为闭区间[a,b][a,b][a,b
- 【iSAQB软件架构】原型和技术概念验证
小马哥编程
ui系统架构架构开发语言产品经理
在软件开发项目的过程中可能会出现许多不同类型的问题。要么利益相关者难以明确(尤其是完整地)阐述需求,要么系统用户和开发人员之间的合作无法正常进行。通常,合作在分析和设计阶段结束,因为开发人员随后会退出,只有在软件完成时才展示他们的工作成果。如果团队要相互学习,团队之间的协调非常重要。各种解决方案必须与客户进行测试和讨论,并且某些需求无法仅根据其理论描述得到保证(例如,实时需求)。因此,在定义阶段完
- 【ISAQB大纲解读】软件密集型系统的三大分类
小马哥编程
系统架构架构
软件密集型系统的类型划分为信息系统、嵌入式系统和移动系统,主要基于其应用场景、功能定位、硬件依赖程度及软件设计逻辑的显著差异。以下从三类系统的核心特征、典型场景及分类逻辑展开分析:一、软件密集型系统的定义与分类逻辑软件密集型系统指软件在系统功能实现中起决定性作用的系统,其分类本质上反映了不同场景下软件与硬件、业务需求的耦合方式。三类系统的划分依据包括:应用领域:商业办公、设备控制、移动交互等不同场
- 嵌入模型 vs 大语言模型:语义理解能力的本质区别与应用场景
chenkangck50
AI大模型语言模型人工智能机器学习
嵌入模型vs大语言模型:语义理解能力的本质区别与应用场景(实战视角)一句话总结嵌入模型的“理解”是向量表示和相似性匹配,适合做召回;大语言模型的“理解”是上下文+逻辑+世界知识综合判断,适合做分析与生成。重点是可以结合prompt和本身具有的知识两类模型的本质区别能力项嵌入模型(如BGE、SBERT)大语言模型(如GPT、GLM、DeepSeek)输出形式向量(如768维)自然语言文本(如答案、解
- 现代Unity架构指南:以ECS为核心的应用程序中构建OOP抽象层
NocturnalSky
Unityunity架构游戏引擎
文章目录现代Unity架构指南:以ECS为核心的应用程序中构建OOP抽象层为什么需要混合架构抽象层设计原则性能关键优化技术典型应用场景实现混合架构性能分析(Unity2023.2实测)关键陷阱与解决方案架构演进策略Unity2023新特性整合现代Unity架构指南:以ECS为核心的应用程序中构建OOP抽象层为什么需要混合架构ECS的核心优势:数据局部性、缓存友好性、高效的并行处理能力,特别适合高性
- ECS抽象层与模块封装:构建可维护的高性能架构
NocturnalSky
Unityunity
文章目录ECS抽象层与模块封装:构建可维护的高性能架构模块化设计的必要性分层抽象架构接口设计准则模块注册机制依赖管理策略性能优化技巧实战应用示例模块热重载实现性能影响分析设计权衡建议ECS抽象层与模块封装:构建可维护的高性能架构模块化设计的必要性问题诊断:大型ECS项目易陷入"系统膨胀"——数百个无关联系统导致代码混乱封装目标:将相关系统/组件组合为功能模块(如AI、物理、库存)核心原则:模块间通
- workflow的可编排协作模式
思绪漂移
人工智能agentworkflow模式
Workflow的可编排协作模式在AI工作流设计中,协作模式的选择直接影响系统效率和结果质量。本文将系统分析6种主流协作模式,探讨选择方案与适合场景。一、传统单一模式特点:使用标准框架进行线性编排所有任务按固定顺序执行适合简单、确定性强的流程适用场景:规则明确、步骤固定的业务流程自动化二、路由模式核心机制:LLM先识别用户意图根据意图路由到对应处理模块优势:意图识别准确率高资源分配精准典型应用:客
- 小红书笔记详情API接口实战:内容数据获取与分析的利器
在数字化时代,数据成为了一种无形的财富,其背后的价值越来越被重视。小红书,作为国内知名的社区分享平台,聚集了大量优质的内容和用户数据。对于企业、个人或者研究机构而言,获取并分析小红书上的笔记详情数据,不仅可以了解用户的行为和兴趣,还可以为决策提供有力的数据支持。本文将介绍如何通过小红书的API接口获取笔记详情数据,并对其进行深入分析,同时附上实战代码,帮助读者更好地理解和应用。二、小红书API接口
- 小红书笔记详情API接口概述及JSON数据返回参考
Json_18179014480
APIjson大数据数据库大数据json
前言一、接口概述小红书笔记详情API接口是小红书开放平台提供的一项服务,允许开发者通过编程方式获取小红书上特定笔记的详细信息。该接口的核心功能包括:获取笔记内容:标题、正文、图片、视频等多媒体信息。用户互动数据:点赞数、评论数、收藏数、分享数等。作者信息:作者昵称、头像、粉丝数等。发布信息:发布时间、标签列表等。通过该接口,开发者可以构建内容分析工具、笔记推荐系统、数据爬虫等应用,帮助企业或个人进
- 网络延迟诊断与优化:从路由到应用层的全链路分析
Clownseven
网络智能路由器
更多云服务器知识,尽在hostol.com你有没有在日常工作中体验过网络延迟带来的焦虑?浏览器页面加载缓慢,API请求几秒钟不返回,甚至服务器上的某个微服务迟迟无法响应。每次点击刷新页面,你的心里是否已经准备好迎接各种负面反馈?网络延迟,简而言之,就是数据从源头传输到目的地所花的时间。你可能认为延迟只是一个小问题,可当延迟问题累积,可能就会变成让整个系统瘫痪的大隐患。我们每一个运维人员都清楚,任何
- 1688按关键词获取商品列表API接口详解
蓝倾976
python开发语言电商开放平台开放API1688开放平台
一、接口功能概述1688商品列表API是阿里巴巴开放平台提供的核心接口之一,主要用于通过关键词、价格区间、销量范围、类目ID等条件筛选商品,并返回商品标题、价格、销量、图片等基本信息。该接口广泛应用于电商数据分析、竞品调研、商品监控、价格比对等场景,助力开发者高效获取1688平台商品数据。二、接口调用流程1.注册与认证注册账号:在1688开放平台/万邦开放平台注册开发者账号,完成企业或个人资质审核
- 结构性变革与新兴机遇
倒霉男孩
经济学
近年来,全球就业市场正经历深刻的结构性变革。受技术进步、产业升级、人口结构变化及全球经济格局调整的影响,传统就业模式被重塑,新的职业机会不断涌现。本文将分析当前就业市场的主要趋势,并探讨其对劳动者、企业和政策制定者的启示。###**一、技术驱动下的就业结构变化**1.**人工智能与自动化替代部分传统岗位**-麦肯锡全球研究院预测,到2030年,全球约14%的劳动者(3.75亿人)可能因自动化技术而
- .NET 8/9异步编程黄金法则:零缺陷与性能飞跃实战
——从“未等待任务”到线程池优化的深度避坑指南异步编程的“暗礁”与.NET8/9的破局之道在.NET应用中,异步编程是提升响应性和资源利用率的核心技术,但不当使用可能导致线程死锁、内存泄漏、未捕获异常等致命问题。.NET8/9通过托管线程池优化、服务器GC改进和编译器增强,为开发者提供了更安全高效的异步编程环境。本文将通过10个真实场景、20段代码示例和深度性能分析,手把手教你规避异步开发的常见陷
- Java线程池源码分析,深度解读
努力的橙子go
java开发语言
前言本文将深入分析Java线程池的源码,包括线程池的创建、任务提交、工作线程的执行和线程池的关闭等过程。通过对线程池源码的解析,我们能够更好地理解线程池的原理和机制,为我们在实际开发中合理使用线程池提供指导。文章内容较长,建议找个安静的环境慢慢细读。线程池简介概念在传统的多线程编程中,每次需要执行任务时都会创建一个新的线程,任务执行完毕后再销毁该线程。这种方式存在一些问题,例如频繁创建和销毁线程会
- Python爬取与可视化-豆瓣电影数据
木子空间Pro
项目集锦#课程设计python信息可视化开发语言
引言在数据科学的学习过程中,数据获取与数据可视化是两项重要的技能。本文将展示如何通过Python爬取豆瓣电影Top250的电影数据,并将这些数据存储到数据库中,随后进行数据分析和可视化展示。这个项目涵盖了从数据抓取、存储到数据可视化的整个过程,帮助大家理解数据科学项目的全流程。环境配置与准备工作在开始之前,我们需要确保安装了一些必要的库:urllib:用于发送HTTP请求和获取网页数据Beauti
- Java中ThreadPoolExecutor源码深度解析
振华少爷
java开发语言前端
Java中ThreadPoolExecutor源码深度解析目录引言ThreadPoolExecutor的数据结构核心方法分析构造方法execute方法shutdown方法shutdownNow方法性能分析使用注意事项总结引言ThreadPoolExecutor是Java并发包中的一个线程池实现类,它提供了灵活的线程池管理功能,可以根据需要创建、管理和销毁线程。ThreadPoolExecutor通
- 基于Python的携程景点评价爬虫与情感评分分析
程序员威哥
python爬虫开发语言
一、项目背景携程(Ctrip)是中国最流行的旅游预订平台之一,其景点用户评论包含了大量真实的游客反馈。通过分析评论的情感倾向,可以:为用户提供更可靠的景点推荐辅助景区运营方了解用户口碑构建情感评分系统,为评分失衡提供补充二、项目目标自动化抓取携程指定景点的用户评论清洗与分词评论文本对评论进行情感分析打分分析整体情绪趋势并可视化结果三、技术栈与工具模块工具/库数据爬取requests,re,json
- 从0到1构建智能招聘数据引擎:基于 Python 的 BOSS直聘信息采集实战与反爬破解指南
程序员威哥
python开发语言
前言在大数据浪潮席卷的时代,招聘平台蕴藏着海量的岗位信息,揭示着行业走向、人才趋势、薪资结构等核心价值。BOSS直聘作为国内极具代表性的直招平台,其数据对职业分析、市场监测甚至智能推荐系统的构建都有着重要意义。本文将手把手带你打造一个高质量、抗封锁的Python爬虫系统,精准采集BOSS直聘网的岗位数据,并全面解析其中涉及的反爬机制识别、加密参数处理、数据提取与存储等高级技巧,助你在Web数据采集
- Enum 枚举
120153216
enum枚举
原文地址:http://www.cnblogs.com/Kavlez/p/4268601.html Enumeration
于Java 1.5增加的enum type...enum type是由一组固定的常量组成的类型,比如四个季节、扑克花色。在出现enum type之前,通常用一组int常量表示枚举类型。比如这样:
public static final int APPLE_FUJI = 0
- Java8简明教程
bijian1013
javajdk1.8
Java 8已于2014年3月18日正式发布了,新版本带来了诸多改进,包括Lambda表达式、Streams、日期时间API等等。本文就带你领略Java 8的全新特性。
一.允许在接口中有默认方法实现
Java 8 允许我们使用default关键字,为接口声明添
- Oracle表维护 快速备份删除数据
cuisuqiang
oracle索引快速备份删除
我知道oracle表分区,不过那是数据库设计阶段的事情,目前是远水解不了近渴。
当前的数据库表,要求保留一个月数据,且表存在大量录入更新,不存在程序删除。
为了解决频繁查询和更新的瓶颈,我在oracle内根据需要创建了索引。但是随着数据量的增加,一个半月数据就要超千万,此时就算有索引,对高并发的查询和更新来说,让然有所拖累。
为了解决这个问题,我一般一个月会进行一次数据库维护,主要工作就是备
- java多态内存分析
麦田的设计者
java内存分析多态原理接口和抽象类
“ 时针如果可以回头,熟悉那张脸,重温嬉戏这乐园,墙壁的松脱涂鸦已经褪色才明白存在的价值归于记忆。街角小店尚存在吗?这大时代会不会牵挂,过去现在花开怎么会等待。
但有种意外不管痛不痛都有伤害,光阴远远离开,那笑声徘徊与脑海。但这一秒可笑不再可爱,当天心
- Xshell实现Windows上传文件到Linux主机
被触发
windows
经常有这样的需求,我们在Windows下载的软件包,如何上传到远程Linux主机上?还有如何从Linux主机下载软件包到Windows下;之前我的做法现在看来好笨好繁琐,不过也达到了目的,笨人有本方法嘛;
我是怎么操作的:
1、打开一台本地Linux虚拟机,使用mount 挂载Windows的共享文件夹到Linux上,然后拷贝数据到Linux虚拟机里面;(经常第一步都不顺利,无法挂载Windo
- 类的加载ClassLoader
肆无忌惮_
ClassLoader
类加载器ClassLoader是用来将java的类加载到虚拟机中,类加载器负责读取class字节文件到内存中,并将它转为Class的对象(类对象),通过此实例的 newInstance()方法就可以创建出该类的一个对象。
其中重要的方法为findClass(String name)。
如何写一个自己的类加载器呢?
首先写一个便于测试的类Student
- html5写的玫瑰花
知了ing
html5
<html>
<head>
<title>I Love You!</title>
<meta charset="utf-8" />
</head>
<body>
<canvas id="c"></canvas>
- google的ConcurrentLinkedHashmap源代码解析
矮蛋蛋
LRU
原文地址:
http://janeky.iteye.com/blog/1534352
简述
ConcurrentLinkedHashMap 是google团队提供的一个容器。它有什么用呢?其实它本身是对
ConcurrentHashMap的封装,可以用来实现一个基于LRU策略的缓存。详细介绍可以参见
http://code.google.com/p/concurrentlinke
- webservice获取访问服务的ip地址
alleni123
webservice
1. 首先注入javax.xml.ws.WebServiceContext,
@Resource
private WebServiceContext context;
2. 在方法中获取交换请求的对象。
javax.xml.ws.handler.MessageContext mc=context.getMessageContext();
com.sun.net.http
- 菜鸟的java基础提升之道——————>是否值得拥有
百合不是茶
1,c++,java是面向对象编程的语言,将万事万物都看成是对象;java做一件事情关注的是人物,java是c++继承过来的,java没有直接更改地址的权限但是可以通过引用来传值操作地址,java也没有c++中繁琐的操作,java以其优越的可移植型,平台的安全型,高效性赢得了广泛的认同,全世界越来越多的人去学习java,我也是其中的一员
java组成:
- 通过修改Linux服务自动启动指定应用程序
bijian1013
linux
Linux中修改系统服务的命令是chkconfig (check config),命令的详细解释如下: chkconfig
功能说明:检查,设置系统的各种服务。
语 法:chkconfig [ -- add][ -- del][ -- list][系统服务] 或 chkconfig [ -- level <</SPAN>
- spring拦截器的一个简单实例
bijian1013
javaspring拦截器Interceptor
Purview接口
package aop;
public interface Purview {
void checkLogin();
}
Purview接口的实现类PurviesImpl.java
package aop;
public class PurviewImpl implements Purview {
public void check
- [Velocity二]自定义Velocity指令
bit1129
velocity
什么是Velocity指令
在Velocity中,#set,#if, #foreach, #elseif, #parse等,以#开头的称之为指令,Velocity内置的这些指令可以用来做赋值,条件判断,循环控制等脚本语言必备的逻辑控制等语句,Velocity的指令是可扩展的,即用户可以根据实际的需要自定义Velocity指令
自定义指令(Directive)的一般步骤
&nbs
- 【Hive十】Programming Hive学习笔记
bit1129
programming
第二章 Getting Started
1.Hive最大的局限性是什么?一是不支持行级别的增删改(insert, delete, update)二是查询性能非常差(基于Hadoop MapReduce),不适合延迟小的交互式任务三是不支持事务2. Hive MetaStore是干什么的?Hive persists table schemas and other system metadata.
- nginx有选择性进行限制
ronin47
nginx 动静 限制
http {
limit_conn_zone $binary_remote_addr zone=addr:10m;
limit_req_zone $binary_remote_addr zone=one:10m rate=5r/s;...
server {...
location ~.*\.(gif|png|css|js|icon)$ {
- java-4.-在二元树中找出和为某一值的所有路径 .
bylijinnan
java
/*
* 0.use a TwoWayLinkedList to store the path.when the node can't be path,you should/can delete it.
* 1.curSum==exceptedSum:if the lastNode is TreeNode,printPath();delete the node otherwise
- Netty学习笔记
bylijinnan
javanetty
本文是阅读以下两篇文章时:
http://seeallhearall.blogspot.com/2012/05/netty-tutorial-part-1-introduction-to.html
http://seeallhearall.blogspot.com/2012/06/netty-tutorial-part-15-on-channel.html
我的一些笔记
===
- js获取项目路径
cngolon
js
//js获取项目根路径,如: http://localhost:8083/uimcardprj
function getRootPath(){
//获取当前网址,如: http://localhost:8083/uimcardprj/share/meun.jsp
var curWwwPath=window.document.locati
- oracle 的性能优化
cuishikuan
oracleSQL Server
在网上搜索了一些Oracle性能优化的文章,为了更加深层次的巩固[边写边记],也为了可以随时查看,所以发表这篇文章。
1.ORACLE采用自下而上的顺序解析WHERE子句,根据这个原理,表之间的连接必须写在其他WHERE条件之前,那些可以过滤掉最大数量记录的条件必须写在WHERE子句的末尾。(这点本人曾经做过实例验证过,的确如此哦!
- Shell变量和数组使用详解
daizj
linuxshell变量数组
Shell 变量
定义变量时,变量名不加美元符号($,PHP语言中变量需要),如:
your_name="w3cschool.cc"
注意,变量名和等号之间不能有空格,这可能和你熟悉的所有编程语言都不一样。同时,变量名的命名须遵循如下规则:
首个字符必须为字母(a-z,A-Z)。
中间不能有空格,可以使用下划线(_)。
不能使用标点符号。
不能使用ba
- 编程中的一些概念,KISS、DRY、MVC、OOP、REST
dcj3sjt126com
REST
KISS、DRY、MVC、OOP、REST (1)KISS是指Keep It Simple,Stupid(摘自wikipedia),指设计时要坚持简约原则,避免不必要的复杂化。 (2)DRY是指Don't Repeat Yourself(摘自wikipedia),特指在程序设计以及计算中避免重复代码,因为这样会降低灵活性、简洁性,并且可能导致代码之间的矛盾。 (3)OOP 即Object-Orie
- [Android]设置Activity为全屏显示的两种方法
dcj3sjt126com
Activity
1. 方法1:AndroidManifest.xml 里,Activity的 android:theme 指定为" @android:style/Theme.NoTitleBar.Fullscreen" 示例: <application
- solrcloud 部署方式比较
eksliang
solrCloud
solrcloud 的部署其实有两种方式可选,那么我们在实践开发中应该怎样选择呢? 第一种:当启动solr服务器时,内嵌的启动一个Zookeeper服务器,然后将这些内嵌的Zookeeper服务器组成一个集群。 第二种:将Zookeeper服务器独立的配置一个集群,然后将solr交给Zookeeper进行管理
谈谈第一种:每启动一个solr服务器就内嵌的启动一个Zoo
- Java synchronized关键字详解
gqdy365
synchronized
转载自:http://www.cnblogs.com/mengdd/archive/2013/02/16/2913806.html
多线程的同步机制对资源进行加锁,使得在同一个时间,只有一个线程可以进行操作,同步用以解决多个线程同时访问时可能出现的问题。
同步机制可以使用synchronized关键字实现。
当synchronized关键字修饰一个方法的时候,该方法叫做同步方法。
当s
- js实现登录时记住用户名
hw1287789687
记住我记住密码cookie记住用户名记住账号
在页面中如何获取cookie值呢?
如果是JSP的话,可以通过servlet的对象request 获取cookie,可以
参考:http://hw1287789687.iteye.com/blog/2050040
如果要求登录页面是html呢?html页面中如何获取cookie呢?
直接上代码了
页面:loginInput.html
代码:
<!DOCTYPE html PUB
- 开发者必备的 Chrome 扩展
justjavac
chrome
Firebug:不用多介绍了吧https://chrome.google.com/webstore/detail/bmagokdooijbeehmkpknfglimnifench
ChromeSnifferPlus:Chrome 探测器,可以探测正在使用的开源软件或者 js 类库https://chrome.google.com/webstore/detail/chrome-sniffer-pl
- 算法机试题
李亚飞
java算法机试题
在面试机试时,遇到一个算法题,当时没能写出来,最后是同学帮忙解决的。
这道题大致意思是:输入一个数,比如4,。这时会输出:
&n
- 正确配置Linux系统ulimit值
字符串
ulimit
在Linux下面部 署应用的时候,有时候会遇上Socket/File: Can’t open so many files的问题;这个值也会影响服务器的最大并发数,其实Linux是有文件句柄限制的,而且Linux默认不是很高,一般都是1024,生产服务器用 其实很容易就达到这个数量。下面说的是,如何通过正解配置来改正这个系统默认值。因为这个问题是我配置Nginx+php5时遇到了,所以我将这篇归纳进
- hibernate调用返回游标的存储过程
Supanccy2013
javaDAOoracleHibernatejdbc
注:原创作品,转载请注明出处。
上篇博文介绍的是hibernate调用返回单值的存储过程,本片博文说的是hibernate调用返回游标的存储过程。
此此扁博文的存储过程的功能相当于是jdbc调用select 的作用。
1,创建oracle中的包,并在该包中创建的游标类型。
---创建oracle的程
- Spring 4.2新特性-更简单的Application Event
wiselyman
application
1.1 Application Event
Spring 4.1的写法请参考10点睛Spring4.1-Application Event
请对比10点睛Spring4.1-Application Event
使用一个@EventListener取代了实现ApplicationListener接口,使耦合度降低;
1.2 示例
包依赖
<p