【树模型与集成学习】(task4)两种并行集成的树模型
学习总结
(1)随机森林中的随机主要来自三个方面:
- 其一为bootstrap抽样导致的训练集随机性,
- 其二为每个节点随机选取特征子集进行不纯度计算的随机性,
- 其三为当使用随机分割点选取时产生的随机性(此时的随机森林又被称为Extremely Randomized Trees)。
(2)孤立森林算法类比在切蛋糕,点密度稠密的一块需要切很多刀才能分割完,而那些很早能分割完的应该是异常点(我们要把每个点都单独存在一个子空间中)。
孤立森林通过随机选择特征,然后随机选择特征的分割值,递归地生成数据集的分区。和数据集中「正常」的点相比,要隔离的异常值所需的随机分区更少,因此异常值是树中路径更短的点,路径长度是从根节点经过的边数。
【内容概要】理解随机森林的训练和预测流程,特征重要性和oob得分计算,孤立森林的原理以及训练和预测流程
【打卡内容】侧边栏练习,知识回顾后三题,实现孤立森林算法和用于分类的随机森林算法(可以用sklearn的决策树或task2中自己实现的分类cart树)
文章目录
- 学习总结
- 一、随机森林
-
- 1.1 算法介绍
- 1.2 Totally Random Trees Embedding
- 二、孤立森林
-
- 2.1 异常得分的计算
- 2.2 限定树的高度
- 2.3 训练与预测
- 三、作业
-
- 5.1 什么是随机森林的oob得分?
- 5.2 随机森林是如何集成多个决策树模型的?
- 5.3 叙述孤立森林的算法原理和流程。
- 5.4 实现孤立森林算法
- 5.5 实现用于分类的随机森林算法
-
- 5.6 随机森林的主要参数
-
- (1)RF框架的参数
- (2)RF决策树的参数
- Reference
一、随机森林
1.1 算法介绍
随机森林是以决策树(常用CART树)为基学习器的bagging算法。
(1)随机森林当处理回归问题时,输出值为各学习器的均值;
(2)随机森林当处理分类问题时有两种策略:
- 第一种是原始论文中使用的投票策略,即每个学习器输出一个类别,返回最高预测频率的类别;
- 第二种是sklearn中采用的概率聚合策略,即通过各个学习器输出的概率分布先计算样本属于某个类别的平均概率,在对平均的概率分布取 arg max \arg\max argmax以输出最可能的类别。
随机森林中的随机主要来自三个方面:
- 其一为bootstrap抽样导致的训练集随机性,
- 其二为每个节点随机选取特征子集进行不纯度计算的随机性,
- 其三为当使用随机分割点选取时产生的随机性(此时的随机森林又被称为Extremely Randomized Trees)。
随机森林中特征重要性的计算方式为:利用相对信息增益来度量单棵树上的各特征特征重要性(与决策树计算方式一致),再通过对所有树产出的重要性得分进行简单平均来作为最终的特征重要性。
【练习】
r2_score和均方误差的区别是什么?它具有什么优势?
r2_score是判定系数:回归模型的方差系数。
r2_score的计算公式如下:(本质上是以均值模型作为baseline model,计算该模型相较于它的好坏) R 2 ( y , y ^ ) = 1 − ∑ i = 0 n − 1 ( y i − y i ^ ) 2 ∑ i = 0 n − 1 ( y i − y ‾ ) 2 R^2(y,\hat{y})=1-\frac{\sum_{i=0}^{n-1}(y_i-\hat{y_i})^2}{\sum_{i=0}^{n-1}(y_i-\overline{y})^2} R2(y,y^)=1−∑i=0n−1(yi−y)2∑i=0n−1(yi−yi^)2MSE是均方误差,即线性回归的损失函数,计算公式如下: M S E ( y , y ^ ) = 1 n ∑ i = 0 n − 1 ( y i − y ^ i ) 2 MSE(y,\hat{y})=\frac{1}{n}\sum_{i=0}^{n-1}(y_i-\hat{y}_i)^2 MSE(y,y^)=n1i=0∑n−1(yi−y^i)2其中分子是训练出的模型的所有误差,分母是使用y真=y真平均 预测产生的误差。
二者的区别是 R 2 ( y , y ^ ) = 1 − M S E ( y , y ^ ) σ 2 R^2(y,\hat{y})=1-\frac{MSE(y,\hat{y})}{\sigma^2} R2(y,y^)=1−σ2MSE(y,y^),其中 σ \sigma σ表示y的标准差。
MSE是带量纲的,而且结果为量纲的平方,而r2_score是不带量纲的,可以比较模型在不同量纲数据(不同问题)上的好坏。
【oob样本】:在训练时,一般而言我们总是需要对数据集进行训练集和验证集的划分,但随机森林由于每一个基学习器使用了重复抽样得到的数据集进行训练,因此总存在比例大约为 e − 1 e^{-1} e−1的数据集没有参与训练,这一部分数据称为out-of-bag样本(即oob样本)。
对每一个基学习器训练完毕后,我们都对oob样本进行预测,每个样本对应的oob_prediction_值为所有没有采样到该样本进行训练的基学习器预测结果均值,这一部分的逻辑参见此处的源码实现。
在得到所有样本的oob_prediction_后:
(1)对于回归问题,使用r2_score来计算对应的oob_score_;
(2)而对于分类问题,直接使用accuracy_score来计算oob_score_。
1.2 Totally Random Trees Embedding
介绍一种Totally Random Trees Embedding方法,它能够基于每个样本在各个决策树上的叶节点位置,得到一种基于森林的样本特征嵌入。
【栗子】假设现在有4棵树且每棵树有4个叶子节点(共16个节点),依次对它们进行从0至15的编号,记样本 i i i在4棵树叶子节点的位置编号为 [ 0 , 7 , 8 , 14 ] [0,7,8,14] [0,7,8,14],样本 j j j的编号为 [ 1 , 7 , 9 , 13 ] [1,7,9,13] [1,7,9,13],此时这两个样本的嵌入向量即为 [ 1 , 0 , 0 , 0 , 0 , 0 , 0 , 1 , 1 , 0 , 0 , 0 , 0 , 0 , 1 , 0 ] [1,0,0,0,0,0,0,1,1,0,0,0,0,0,1,0] [1,0,0,0,0,0,0,1,1,0,0,0,0,0,1,0]和 [ 0 , 1 , 0 , 0 , 0 , 0 , 0 , 1 , 0 , 1 , 0 , 0 , 0 , 1 , 0 , 0 ] [0,1,0,0,0,0,0,1,0,1,0,0,0,1,0,0] [0,1,0,0,0,0,0,1,0,1,0,0,0,1,0,0]
假设样本 k k k对应的编号为 [ 0 , 6 , 8 , 14 ] [0,6,8,14] [0,6,8,14],那么其对应嵌入向量的距离应当和样本 i i i 较近,而离样本 j j j 较远,即两个样本在不同树上分配到相同的叶子结点次数越多,则越接近。因此,这个方法巧妙地利用树结构获得了样本的隐式特征。
【练习】假设使用闵氏距离来度量两个嵌入向量之间的距离,此时对叶子节点的编号顺序会对距离的度量结果有影响吗?
没有关系。
闵式距离为: D ( x , y ) = ( ∑ u = 1 n ∣ x u − y u ∣ p ) 1 p D(x,y)=(\sum_{u=1}^n|x_u-y_u|^p)^{\frac{1}{p}} D(x,y)=(u=1∑n∣xu−yu∣p)p1 嵌入向量是依据决策森林样本叶节点落位而进行multi_hot encoding的一个结果(对应位取值为1,其他为0),只要叶子节点编号的每个维度的权重一样(这里都是1)。
二、孤立森林
孤立森林算法是基于 Ensemble 的异常检测方法,因此具有线性的时间复杂度。且精准度较高,在处理大数据时速度快,所以目前在工业界的应用范围比较广。常见的场景包括:网络安全中的攻击检测、金融交易欺诈检测、疾病侦测、噪声数据过滤(数据清洗)等。
孤立森林是基于决策树的算法。从给定的特征集合中随机选择特征,然后在特征的最大值和最小值间随机选择一个分割值,来隔离离群值。这种特征的随机划分会使异常数据点在树中生成的路径更短,从而将它们和其他数据分开。孤立森林不通过显式地隔离异常,来隔离了数据集中的异常点。
孤立森林的基本思想是:多次随机选取特征和对应的分割点以分开空间中样本点,那么异常点很容易在较早的几次分割中就已经与其他样本隔开,正常点由于较为紧密故需要更多的分割次数才能将其分开。
孤立森林的优势:
- Partial models:在训练过程中,每棵孤立树都是随机选取部分样本;
- No distance or density measures:不同于 KMeans、DBSCAN 等算法,孤立森林不需要计算有关距离、密度的指标,可大幅度提升速度,减小系统开销;
- Linear time complexity:因为基于 ensemble,所以有线性时间复杂度。通常树的数量越多,算法越稳定;
- Handle extremely large data size:由于每棵树都是独立生成的,因此可部署在大规模分布式系统上来加速运算。
2.1 异常得分的计算
下图中体现了两个特征下的4次分割过程,可见右上角的异常点已经被单独隔离开。

对于 n n n 个样本而言,我们可以构建一棵在每个分支进行特征大小判断的树来将样本分派到对应的叶子节点,为了定量刻画异常情况,在这篇文献中证明了树中的平均路径(即树的根节点到叶子结点经过的节点数)长度 c c c为
c ( n ) = 2 H ( n − 1 ) − 2 ( n − 1 ) n c(n) = 2H(n-1)-\frac{2(n-1)}{n} c(n)=2H(n−1)−n2(n−1)
其中 H ( k ) H(k) H(k)为调和级数 ∑ p = 1 k 1 p \sum_{p=1}^k\frac{1}{p} p=1∑kp1
此时对于某个样本 x x x,假设其分派到叶子节点的路径长度为 h ( x ) h(x) h(x),我们就能用 h ( x ) c ( n ) \frac{h(x)}{c(n)} c(n)h(x)的大小来度量异常的程度,该值越小则越有可能为异常点。由于单棵树上使用的是随机特征的随机分割点,稳健度较差,因此孤立森林将建立 t t t棵树(默认100),每棵树上都在数据集上抽样出 ψ \psi ψ个样本(默认256个)进行训练。为了总和集成的结果,我们定义指标——异常得分
s ( x , n ) = 2 − E h ( x ) c ( n ) s(x,n)=2^{-\frac{\mathbb{E}h(x)}{c(n)}} s(x,n)=2−c(n)Eh(x)
指数上的 E h ( x ) \mathbb{E}h(x) Eh(x)表示样本 x x x在各树的路径平均值:
(1)当这个均值趋于0时,异常得分 s ( x , n ) s(x,n) s(x,n)趋于1;
(2)当其趋于 n − 1 n-1 n−1时( n n n个样本最多需要 n − 1 n-1 n−1次分割,故树深度最大为 n − 1 n-1 n−1), s ( x , n ) s(x,n) s(x,n)趋于0(特别是在大样本情况下 c ( n ) c(n) c(n)远小于 E h ( x ) \mathbb{E}h(x) Eh(x));
(3)当其趋于平均路径长度 E h ( x ) \mathbb{E}h(x) Eh(x)时, s ( x , n ) s(x,n) s(x,n)趋于 1 2 \frac{1}{2} 21。变化关系如图所示。

2.2 限定树的高度
虽然上述步骤明确了异常得分的计算,但是却还没有说明训练时树究竟应当在何时生长停止。可以规定树的生长停止当且仅当树的高度(路径的最大值)达到了给定的限定高度,或者叶子结点样本数仅为1,或者叶子节点样本数的所有特征值完全一致(即空间中的点重合,无法分离)。
如何决定树的限定高度呢?
在异常点判别的问题中,异常点往往是少部分的因此绝大多数的异常点都会在高度达到 c ( n ) c(n) c(n)前被分配至路径较短的叶子结点,由于调和级数有如下关系(其中 γ ≈ 0.5772 \gamma\approx0.5772 γ≈0.5772为欧拉常数):
lim n → ∞ H ( n ) − log n = γ \lim_{n\to\infty} H(n)-\log n = \gamma n→∞limH(n)−logn=γ
因此 c ( n ) c(n) c(n)与 log n \log n logn数量级相同,故给定的限定高度可以设置为 log n \log n logn。
2.3 训练与预测
此时,我们可以写出模型训练的伪代码(图片直接来自于原始论文):
首先下面这段是创建孤立树。树的高度限制I与子样本数量有关。限制树的高度是因为我们需要找路径长度较短的点(它们更可能是异常点)。

第二段就是是每课孤立树的生长(即训练过程)。

在预测阶段,应当计算样本在每棵树上被分配到叶子的路径,但我们还需要在路径长度上加上一项 c ( T . s i z e ) c(T.size) c(T.size),其含义为当前叶子节点上样本数对应可生长的平均路径值,这是由于我们为了快速训练而对树高度进行了限制,事实上那些多个样本的节点仍然可以生长下去得到更长的路径,而新样本到达叶子结点后,平均而言还需要 c ( T . s i z e ) c(T.size) c(T.size)的路径才能达到真正(完全生长的树)的叶子结点。从而计算路径的伪代码如下图所示:

在得到了各个树对应的路径后,我们就自然能计算 s ( x , n ) s(x,n) s(x,n)了。假设我们需要得到前 5 % 5\% 5%最可能为异常的点时,只需要对所有新样本的 s ( x , n ) s(x,n) s(x,n)排序后输出前 5 % 5\% 5%大的异常得分值对应的样本即可。
三、作业
5.1 什么是随机森林的oob得分?
在训练时,一般而言我们总是需要对数据集进行训练集和验证集的划分,但随机森林由于每一个基学习器使用了重复抽样得到的数据集进行训练,因此总存在比例大约为 e − 1 e^{-1} e−1的数据集没有参与训练,我们把这一部分数据称为out-of-bag样本,简称oob样本。
每一个基学习器训练完后,我们都对oob数据进行预测,每个样本对应的oob_prediction_ 值为所有没有采样到该样本进行训练的基学习器预测结果均值。在得到所有的oob_prediction_ 的值后,如果问题是回归问题,则用r2_score来计算oob_score_ ,如果问题是分类问题,则用accuarcy_score来计算oob得分。
5.2 随机森林是如何集成多个决策树模型的?
(1)随机森林当处理回归问题时,输出值为各学习器的均值;
(2)随机森林当处理分类问题时有两种策略:
- 第一种是原始论文中使用的投票策略,即每个学习器输出一个类别,返回最高预测频率的类别;
- 第二种是sklearn中采用的概率聚合策略,即通过各个学习器输出的概率分布先计算样本属于某个类别的平均概率,在对平均的概率分布取 arg max \arg\max argmax以输出最可能的类别。
(绝对平均和加权平均的区别)
5.3 叙述孤立森林的算法原理和流程。
主要思想:多次随机选取特征和对应的分割点以分开空间中样本点,那么异常点很容易在较早的几次分割中就已经与其他样本隔开,正常点由于较为紧密故需要更多的分割次数才能将其分开。
孤立森林通过随机选择特征,然后随机选择特征的分割值,递归地生成数据集的分区。和数据集中「正常」的点相比,要隔离的异常值所需的随机分区更少,因此异常值是树中路径更短的点,路径长度是从根节点经过的边数。
孤立森林算法的流程:
(1)训练iForest:
从训练集中采样,构建孤立树,对森林中每棵孤立树进行测试,记录路径长度;
(2)计算异常分数:
代入论文中证明的异常得分公式 s ( x , n ) = 2 − E h ( x ) c ( n ) s(x,n)=2^{-\frac{\mathbb{E}h(x)}{c(n)}} s(x,n)=2−c(n)Eh(x)指数上的 E h ( x ) \mathbb{E}h(x) Eh(x)表示样本 x x x在各树的路径平均值:
(1)当这个均值趋于0时,异常得分 s ( x , n ) s(x,n) s(x,n)趋于1;
(2)当其趋于 n − 1 n-1 n−1时( n n n个样本最多需要 n − 1 n-1 n−1次分割,故树深度最大为 n − 1 n-1 n−1), s ( x , n ) s(x,n) s(x,n)趋于0(特别是在大样本情况下 c ( n ) c(n) c(n)远小于 E h ( x ) \mathbb{E}h(x) Eh(x));
(3)当其趋于平均路径长度 E h ( x ) \mathbb{E}h(x) Eh(x)时, s ( x , n ) s(x,n) s(x,n)趋于 1 2 \frac{1}{2} 21。变化关系如图所示。

5.4 实现孤立森林算法
from pyod.utils.data import generate_data
import matplotlib.pyplot as plt
import numpy as np
class Node:
def __init__(self, depth):
self.depth = depth
self.left = None
self.right = None
self.feature = None
self.pivot = None
class Tree:
def __init__(self, max_height):
self.root = Node(0)
self.max_height = max_height
self.c = None
def _build(self, node, X,):
if X.shape[0] == 1:
return
if node.depth+1 > self.max_height:
node.depth += self._c(X.shape[0])
return
node.feature = np.random.randint(X.shape[1])
pivot_min = X[:, node.feature].min()
pivot_max = X[:, node.feature].max()
node.pivot = np.random.uniform(pivot_min, pivot_max)
node.left, node.right = Node(node.depth+1), Node(node.depth+1)
self._build(node.left, X[X[:, node.feature]<node.pivot])
self._build(node.right, X[X[:, node.feature]>=node.pivot])
def build(self, X):
self.c = self._c(X.shape[0])
self._build(self.root, X)
def _c(self, n):
if n == 1:
return 0
else:
return 2 * ((np.log(n-1) + 0.5772) - (n-1)/n)
# 查找某个样本的深度(即h(x))
def _get_h_score(self, node, x):
if node.left is None and node.right is None:
return node.depth
if x[node.feature] < node.pivot:
return self._get_h_score(node.left, x)
else:
return self._get_h_score(node.right, x)
def get_h_score(self, x):
return self._get_h_score(self.root, x)
class IsolationForest:
def __init__(self, n_estimators=100, max_samples=256):
self.n_estimator = n_estimators
self.max_samples = max_samples
self.trees = []
def fit(self, X):
for tree_id in range(self.n_estimator):
# 取样
random_X = X[np.random.randint(0, X.shape[0], self.max_samples)]
tree = Tree(np.log(random_X.shape[0]))
tree.build(X)
# trees树列表增加新树
self.trees.append(tree)
def predict(self, X):
result = []
for x in X:
h = 0
for tree in self.trees:
h += tree.get_h_score(x) / tree.c
# power
score = np.power(2, - h/len(self.trees))
result.append(score)
return np.array(result)
if __name__ == "__main__":
np.random.seed(0)
# 生成数据,1%异常点,generate_data用的还是pyod库的函数
X_train, X_test, y_train, y_test = generate_data(
n_train=1000, n_test=500,
contamination=0.05, behaviour="new", random_state=0
)
IF = IsolationForest()
IF.fit(X_train)
res = IF.predict(X_test)
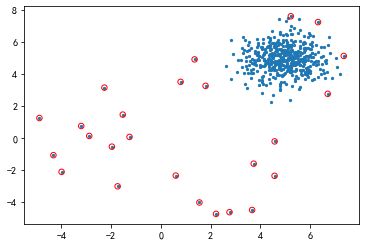
abnormal_X = X_test[res > np.quantile(res, 0.95)]
# 数据可视化
plt.scatter(X_test[:, 0], X_test[:, 1], s=5)
plt.scatter(
abnormal_X[:, 0], abnormal_X[:, 1],
s=30, edgecolors="Red", facecolor="none"
)
plt.show()
5.5 实现用于分类的随机森林算法
(可以用sklearn的决策树或task2中自己实现的分类cart树)
# -*- coding: utf-8 -*-
"""
Created on Sun Oct 24 22:39:03 2021
@author: 86493
"""
import numpy as np
from sklearn.tree import DecisionTreeClassifier as Tree
class RandomForest:
def __init__(self, n_estimators, max_depth):
self.n_estimators = n_estimators
self.max_depth = max_depth
self.trees = []
def fit(self, X, y):
for tree_id in range(self.n_estimators):
indexes = np.random.randint(0, X.shape[0], X.shape[0])
random_X = X[indexes]
random_y = y[indexes]
tree = Tree(max_depth=3)
tree.fit(random_X, random_y)
self.trees.append(tree)
def predict(self, X):
results = []
for x in X:
result = []
for tree in self.trees:
result.append(tree.predict(x.reshape(1, -1))[0])
results.append(np.argmax(np.bincount(result))) # 返回该样本的预测结果,采取方案:多数投票
return np.array(results)
from sklearn.datasets import make_classification
from sklearn.ensemble import RandomForestClassifier as RF
# from RandomForest_Classifier import RandomForest
import numpy as np
if __name__ == "__main__":
X, y = make_classification(n_samples=200, n_features=8, n_informative=4, random_state=0)
RF1 = RandomForest(n_estimators=100, max_depth=3)
RF2 = RF(n_estimators=100, max_depth=3)
RF1.fit(X, y)
res1 = RF1.predict(X)
RF2.fit(X, y)
res2 = RF2.predict(X)
print('预测一致的比例', (np.abs(res1 - res2) < 1e-5).mean())
# 预测一致的比例 0.985
5.6 随机森林的主要参数
(1)RF框架的参数
RF框架的参数:RandomForestClassifier和RandomForestRegressor参数绝大部分相同。
(1)n_estimators(重点): 也就是最大的弱学习器的个数。一般来说n_estimators太小,容易欠拟合,n_estimators太大,计算量会太大,并且n_estimators到一定的数量后,再增大n_estimators获得的模型提升会很小,所以一般选择一个适中的数值。默认是100。
(2)oob_score :即是否采用袋外样本来评估模型的好坏。默认识False。个人推荐设置为True,因为袋外分数反应了一个模型拟合后的泛化能力。
(3)criterion: 即CART树做划分时对特征的评价标准。分类模型和回归模型的损失函数是不一样的。分类RF对应的CART分类树默认是基尼系数gini,另一个可选择的标准是信息增益。回归RF对应的CART回归树默认是均方差mse,另一个可以选择的标准是绝对值差mae。一般来说选择默认的标准就已经很好的。
(2)RF决策树的参数
(1)RF划分时考虑的最大特征数max_features: 可以使用很多种类型的值,默认是"auto",意味着划分时最多考虑 N \sqrt{N} N个特征;如果是"log2"意味着划分时最多考虑 log 2 N \log _{2} N log2N个特征;如果是"sqrt"或者"auto"意味着划分时最多考虑 N \sqrt{N} N个特征。如果是整数,代表考虑的特征绝对数。如果是浮点数,代表考虑特征百分比,即考虑(百分比xN)取整后的特征数。其中N为样本总特征数。一般我们用默认的"auto"就可以了,如果特征数非常多,我们可以灵活使用刚才描述的其他取值来控制划分时考虑的最大特征数,以控制决策树的生成时间。
(2)决策树最大深度max_depth: 默认可以不输入,如果不输入的话,决策树在建立子树的时候不会限制子树的深度。一般来说,数据少或者特征少的时候可以不管这个值。如果模型样本量多,特征也多的情况下,推荐限制这个最大深度,具体的取值取决于数据的分布。常用的可以取值10-100之间。
(3)内部节点再划分所需最小样本数min_samples_split: 这个值限制了子树继续划分的条件,如果某节点的样本数少于min_samples_split,则不会继续再尝试选择最优特征来进行划分。 默认是2.如果样本量不大,不需要管这个值。如果样本量数量级非常大,则推荐增大这个值。
(4)叶子节点最少样本数min_samples_leaf: 这个值限制了叶子节点最少的样本数,如果某叶子节点数目小于样本数,则会和兄弟节点一起被剪枝。 默认是1,可以输入最少的样本数的整数,或者最少样本数占样本总数的百分比。如果样本量不大,不需要管这个值。如果样本量数量级非常大,则推荐增大这个值。
(5)叶子节点最小的样本权重和min_weight_fraction_leaf:这个值限制了叶子节点所有样本权重和的最小值,如果小于这个值,则会和兄弟节点一起被剪枝。 默认是0,就是不考虑权重问题。一般来说,如果我们有较多样本有缺失值,或者分类树样本的分布类别偏差很大,就会引入样本权重,这时我们就要注意这个值了。
(6)最大叶子节点数max_leaf_nodes: 通过限制最大叶子节点数,可以防止过拟合,默认是"None”,即不限制最大的叶子节点数。如果加了限制,算法会建立在最大叶子节点数内最优的决策树。如果特征不多,可以不考虑这个值,但是如果特征分成多的话,可以加以限制,具体的值可以通过交叉验证得到。
(7)节点划分最小不纯度min_impurity_split: 这个值限制了决策树的增长,如果某节点的不纯度(基于基尼系数,均方差)小于这个阈值,则该节点不再生成子节点。即为叶子节点 。一般不推荐改动默认值1e-7。
上面决策树参数中最重要的包括最大特征数max_features, 最大深度max_depth, 内部节点再划分所需最小样本数min_samples_split和叶子节点最少样本数min_samples_leaf。
Reference
(1)https://datawhalechina.github.io/machine-learning-toy-code/index.html
(2)https://zhuanlan.zhihu.com/p/113325296
(3)异常检测算法 – 孤立森林(Isolation Forest)剖析—best
(4)https://github.com/buaamse/2021-08/blob/main/task04_RandomiForestTree.ipynb
(5)机器之心—孤立森林进行异常检测(sklearn版)
(7)a同学笔记,b同学笔记
(8)scikit-learn随机森林调参小结-刘建平