A graph auto-encoder model for miRNA-disease associations prediction 论文解析
A graph auto-encoder model for miRNA-disease associations prediction(用于miRNA-disease关联预测的图自动编码模型)该文章由中科院新疆理化所的尤老师组,发表在《Briefings in Bioinformatics》期刊上
文章目录
- 摘要
- 一、Introduction
- 二、Materials and methods
-
- 1.Human miRNA-disease associations
- 2.MiRNA functional similarity
- 3.Disease semantic similarity
- 4.Gaussian interaction profile kernel similarity for miRNAs and diseases
- 5.Integrated similarity for miRNAs and diseases
- 6.GAEMDA
- 三、Results
-
- 1.Implementation details and evaluation metrics
- 2.Performance evaluation
- 3.Effect of projection dimensions
- 4.Effect of the number of encoder layers
- 5.Comparison of GAEMDA with other related models
- 6.Case studies
- 四、Discussion
- 五、Conclusion
- 个人总结
摘要
新的证据表明,miRNA的异常表达参与了各种人类复杂疾病的进化和发展。识别疾病相关miRNA作为新的生物标志物,可以促进疾病病理学和临床医学的发展。然而,设计生物实验来验证疾病相关的miRNA通常既耗时又昂贵。因此,迫切需要设计有效的计算方法来预测潜在的miRNA-疾病关联。受图神经网络在链接预测方面的巨大进步的启发,我们提出了一种新的图自动编码模型GAEMDA,以端到端的方式识别潜在的miRNA-疾病关联。更具体地说,GAEMDA模型采用基于图神经网络的编码器,该编码器包含聚合功能和用于聚集节点邻域信息的多层感知器,以生成miRNA和疾病节点的低维嵌入,并实现异构信息的有效融合。然后,miRNA和疾病节点的嵌入被馈送到双线性解码器,以识别miRNA和疾病节点之间的潜在联系。实验结果表明,在5倍交叉验证下,该算法在曲线下的平均面积为93.56±0.44%。此外,我们还开展了结肠肿瘤、食管肿瘤和肾脏肿瘤的病例研究。结果,与这些疾病相关的前50个预测的miRNA中的48个分别被人类癌症中差异表达的微小核糖核酸数据库和人类疾病数据库中的微小核糖核酸去调控所证实。令人满意的预测性能表明,GAEMDA模型可以作为一个可靠的工具来指导对微小核糖核酸调节作用的后续研究。
代码:https://github.com/chimianbuhetang/GAEMDA
一、Introduction
微小核糖核酸是一种小的内源性非编码单链核糖核酸分子,长度约为22个核苷酸,可在转录后水平调节基因表达[1,2]。自从最初的两个miRNAs lin-4和let-7在秀丽隐杆线虫中被发现以来,已经过去了二十多年[3,4]。在此期间,越来越多的研究分析表明,微小核糖核酸在各种复杂的生物过程中起着关键作用,如细胞增殖、分化、信号转导、病毒感染等[5]。此外,新出现的实验证据也表明,微小核糖核酸的突变或异常表达经常导致许多复杂的人类疾病的进化和进展[6]。例如,已证实hsa-mir-449a在CL1-0中的过度表达将增加辐射诱导的DNA损伤和凋亡,改变细胞周期的分布,并最终导致CL1-0对辐射的敏感性[7]。此外,hsa-mir-195和hsa-mir-497被证实在乳腺癌恶性肿瘤中起关键抑制作用,甚至可以成为潜在的诊断靶点[8]。因此,采用适当的实验或计算方法探索微小核糖核酸与疾病之间的联系,可以使微小核糖核酸作为肿瘤抑制剂或生物标志物,帮助医务人员从分子角度深入了解各种复杂疾病的病理机制,开发相关新药[9]。
用于鉴定潜在miRNA-疾病关联的传统实验方法主要包括逆转录聚合酶链反应[10],northern印迹[11]和微阵列分析[12]。一般来说,实验方法往往效率低下,需要大量的时间和金钱投入。然而,由于实验方法的可靠性,研究人员已经建立了许多权威的生物信息学数据库来存储实验证实的微小核糖核酸-疾病关联,例如人类癌症中差异表达的微小核糖核酸数据库(dbDEMC) [13]、人类微小核糖核酸疾病数据库(HMDD) [14]和人类疾病中微小核糖核酸去调控数据库(MiR 2 dSease)[15]。同时,计算机的计算和存储性能也有了很大的提高。因此,设计优秀的计算方法来预测潜在的微小核糖核酸-疾病关联成为可能。计算方法通常是有效和经济的,根据它们对特定疾病的影响,可能对微小核糖核酸进行优先排序。因此,计算方法可以为研究人员提供一个新的视角来研究排名靠前的微小核糖核酸,并促使他们进行相关的实验方法来进一步验证这些联系。
在过去的十年中,已经提出了许多潜在的微小核糖核酸-疾病关联预测的计算方法。在这些方法中,基于相似性度量的方法是一种经典的计算方法,它基于具有相似功能的微小核糖核酸往往与具有表型相似性的疾病相关的假设,对疾病相关的微小核糖核酸进行优先排序[16]。例如,姜等人[17]首先开发了一种基于人类现象-微脑网络的计算方法,以检测功能相关的微小核糖核酸与表型相似疾病之间的关系。然而,由于miRNA-靶关联中存在高假阳性和假阴性率,该方法的预测性能受到限制。后来,陈等人[18]将高斯相互作用谱核相似性集成到miRNA和疾病相似性的计算中,预测疾病相关的miRNA而不预测任何已知的相关miRNA(WBSMDA)。请注意,WBSMDA方法是一种全局排名方法,它允许同时对所有疾病的微小核糖核酸进行优先排序。Che等人[19]设计了一种基于两个miRNAs序列之间的Levenshtein距离计算MiRNAs功能相似性的新算法,并提出了预测潜在关联的LFEMDA方法。此外,张等[20]发展了预测潜在微小核糖核酸病关联的FLNSNLI方法。该方法首先将微小核糖核酸和疾病表达为关联图谱,然后结合快速线性邻域相似性度量来计算微小核糖核酸和疾病的相似性。最后,FLNSNLI对这两种相似度采用标签传播算法,并利用加权平均策略获得最终的预测得分。马等人[21]通过整合疾病基因信息、miRNA靶基因信息和基因相似性信息等多种数据源,构建了新的miRNA与疾病相似性矩阵,并应用核邻域相似性算法计算miRNA与疾病的核邻域相似性。最后,他们应用双向传播算法来获得预测分数。
基于机器学习的方法是另一种常用的计算方法,用于预测潜在的miRNA-疾病关联。为了预测不同类型的微小核糖核酸与疾病的关联,陈等人[22]利用受限玻尔兹曼机作为分类器,提出了径向基函数神经网络模型。随后,陈等人[23]提出了一种结合基于滤波器的特征选择策略和随机森林分类器的RFMDA方法,以提高预测性能。随后,在RFMDA的启发下,姚等人[24]进一步设计了IRFMDA方法,优化了模型的预测能力。该算法利用一种基于随机森林可变重要度的特征选择策略,采用随机森林回归预测未知关联。此外,严等人[25]还提出了利用逻辑斯蒂矩阵分解和动态邻域正则化计算微小核糖核酸与疾病关联概率的方法。彭等[26]提出了MDA-CNN方法,该方法采用三层网络和自动编码器捕获重要的miRNAdisease特征组合,采用卷积神经网络获得最终预测得分。郑等[27]在k-mer稀疏矩阵的基础上,充分利用miRNA序列信息,开发了MLMDA,并采用随机森林分类器获得预测概率。周等[28]提出了一种新的微小核糖核酸疾病关联预测方法,GBDT-LR,该方法首先对未知微小核糖核酸疾病关联采用k-均值聚类方法筛选阴性样本,然后应用梯度提升决策树挖掘出更多的判别特征,并采用logistic回归模型得到最终的预测得分。同时,通过将miRNA、疾病、lncRNA、药物和蛋白质之间的相互作用整合到一个异构网络中,Ji等人[29]采用具有全局结构信息(GraRep)的学习图表示方法来获得整合的miRNA和疾病的特征,并采用随机森林分类器进行预测。
受图神经网络在图结构数据(例如Cora [30],MovieLens [31],Reddit [32]和蛋白质-蛋白质相互作用(PPI)[33])数据集上取得的巨大进步的影响,许多基于图神经网络的数据 新兴的方法正在解决潜在的miRNA疾病关联的预测。 例如,Li等。 [34]提出了HGCNMDA方法来推断与疾病相关的miRNA,它采用node2vec算法和PPI网络上的图卷积网络来获取miRNA与疾病的交叉特征,并设计了用于潜在关联预测的边缘特征提取组件。 为了获得miRNA和疾病的更多有价值的特征,Li等人。 [35]分别在miRNA相似性网络和疾病相似性网络上进行了图卷积网络,并提出了NIMCGCN方法基于神经归纳矩阵完成生成miRNA-疾病关联。 通过将miRNA相似性和疾病相似性结合成一个完全相连的同质图,Li等人。 [36]提出了一种基于图卷积网络的全连接图卷积网络,用于潜在的miRNA-疾病关联预测。
在本文中,我们提出了一种新的图自动编码器模型GAEMDA,用于潜在的miRNA-疾病关联预测。 具体来说,我们首先构建了一个miRNA-疾病二分图以表达miRNA与疾病之间的关联,其中每个节点均由相应的相似性信息表示,每个连接均表示相应的关联。 其次,考虑到miRNA和疾病节点的异质性,我们设计了节点类型转换矩阵以将miRNA和疾病节点投影到相同的载体空间中。 第三,为了充分探索丰富的miRNA-疾病相互作用信息,我们通过基于图神经网络的编码器将节点的异质邻域特征聚合到其原始特征中,从而生成了节点的嵌入。 第四,将miRNA和疾病节点的嵌入信息输入双线性解码器,以重建miRNA节点和疾病节点之间的联系。 然后,利用交叉熵损失和反向传播算法以端到端的方式训练整个模型。 此外,我们基于5倍交叉验证评估了GAEMDA模型的预测性能。 最终,GAEMDA曲线下平均面积(AUC)为93.56±0.44%,准确度为84.93±0.95%,精度为81.37±1.98%,召回率为90.70±1.27%,F1-得分为85.75±0.76% 。为了进一步验证GAEMDA在预测某些疾病的潜在相关miRNA方面的性能,对结肠肿瘤,食道肿瘤和肾脏肿瘤进行了案例研究。 结果表明,可以通过dbDEMC和miR2Disease分别确认这些肿瘤的前50个预测miRNA中的48个。 我们的模型提供了一个新颖的视角,可以利用现有的miRNA-疾病相互作用信息来解决图神经网络与miRNA-疾病的关联任务。 所有结果表明,GAEMDA可以用作指导miRNA调控作用的以下研究的有力工具。
二、Materials and methods
1.Human miRNA-disease associations
在这项研究中,我们采用HMDD v2.0作为基准数据集,并直接从https://www.cuilab.cn/hmdd [14]下载了经过实验验证的miRNA-疾病关联。 最后,我们可以获得383种疾病和495个miRNA之间的5430个经过实验验证的miRNA疾病关联。 为了方便起见,我们使用了具有383行和495列的二进制矩阵DM来存储关联。 如果疾病与miRNA相关,则在矩阵DM相应位置的元素值设置为1,否则设置为0。请注意,在我们的以下实验中,所有经过实验验证的关联均被选为阳性样品。
2.MiRNA functional similarity
根据Wang等人提供的miRNA功能相似性计算,假设具有相似表型的疾病更可能与功能相似的miRNA相关,反之亦然,我们可以从https://获得miRNA功能相似性www.cuilab.cn/files/images/cuila b / misim.zip [37]。 在这里,我们建立了495行和495列的矩阵MFSM来存储miRNA功能相似性,其中MFSM(mi,mj)表示miRNA mi与mj之间的miRNA功能相似性评分。
3.Disease semantic similarity
基于先前的研究[38],可以基于医学主题词(MeSH)描述符(可在https://www.ncbi.nlm.nihgov /上获得)来计算疾病的语义相似度。在这里,我们将每种疾病制定为有向无环图(DAG)。 具体来说,我们可以采用DAG(di)=(di,T(di),E(di))来描述疾病di,其中T(di)代表由节点di及其祖先节点E( di)表示对应的边集,其中包含从父节点到子节点的直接链接。 然后,我们可以计算出疾病d对di的语义贡献如下:
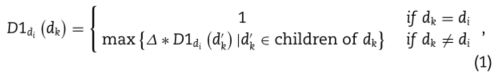
其中∆表示语义贡献衰减因子,根据先前的研究,我们将其设置为0.5 [37]。 疾病di本身的语义贡献值为1,疾病dk疾病di的语义贡献值将随着它们之间的距离增加而减小。 因此,疾病的语义值可以定义如下:

基于以下假设:如果两种疾病共享其DAG的较大部分,则可以认为它们更相似,我们可以得出疾病di和dj之间的疾病语义相似度DSSM1(di,dj)如下:

其中DSSM1是一个383×383矩阵,用于存储第一类疾病的语义相似性。
但是,考虑到出现在更多DAG中的疾病可能更常见,出现在更少DAG中的疾病可能更具体,因此DAG同一层中疾病的语义贡献值应该有所不同。 因此,我们采用了另一个在先前研究的基础上计算疾病语义相似度的方法[39]。 在这里,疾病dk对 di的语义贡献可以描述如下:

相应地,我们可以从等式(5)获得疾病di的语义值,并从等式(6)获得疾病di和dj之间的疾病语义相似性DSSM2(di,dj)。
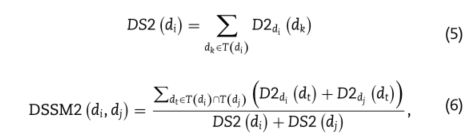
其中DSSM2是一个383×383矩阵,用于存储第二种疾病的语义相似性。
为了获得更合理的疾病语义相似度,我们在之前的研究基础上,将这两种疾病的语义相似度进行了合成,以计算最终的疾病语义相似度[18]。 最后,我们可以根据以下等式获得疾病di和dj之间的疾病语义相似度DSSM(di,dj):
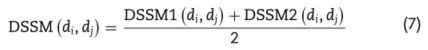
4.Gaussian interaction profile kernel similarity for miRNAs and diseases
基于先前的研究,可以通过一个假设来计算高斯相互作用谱的核相似性,即一个类似的miRNA更可能与相似的疾病相关[18]。 具体地,构建位于矩阵DM的第i列的二元载体IP(mi)以代表miRNA mi和每种疾病之间的关联。 然后,可以如下计算miRNA mi和mj之间的miRNA MGSM(mi,mj)的高斯相互作用分布图内核相似度:
![]()
其中rm是用于控制内核的带宽。 在这里,我们可以根据以下规范化原始内核带宽r’m来计算rm:

其中nm代表所有miRNA的数量,在我们的研究中等于495,参考先前的研究,r’设置为1 [18]。类似地,疾病di和dj之间疾病DGSM(di,dj)的高斯相互作用分布图内核相似度可以根据以下两个公式计算

其中位于矩阵DM的第i行的二元向量IP(di)表示疾病di与每个miRNA之间的关联,nd表示所有疾病的数量,在我们的研究中等于383,r’d相应地设置为1。
5.Integrated similarity for miRNAs and diseases
考虑到所获得的miRNA功能相似度矩阵和疾病语义相似度矩阵中存在很多稀疏值,我们将高斯相互作用谱核相似度纳入了miRNA和疾病相似度矩阵中。根据Chen的研究[18],计算miRNA mi与mj之间的miRNA IM(mi,mj)的整体相似度为等式(12),计算疾病di与 dj之间的疾病ID(di,dj)的整体相似度为等式(13)。

6.GAEMDA
受图神经网络在链接预测任务[31,40–42]中取得的巨大进步的启发,我们提出了一种图自动编码器模型,该模型将基于图神经网络的编码器和双线性解码器相结合,用于潜在的miRNA-疾病关联预测 (GAEMD)。GAEMDA可以描述为五个步骤(请参见图1):(i)构建miRNA-疾病二分图,(ii)将miRNA和疾病节点投影到相同的向量空间中,(iii)应用基于神经网络的图编码器生成 (iv)应用双线性解码器重建二部图中的链接,(v)应用交叉熵损失函数以端到端的方式训练整个模型。 接下来,我们将讨论每个步骤的具体实现细节。

在第1步中,我们将多个数据源整合到一个miRNAd-isease二分图中,其中包含495个miRNA节点和383个疾病节点。 众所周知,HMDD v2.0中共有5430个经过实验验证的miRNA疾病关联[14]。 在这里,我们将所有这些5430关联视为miRNA节点与疾病节点之间的正例连接。 此外,为了更好地训练模型,我们需要构造相等数量的负例连接来平衡样本集。 考虑到miRNA与疾病之间未知关联的数量远大于已知关联的数量,我们在这里从未知关联中随机选择5430个关联作为负关联,并将它们作为负链接添加到miRNA疾病二分图中。 然后,将所有正例连接标记为1,将所有负例连接标记为0,以进行后续模型训练。 此外,我们将miRNA和疾病的综合相似性分别视为miRNA特征和疾病特征。 具体而言,miRNA mi可描述为495维向量Fmi:
![]()
其中Fmi代表矩阵IM的第i行,vj代表miRNA mi与mj之间的综合相似性值。
类似地,疾病di可被描述为以下383维向量Fdi:
![]()
其中Fdi表示矩阵ID的第i行,wj表示疾病di和dj之间的综合相似性值。 然后,分别在miRNA疾病二分图中将495维miRNA特征Fm添加到miRNA节点,并在383维疾病特征Fd中添加到疾病节点。
在第2步中,我们将异构图中miRNA节点和疾病节点投影到相同的向量空间中。 由于miRNA疾病二分图中节点的异质性,miRNA节点和疾病节点在第1步中属于不同的特征空间。为了便于后续计算,我们设计了节点类型转换矩阵来投影miRNA节点和疾病节点的特征。 进入相同的向量空间 miRNA节点的投影过程可以描述如下:
![]()
其中Hm表示miRNA节点的投影特征,Fm表示miRNA节点的原始特征,W∅m表示将495维miRNA节点投影到E维空间的线性变换矩阵。 同样,疾病节点的投影过程可以如下所示:
![]()
其中Hd表示疾病结点的投影特征,Fd表示疾病结点的原始特征,而W∅表示一个383维疾病节点投影到E维空间的线性变换矩阵。然后,miRNA和疾病节点特征都在E维向量空间中。
在第3步中,我们使用基于图神经网络的编码器生成了miRNA和疾病节点及其直接邻居信息的嵌入。 例如,对于miRNA节点mi,我们首先计算其直接邻居特征的集合,如下所示:
![]()
Ha表示节点mi的邻居特征聚合。 g(·)表示聚合函数,例如sum(·),i.e.element-wise summation of all coming messages, max(·) i.e.element-wise max-pooling of all coming messages, or mean(·) i.e. element-wise mean-pooling of all coming messages. 请注意,sum(·)函数被设置为默认的聚合器。 疾病{d1,d2 ,. 。 。}代表miRNA节点mi的直接邻居。 Dmi是归一化常数,我们选择其为节点mi的度值。 然后,为了将聚合的特征融合到miRNA 节点mi的原始特征中,我们将特征Hmi和Ha mi串联在一起,并应用多层感知器(MLP)来更新节点mi的特征:
![]()
其中H’表示节点mi的更新特征,L表示串联操作,f(·)表示具有E输出的单个MLP层,其等于投影尺寸。LeakyReLU(·)是具有负输入斜率0.2的非线性激活函数。 以类似的方式,我们可以通过以下两个方程式更新疾病节点dj的特征:
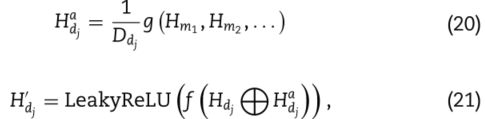
其中Ha dj表示节点dj miRNA {m1,m2,。 。 }是疾病节点dj的直接邻居,Ddj表示节点dj的度值,而H’dj表示节点dj的更新特征。 注意,上述操作可以同时应用于所有miRNA和疾病节点。我们将这些操作称为基于单层图神经网络的编码器。 考虑到这种基于单层图神经网络的编码器的输入和输出均为E维向量,我们可以堆叠L层基于图神经网络的编码器以聚合多个邻居的特征并增强miRNA的特征表示能力 和疾病节点。 因此,我们可以获得miRNA节点的最终嵌入HL疾病节点的嵌入和 通过L层图神经网络为基础的编码器的HLd。
在第4步中,我们采用了双线性解码器来重建miRNA与疾病节点之间的联系。 由于S形激活函数在处理二值化分类问题方面具有很大的优势,因此我们在此引入双线性运算,然后再使用S形函数来预测将miRNA节点mi与疾病节点dj链接的概率yij:
![]()
其中Q是具有E×E维的可训练参数矩阵,并且Sigmoid函数可以定义为sigmoid(x)= 1 /(1 + ex)。
在第5步中,我们对所有训练样本应用了交叉熵损失LOSS来优化模型参数,如下所示:

其中yij表示链接的真实标签,将为1或0,Y和Y-分别表示包含在正链接集和负链接集中的所有节点的集合。 然后,我们可以通过反向传播算法以端到端的方式训练整个模型。
三、Results
1.Implementation details and evaluation metrics
我们基于带有MXNet后端的Deep Graph Library实现了GAEMDA模型[43]。 在训练阶段,我们使用Xavier初始化[44]随机初始化模型参数,使用Adam [45]优化模型参数。 此外,我们通过网格搜索找到最佳的超参数,并将学习速率设置为0.001,权重衰减设置为1e-3。 为了避免过度拟合问题,我们在投影操作和每个MLP层之后随机删除了隐藏单元。 我们从{0.1、0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9}中搜索了最佳辍学率,并在实验中将其设置为0.7。 我们将模型训练了1000个时期,并每10个时期打印出测试集结果。 所有样本均基于HMDD v2.0构建[14]。 该实验在Nvidia Tesla P100 Cluster中进行。 所有代码和数据集都可以在https://github.com/chimianbuhetang/ GAEMDA。
为了确保公正的比较,使用5倍交叉验证来评估GAEMDA的性能。 特别地,将所有样本分为五个相等的部分,将每个部分视为测试集,将其他四个部分依次作为训练集。 因此,训练集和测试集之间不会有重叠,并且每个样本都可以通过我们的模型在进行5倍交叉验证后进行测试。 此外,采用了四个常用的评估指标来衡量GAEMAD的性能,即准确性(Acc。),精度(Prec。),召回率和F1得分。 同时,我们绘制了接收机工作特性(ROC)曲线以直观地显示我们模型的性能,并利用AUC来全面评估该模型的性能。 通常,较高的AUC值表示较好的预测性能,而低于0.5的AUC值表示随机分类能力。
2.Performance evaluation
3.Effect of projection dimensions
4.Effect of the number of encoder layers
5.Comparison of GAEMDA with other related models
6.Case studies
四、Discussion
通常,异源miRNA和疾病特征属于不同的维数空间。并且,很难对不同的尺寸特征执行操作。因此,我们将异源miRNA和疾病特征投影到相同的载体空间中。 然后,可以在相同向量空间中计算异构特征。 同时,考虑到图神经网络可以有效地聚合图中的相邻特征并增强节点的特征表示,我们设计了一种基于图神经网络的编码器,以探索丰富的miRNA-疾病相互作用信息,并生成有效的miRNA和疾病节点嵌入 。然后,应用双线性解码器,然后加上S型激活函数,以获得miRNA-疾病关联评分。此外,我们的模型采用的端到端训练模式可以根据特定的链接预测任务更好地预测潜在的miRNA-疾病关联。 但是,由于图神经网络的过度平滑问题,我们的编码器模型很难扩展到很深的层。
五、Conclusion
在许多复杂的人类疾病的发展过程中,已经广泛观察到异常的miRNA表达。 疾病相关miRNA的鉴定可以促进相关疾病的病理学研究,促进临床医学的发展。 在本文中,我们设计了一种新颖的图自动编码器模型来预测miRNA与疾病之间的潜在关联,称为GAEMDA。 GAEMDA采用基于图神经网络的编码器生成miRNA和疾病特征的嵌入,然后应用双线性解码器重建miRNA与疾病之间的联系。此外,在5种交叉验证下的几种评估指标以及对三种常见复杂疾病的案例研究均证明了GAEMDA的令人满意的预测性能.因此,GAEMDA可作为指导研究人员研究相关miRNA调控作用的有力工具。 但是,考虑到miRNA与疾病之间已验证的关联比例很小,并且我们的模型严重依赖于异构节点之间的直接邻居信息,因此我们计划寻求方法来扩展聚合信息的范围,例如聚合多种信息。 多 -跳邻居,以进一步增强预测性能。 此外,嵌入更多的生物学信息(例如miRNA序列或靶标信息)以及设计更有效的聚合器(例如注意力机制)也是我们接下来的研究重点。
个人总结
1.该文章提到图神经网络自动编码器,但是本文是先找到节点的邻居信息,然后使用MLP这种方式来聚合邻居信息,感觉并不能算严格意义上的图神经网络。
2.没有什么好总结的了。。。。。。