Nature:用2斤DNA就能存储世界上所有的数据
◆ ◆ ◆
前言
现代存储技术已经无法满足字节的海啸式增长,但是大自然也许已为这个难题提供了解决方案。
对尼克•高德曼(Nick Goldman)而言,用DNA来编码数据始于一个玩笑。
那是2011年的2月16日,星期三。高德曼正在德国汉堡的一个酒店里,与几个生物信息学家讨论如何解决铺天盖地而来的海量基因组序列以及其他数据的存储难题。他记得科学家们差不多要被传统计算技术的所耗费用和局限性难倒时,他们开始对其他方案开起了玩笑。“我们想,会有什么能阻止我们用DNA来存储信息呢?”
这时,笑声停下来。“那是灵光一现的时刻。”高德曼说。那时,他是位于英国欣克斯顿的欧洲生物信息研究所(European Bioinformatics Institute (EBI))的小组负责人。的确,与硅质存储芯片的毫秒级读写速度相比,DNA存储可能慢的可怜。碱基通过特定模式合成DNA链要花数小时,而通过测序来解读信息又要花费数小时。但是用DNA,人类全基因组都可以存储到一个肉眼所不能见的细胞内。仅从信息的存储密度而言,DNA是大于硅的几个数量级倍。因此对于长期存储,DNA能带来更高的存储密度。
高德曼介绍说:我们在酒吧里坐下来,就用餐巾纸和圆珠笔,开始勾画“如何做才能实现这个想法?”。研究人员最大的顾虑是DNA的合成和排序的过程中核甘酸通常会发生1%的错误,这使大规模的存储信息变得不可信赖,除非他们能找到一个可实施的纠错体系。他们通宵讨论了是否能找到一个方法,把信息编码到碱基对里,并同时允许他们检测和纠错。
最后,高德曼说“我们知道可以做到”。然后,他和他的EBI同事伊万•伯尼(Ewan Birney)带着这个想法回到了实验室。两年后他们宣布成功地用DNA编码了5个文件,包括莎士比亚的十四行诗和马丁•路德•金“我有一个梦想”的演讲片段。那时,哈佛大学的生物学家乔治•丘奇(George Church)和他的团队也发表了一种DNA编码的实例。
当时739KB数据量的EBI文件构成了有史以来的最大DNA存储;但是在2016年7月,微软和华盛顿大学的研究人员宣布,利用DNA存储技术完成了约200MB数据的保存,是信息存储的一个飞跃。
目前,全球范围内都面临着数据量的快速增长。最近的实验有迹象表明,对使用DNA作为存储介质的兴趣已经远远超过了基因组领域。在2020年之前,从天文图像、杂志文章到YouTube视频,全球的数据存储量预计将达到44兆GB,相比2013年将增长10倍。到2040年,如果所有的信息都使用即时存储,比如像U盘那样的闪存芯片,存储耗费的微芯片级硅为预期供应量的10-100倍,这就是目前为什么数据的永久存储罕用老式磁带的原因之一。
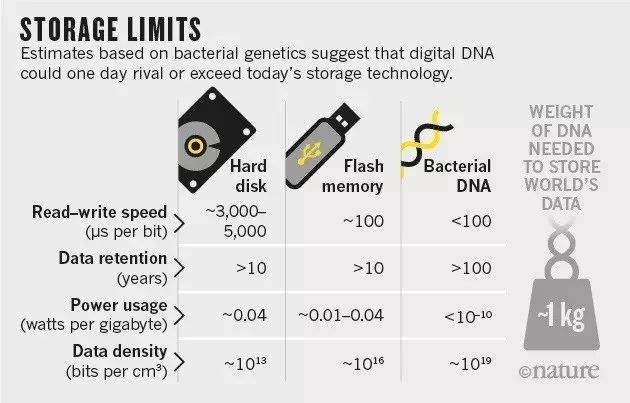
这种介质对信息的封装比硅紧密很多,但是读起来较慢,然而即使这种方法也正在变得不可持续。大卫•马科维茨(David Markowitz)是位于美国华盛顿特区高级智慧研究计划(IntelligenceAdvanced Research Projects Activity (IARPA))的一名计算神经学家。他说,可以想象一个数据中心有一个艾(exabyte,等于十亿个G)的数据存储在磁带上,但是这样一个中心需要十亿美元和十年以上的时间来建造和维护,以及上亿度电的耗费,而分子数据存储器可以将所有这些要求降低3个数量级。如果信息封装密度像大肠杆菌的基因那么高,全世界的存储需求可用1千克的DNA来满足。(详见“存储限制”)
达到这个目的并非容易,在DNA成为传统存储技术的有力竞争对手之前,研究人员还有许多挑战,从把信息可靠地编码到DNA中、解码某个用户想要的特定信息,到能够更加便宜、快捷地得到核苷酸排序,克服这些挑战需要付出巨大努力。半导体研究委员会(The Semiconductor Research Corporation (SRC))是由芯片制造商财团赞助的基金会(位于北卡罗来纳州达勒姆市),正在为DNA存储工作提供支持。高德曼和伯尼得到了英国政府的资助,利用下一代(next-generation)方法进行DNA存储的试验,并在此研究的基础上计划设立公司。高级智慧研究计划(IARPA)和半导体研究委员会(以下简称SRC)在4月举办了面向学术界和工业界(如IBM)研究人员的研讨会,来指导该领域的研究。SRC主任兼首席科学家维克多•哲诺夫(Victor Zhirnov)提到,十年来我们一直在寻找硅以外的数据存储方法,而DNA被认为有可能是替代磁带的最佳候选之一。
◆ ◆ ◆
长期存储器
1988年,艺术家乔•戴维斯与哈佛的学者合作,第一个将数字信号0和1对应到DNA的四个碱基。他们把DNA序列插入到大肠杆菌里,仅仅编码了35个字节。当排列成一个5*7的矩阵时,1对应到暗像素,0对应到亮像素,它们组成了一幅古代日耳曼如尼字母图画,代表生命和女性的地球。
现在戴维斯已经加入了丘奇的实验室,该实验室2011年起开始探索DNA数据存储。哈佛团队希望该应用可以减少合成DNA的高成本,就像基因组学的测序成本已经降低了许多。丘奇与加州大学洛杉矶分校的瑟里• 库苏里(Sri Kosuri)以及约翰•霍普金斯大学的基因组专家高原(Yuan Gao)于2011年11月实施了概念证明性实验。团队使用了很多短DNA片段编码了一本丘奇与他人合写的659KB数据的书。每个片段的一部分用来进行排序后片段组装顺序,剩余部分用于编码数据。将数据保存在DNA之中需要将二进制0和1数据转换为4种核苷酸,其中0用腺苷酸或胞嘧啶来编码,而1则用鸟苷酸或胸腺嘧啶。这种灵活性帮助团队设计序列,避免测序中高GC区读取错误、重复序列或发卡结构导致的绑定彼此的片段发生序列折叠。他们没有做严谨的纠错,而是依靠每个片段拥有多个拷贝的信息冗余。结果对片段测序后,他们发现了22个错误,大大高于可靠存储的要求。
同时在EBI,高德曼、伯尼和他们的同事也在使用很多DNA片段来编码一个739KB的数据存储,包含一个图片、ASCII文本、声音文件和一个PDF版的华生和克里克标志性的双螺旋结构。为了避免重复碱基和其他来源的错误,EBI领导的这个团队使用了一个更加复杂的系统(见“制作存储体”)。一方面是将0和1组成的二进制数据编码修改成以3个数为基础,即0,1和2,然后持续地轮换使用每一个数的代表,因此而避免在读取数据时序列可能出现的问题。通过利用序列重叠,100个碱基长度的片段持续位移25个碱基,EBI的科学家们确保有4个版本的片段来做错误检查和互相比较。
但是他们仍然丢失了25个碱基序列中的2个,具有讽刺的是,恰恰是华生和克里克的文件部分。尽管如此,高德曼确信这些结果证实了DNA极有潜力成为一种廉价的长期存储介质且耗能量较小。究竟能存储多长时间,高德曼例举了2013年公布的在700,000年前陷入冻土中一匹马的骨化石被解码的基因组,他解释说“在数据中心,没有人相信一个三年之久的硬盘,或是一张十年之久的磁带的可用性。如果你需要一个更安全的长寿命拷贝,那么当我们能把数据都保存在DNA中,你就可以把它放到一个山洞里,直到你想读取的那一天。”
◆ ◆ ◆
一个迅速增长的领域
自从华盛顿大学计算机科学家路易斯(Luis Ceze)和在华盛顿州雷德蒙德市微软研究院工作的计算机科学家施特劳斯(KarinStrauss)在2013年访问英国期间,听到Goldman讨论EBI工作之后,那种可能性就激发了他们的想象力。施特劳斯说:“DNA的密度,稳定性和成熟足以让我们对它感到兴奋”。
当他们返回华盛顿州时,施特劳斯和路易斯就与他们在华盛顿大学的同事格奥尔格(Georg Seelig)一同展开了调查。他们的主要关注点之一是DNA面对错误的脆弱性,这已经成为了DNA存储的一个主要缺陷。应用标准的排序方法,只能恢复任意一段数据而不能恢复全部数据,这意味着每一条DNA链都要被读取。这将会极大地增加传统计算机内存的负担,因为计算机内存允许随意接入的能力使得用户能够只读取他所需要的数据。
在四月初乔治亚州亚特兰大市召开的会议上,这个团队概述了他们的解决方案。研究人员们先从DNA存档中取出很小的样本,随后应用聚合酶链反应(PCR)技术去标记和复制那些将他们想要提取的数据进行编码的链条。与先前的方法相比,复制品的增殖使得排序变得更快,更廉价和更精确。此外,这个团队还设计了一个可供替代的错误修正方案,此方案在可靠性上允许对数据进行同EBI方法一样密集的二次编码。微软和华盛顿大学的研究人员们存储了151kB大小的图片作为样品示范:在一个单一链条池中,一些采取EBI方法进行编码,另一些采取他们的新方案进行编码。他们应用类似EBI的方法提取了三种事物,一只猫、悉尼歌剧院和一个卡通猴子,结果得到了一个读取错误,他们不得不通过手工来修复。而应用他们的新方法读取悉尼歌剧院图片时,没有任何错误报出。
◆ ◆ ◆
经济VS化学
伊利诺伊香槟分校的计算机科学家奥尔吉察( Olgica Milenkovic)和她的同事已经研究出一种随机接入的方法,这使他们有能力重写被编码的数据。该方法是将数据存储为DNA长链,这些长链在两端都有位置信息序列。研究人员随后使用PCR方法或者CRISPR–Cas9基因编辑方法,通过这些位置选择,增强和重写链条。这些位置信息旨在消除阻碍读取的序列,同时体现出相互之间的差异以阻止它们被混入错误中。要做到这一点需要进行大量的计算,同时规避诸如分子折叠(因为它们的序列包含可以识别和绑定彼此的片段)等问题。奥尔吉察说:“最开始,我们使用的是计算机搜索,因为很难想到其他拥有这些特性的方法。现在她的团队使用数学公式替代了这种高消耗劳力的方法,使得他们能够设计出更快速的编码方案。
卡苏瑞(Kosuri)提出,其他有关DNA存储的挑战包括规模化和合成分子的速度,这也是他为什么不太看好这种想法的原因。他回忆说:在哈佛的早期实验中,我们有700kB数据量,然后仅是1000倍的增长就达到700MB,就像CD。他认为,真正能够对世界性数据归档问题做出改变意味着至少可以存储千万亿字节的信息,这是可行的。但是人们要意识到规模大约要提高数百万倍。马科维茨(Markowitz)同意那不是件容易的事。他说:“优良的生产方法几乎需要30年的化学过程,即每增加一个碱基将花费400秒的时间。如果持续使用这种方法,数以十亿计的不同的链条需要并行地制作以实现快速写入,而现在最大的并行产出量仅是数以万计的链条”。
一个密切相关的影响因素是合成DNA的成本,它的花费占据了EBI实验全部12,660美金的98%,而排序仅占到了2%。而这还要感谢在2003年人类基因组计划完成后实现的两百万倍的成本减少。尽管这样,卡苏瑞(Kosuri)对DNA合成领域可以达到同样的经济效能并没有信心。他说:“你可以轻易地想像,市场可以测序70亿人,但不可能建立70亿人的基因组”。他认可丘奇(Church)和其他人在六月提议的人类基因组编写计划(HGP-write)的费用成本上会有一些突破。如果项目被资助,它将致力于合成全部的人类基因组:23对染色体,包含32亿核苷酸。但是,即使HGP-write成功了,卡苏瑞认为一个人类基因组仅仅包含0.75GB的信息,而且这个数量还会因合成实际数据存储所带来的挑战而变得更小。然而,日尔诺夫(Zhirnov)对此保持乐观,他认为合成的成本将低于目前的水平达数量级,“没有理由相信其为何如此之高?”。
在四月,微软的研究人员迈出了第一步,他们与一家来自加州旧金山的DNA合成初创公司订购了一千万链条,这可能帮助创始一个必要的需求。施特劳斯(Strauss)和她的同事相信,他们已经使用这些链条推动随机接入存储方法到0.2GB。细节还没有被发表,但是据报道,存档包含了用超过100种语言记录的世界人权宣言,古登堡计划的前100本书和一个种子数据库。尽管这跟HGP-write所面临的挑战相比还微不足道,但是施特劳斯强调了在存储容量上跳跃250倍的重要性。她说:“是时候训练我们处理更大数量的DNA,推动它形成更大规模,并且找出这个过程中的瓶颈。它确实在很多地方都有中断,而且我们正在努力学习去解决”。
高德曼(Goldman)坚信这只是将要面临的难题之一并对此具有信心。他说:“我们的评估是需要十万倍的提升来实现这一技术,而这是可行的。尽管以过去经验并不能确保,但每一两年就有很多新的读取技术在运作。在基因组学,六个数量级并不是难事,你只需要稍微等待”。
原文发布时间为:2016-09-07
本文来自云栖社区合作伙伴“大数据文摘”,了解相关信息可以关注“BigDataDigest”微信公众号