无论是Windows,MacOS, Linux桌面的用户,你都可以也会使用。
写在前面
伴随基因组测序的普及,越来越多物种的基因组测序项目开展并报道,为科研工作者提供了更为全面的参考序列信息。目前,单纯植物类已公布的基因组,应在400~500个物种之间。这带来了更多的数据,也带来了更多的信息分析挑战。物种基因组公布,往往(至少)会公布两个文件:
- 物种基因组序列文件,如 genome.fa
- 物种序列特征注释文件【或更常见的 基因结构注释信息文件】,如 genome.gff3 / genome.gtf
对于具有编程基础或熟悉 Linux 操作系统的数据分析人员,往往可以通过编写脚本或使用诸如 gffreads 等命令行工具来进行序列提取【如:所有基因的 CDS 序列】。但对于可以从数据中受益的更多人而言,这并不是一个完美选项。
三年前,TBtools 释放了 ** GTF/GFF3 Sequences Extract** 功能,应为极少见的【至少我没见过其他】 可实现基于 GTF/GFF3 注释信息 从全基因组序列中提取 指定特征序列集合 的GUI工具。
【注,其中 Start 摁钮为灰色,即不可用,下文有说明】
常见用途
- 提取所有 CDS 序列集合(可用 TBtools 直接翻译成 蛋白序列集合)
- 提取所有 Exon 序列集合(即注释文件中所有转录本序列)
- 提取所有 Gene 序列结合(即基因的所有Exon和Intron序列)
- 提取所有 Promoter 序列集合(或为 UTR及上游1~2kb;或为 ATG及上游1~2kb等)
TBtools 提取出的序列可以保证 ID 完全统一,方便后续任何分析【如基因结构可视化,基因定位在染色体上等等】。当然,TBtools 也提供了 GTF/GFF3 信息整理工具,可方便用于直接整理 ID 重命名的映射文件,随后可用 Fasta Rename 直接进行批量重命名。
以下,大体介绍下两个常见用途(提取 CDS 或 提取 Promoter)的操作姿势
使用到的文件
【注:如果文件是.gz .rar .tar.gz 等后缀的压缩文件,请先解压缩再使用】
使用第一步
GFF3/GTF文件中存储的序列特征很多,对于 GUI 操作,用户往往需要全面了解文件中可供提取的序列特征,如CDS, Exon等。所以使用这一功能的第一步,即 a) 导入GFF3/GTF文件 b) 点击初始化
可以看到,点击初始化之后,Feature ID等均有了变化
同时还会弹出一个文本对话框,这个对话框,事实上用户辅助用户选择 Feature Tag 和 Feature ID。
- Feature Tag,一般是 CDS, Exon, mRNA, Transcript, Gene等,用户往往已经知道自己要提取什么,不需要辅助
- Feature ID,以 CDS 和mRNA为例。我们很清楚,真核生物中,大多数基因均是断裂基因,即 Exon-Intron-Exon... CDS即Exon的部分,所以CDS本身在基因序列的尺度上,也是被 Intron 所分隔。当我们需要提取 CDS 的时候,往往我们说的并不是提取 一截CDS,而是把某个转录本对应的几截甚至几十截 CDS 按照正确的顺序和方向连接起来之后的真实的 CDS 全长。于是 ,如何连接,这个时候,就需要用户指定一个 ID,如下,
可以看到 GFF3 注释文件中,同一个转录本 ATG01010.1 拥有几截CDS。这些 CDS 可以以 Parent 这个 Feature ID 来归组,并组合成 ATG01010.1 的 CDS 全长。事实上,这个文件比较简单,因为 CDS 并没有更多的 Feature ID 可以选择。在其他物种中,常常会出现各种各种的 Feature ID,比如直接是 ID=。
可以看到图片中还有 mRNA,在这个注释文件中,每一个mRNA基友 ID= 也有 Parent=,前者事实上是这个 mRNA 独有的标签,后者则记录了这个 mRNA 属于是哪一个基因的转录本之一。此时,如果是要提取这个mRNA的序列【包括Exon和Intron】,那么设置 Feature ID 为 ID 则是正确的。如果选择 Feature ID 为 Parent,则只会得到不理想的结果。一个基因多个转录本会有完全相同的 Parent 便签,这些转录本会按顺序串联起来,这并不没有生物学意义。
于是,可以开始不同的提取操作了,但是在开始之前,还有一件事情一定不能忘记。
注意事项
TBtools 所有 GUI 功能的设计与使用遵从一个基本逻辑:set [I]nput files,set [O]utput files if needed,click [S]tart button,简称 IOS。
所以,下述任何操作,即点击 Start 之前,先设置输出文件。
提取物种所有 CDS 序列
【注意,必须先设置 输出文件】
很快,可以看到输出文件。其中包括了物种【注释文件中包括】的所有CDS序列全长。
如果需要 蛋白序列 集合,那么只需要直接用 TBtools 的 批量翻译功能即可。
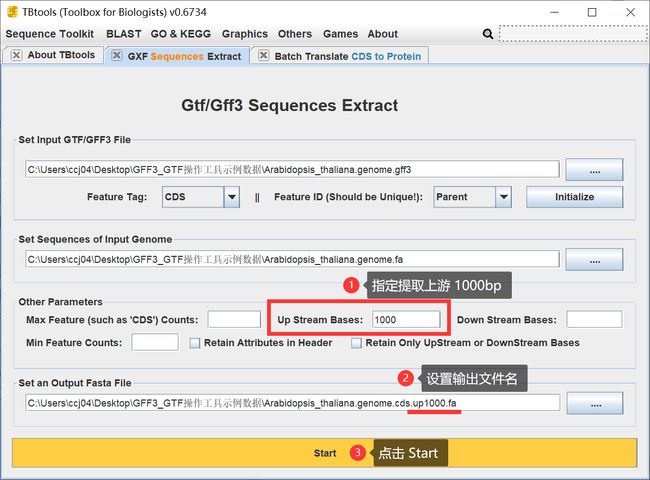
提取所有转录本的 Promoter 序列
Promoter 往往指 UTR上游1~2kb 的区间,这并不绝对,因为顺式作用元件同样可以存在于 Intron 甚至是 Exon 上。目前,大多数物种的基因结构注释信息并不完善,在常见的数据分析中,除少数模式生物,如拟南芥,水稻,人类等,在提及对 Promoter 序列的分析,往往直接使用 翻译起始密码子ATG上游1~2kb的序列。此处,也以同样的方式处理【注,这并不是我本人的生物学认定,仅为演示用】。
于是可以得到,
于是,在所有 CDS 全长的前面,增加了 1000bp 的序列,即上述提及的 Promoter 序列。
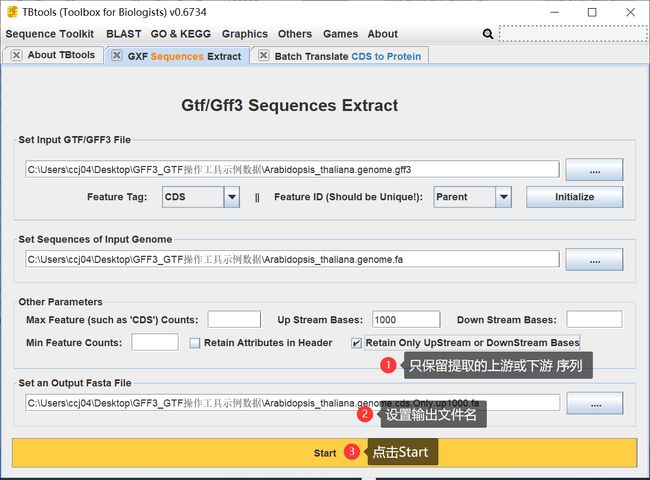
很多时候,大家需要的是不包含CDS序列的,纯粹的 Promoter 序列,这个时候,只需要勾选多一个选项即可,
于是得到
于是,对于其他序列特征,如Exon全长,miRNA前体等等,参考以上就可以了。
其他参数详解
这个功能基本完善于两三年前,但是参数还是比较丰富。
其中:
- 仅提取不多于指定Feature数目的序列,比如,有时候我们只需要 单外显子 转录本的,那么这个时候,指定最多允许1个Exon即可。
- 仅提取不少于指定Feature数据的序列,与1. 正好相反
- 保留注释信息在输出的 Fasta 文件 ID,中,有时候我们希望得到信息相对全面的文件
- 提取 Feature 5’ 端上游指定长度的序列,如上述的 CDS 上游1kb
- 提取 Feature 3’ 端下游指定长度的序列
- 只保留上游或下游序列
补充
当然,你可能还有各式各样的需求。
你只是需要部分基因的Promoter,那就用 TBtools 的 Fasta Extract,
你想要提取GFF3中每个转录本对应的基因ID,
你又想要批量对提取之后的序列进行重命名
你还要想什么?用好搜索框!不用拼命翻菜单。
写在后面
昨天半夜醒来,毫无困意。推文写得七七八八,加上前述也听闻 follower 要在他们的软件里面实现类似的功能,那么索性推出,方便作为他们的参考。整体上,这个功能自从推出后,几乎没有更新过。不过我个人的使用频次较高,主要原因在于,越多物种的基因组数据要分析,越多项目需要开展,那么 ID 的一致和统一就越显得重要,而 TBtools 在这块上从设计之初就很在意。
有时候,我们更多地应该从程序解析或者机械自动化的角度去思考问题,设计和实现功能;而不是纯粹考虑输出结果如何迎合人类偏好。因为,人的欲望是复杂的,甚至是无规律可循。于是,剩下的,就只有断层。
当然,这也是为什么,很多类似生信小工具很少人愿意去写,比如 TBtools 的 Fasta 序列操作(包括提取,重命名,格式化等等)。为什么在 TBtools 之前没有轻便的工具【事实上,除去JRE和一些Wrapper程序,TBtools的整体大小只有几Mb】?
其实道理就这么简单,因为简单而且繁杂的事情,做数据分析/软件开发方向的绝大多数人,并不在意或者并不愿意,因为这看起来并没什么技术含量,就跟我现在一直在做的多数工作一样。
不过,有没有技术含量,跟值不值得,喜不喜欢,似乎并没有关系。市面上,又有多少是有技术含量的呢?如果你认真起来。