首先,为什么要用R呢?我想用paper上同样的方法定量细胞运动轨迹,但是Chemotaxis Tool插件无法导入Manual tracking导出的data,应该是ImageJ的版本太新,导致两个插件不能兼容,但是尝试降级发现只能降到1.49,也不管用,所以开始想用别的软件试试。Cellprofiler安装后打开有问题,怪怪的。Imaris要收费,找了两次学校Imaging部门的人员想学这个软件,结果也是找不到人。但是呢,周一要跟老板talk,周二要讲组会,所以不管怎么样得想办法做个定量的图出来。
经过几天尝试各种软件,我发现其实大部分都是能做tracking,不管是auto还是manual,所以目前的问题就是怎么用导出的数据做xy折线图,而且要在一张图上显示多个轨迹。按理说excel应该就可以,但是我总觉得excel的图丑,然后查着查着发现R可能是个选择。
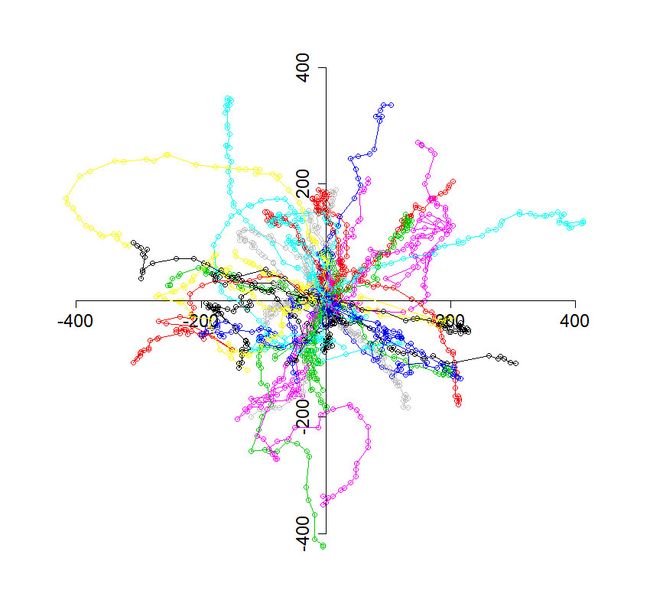
看过的几页R语言几乎没用处,所以上网搜教程,下软件,敲别人的代码研究研究,终于在周六的上午,搞出了跟理想中很接近的图,唯一不足的是我想只在轨迹终点显示dot,以体现运动方向,这个目前还没弄出来。
ok,代码如下:
> hui <- read.csv(file.choose())
> hui2 <- as.tracks(hui, id.column = 1, time.column = 2, pos.columns = c(3, 4))
> plot(hui2, xlim = c(-500, 500), ylim = c(-500, 500), pos = 0, cex.axis = 1.5, cex = 1)
哦对了,要先安装并且加载MotilityLab包,否则as.tracks不能用。尽管这几行代码运行过程中会有warnings,但是还是能运行出结果的。
当当当当~~
嗯..仔细看看还是有问题的,颜色不够多,好像有共用一个颜色的轨迹,先看个趋势吧,下周去学软件,R嘛有机会再学一学,还是很强大的,四行代码就画出来了其实。