偶然发现有个R包可以做相关的QTL 分析。拿来试试。
首先 安装,参考官方文档https://github.com/bmansfeld/QTLseqr.
第一步,如果没有安装devtools的话,install.packages("devtools"),有就直接调用,如果这个不会,就不用往下看了。
第二步,devtools::install_github("bmansfeld/QTLseqr")
从github加载QTLseqr。
拿他的论文实例来演练一次。
library("QTLseqr")
看一下输入文件,文档中说不仅可以识别GATK中VariantsToTable 功能下的table格式的文件,也能识别包含每个批量的等位基因读取深度的分隔文件。调用的功能是importFromGATK and mportFromTable。
我们先从GitHub下载一个Yang2013data 。
命令如下:devtools:: install_github("bmansfeld/Yang2013data")
library("Yang2013data")
导入数据
rawData <-system.file(
"extdata",
"Yang_et_al_2013.table",
package ="Yang2013data",
mustWork =TRUE)
如果自己有数据table格式:
rawdata<-(file_to_path/my_table_rawData <-system.file(
"extdata",
"Yang_et_al_2013.table",
package ="Yang2013data",
mustWork =TRUE)
如果自己有数据
rawdata<-(file_to_path/my_table)
然后我们分别给高低池命名。写染色体编号。
HighBulk <-"SRR834931"
LowBulk <-"SRR834927"
Chroms <-paste0(rep("Chr",12),1: 12
用GATK call snp。table文件用线面这个命令转换。
java -jar GenomeAnalysisTK.jar \
-T VariantsToTable \
-R ${REF} \
-V ${NAME} \
-F CHROM -F POS -F REF -F ALT \
-GF AD -GF DP -GF GQ -GF PL \
-o ${NAME}.table
看看ImportFromGATK 功能
df <-
importFromGATK(
file =rawData,
highBulk =HighBulk,
lowBulk =LowBulk,
chromList =Chroms
)
会计算两个池的等位基因频率,snp-index,还有Δindex。
导入一下分隔文件。可以是csv, tsv或其他格式。格式表头如下。
如果有现成的table文件,也可以importFromTable
df <-importFromTable(file ="Yang2013.csv",
highBulk =HighBulk,lowBulk =LowBulk,
chromList =Chroms)
矩阵如下:## CHROM POS REF ALT AD_REF.LOW AD_ALT.LOW DP.LOW GQ.LOW PL.LOW
## 1 Chr1 31071 A G 34 36 70 99 897,0,855
## 2 Chr1 31478 C T 34 52 86 99 1363,0,844
## 3 Chr1 33667 A G 20 48 68 99 1331,0,438
## 4 Chr1 34057 C T 38 40 78 99 1059,0,996
## 5 Chr1 35239 A C 25 36 61 99 987,0,630
## 6 Chr1 38389 T C 36 42 78 99 1066,0,906
## SNPindex.LOW AD_REF.HIGH AD_ALT.HIGH DP.HIGH GQ.HIGH PL.HIGH
## 1 0.5142857 26 22 48 99 522,0,698
## 2 0.6046512 40 34 74 99 848,0,1099
## 3 0.7058824 24 29 53 99 765,0,599
## 4 0.5128205 29 26 55 99 673,0,772
## 5 0.5901639 40 60 100 99 1632,0,1015
## 6 0.5384615 42 40 82 99 984,0,1105
## SNPindex.HIGH REF_FRQ deltaSNP
## 1 0.4583333 0.5084746 -0.055952381
## 2 0.4594595 0.4625000 -0.145191703
## 3 0.5471698 0.3636364 -0.158712542
## 4 0.4727273 0.5037594 -0.040093240
## 5 0.6000000 0.4037267 0.009836066
## 6 0.4878049 0.4875000 -0.050656660
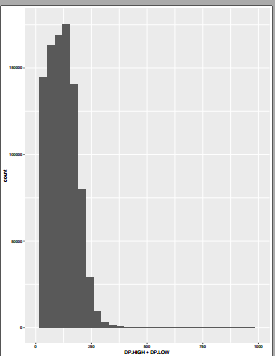
先来看看测序深度的直方图
再来看看参考基因组的频率分布
再来看一下每个混池的snp-index,对于F2群体,大部分位点再0.5.(非连锁位点)
过滤SNP位点
df_filt <-
filterSNPs(
SNPset =df,
refAlleleFreq =0.20,
minTotalDepth =100,
maxTotalDepth =400,
depthDifference =100,
minSampleDepth =40,
minGQ =99,
verbose =TRUE
)
QTLseq analysis
开始分析
df_filt <-runQTLseqAnalysis(df_filt,
windowSize =1e6,
popStruc ="F2",
bulkSize =c(385,430),
replications =10000,
intervals =c(95,99)
)
这里好像只适用于F2与RIL,这个世界总是对BIL充满了恶意。没关系,BIL亲测也适用,
G分析。
df_filt <-runGprimeAnalysis(df_filt,
windowSize =1e6,
outlierFilter ="deltaSNP",
filterThreshold =0.1)
现在开始QTL分析
现在我们对过滤后的数据感到满意,看起来G ’接近于对数正态分布,我们
可以绘制一些全基因组数据并最终尝试识别QTL。
p1 <-plotQTLStats(SNPset =df_filt,var ="nSNPs")
从p1图中,我们可以得到全基因组snp密度。
p2 <-plotQTLStats(SNPset =df_filt,var ="deltaSNP",plotIntervals =TRUE)
超过置信区间的有4个。其中红色p<0.05,绿色P<0.01.
我们再来看一下G'是否是显著的,并且FDR (q)<0.01
p3 <-plotQTLStats(SNPset =df_filt,var ="Gprime",plotThreshold =TRUE,q =0.01)
QTLplots <-plotQTLStats(
SNPset =df_filt,var ="negLog10Pval",
plotThreshold =TRUE,
q =0.01,
subset =c("Chr1","Chr8")
)
提取显著性区间的信息
QTL <-getSigRegions(SNPset =df_filt,alpha =0.01)
提取出csv格式的数据
results <-getQTLTable(SNPset =df_filt,method ="Gprime",alpha =0.01,export =FALSE)