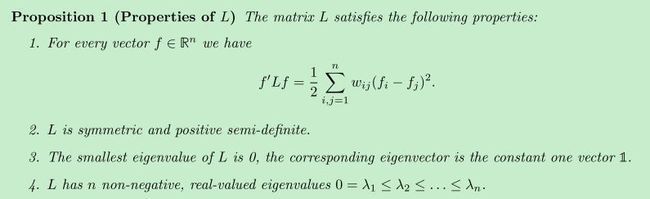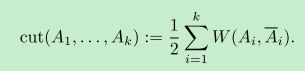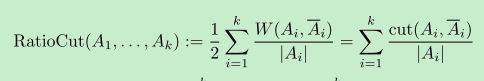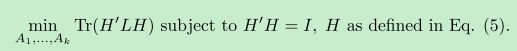谱聚类(spectral clustering)
1. 谱聚类
给你博客园上若干个博客,让你将它们分成K类,你会怎样做?想必有很多方法,本文要介绍的是其中的一种——谱聚类。
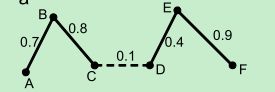
聚类的直观解释是根据样本间相似度,将它们分成不同组。谱聚类的思想是将样本看作顶点,样本间的相似度看作带权的边,从而将聚类问题转为图分割问题:找到一种图分割的方法使得连接不同组的边的权重尽可能低(这意味着组间相似度要尽可能低),组内的边的权重尽可能高(这意味着组内相似度要尽可能高)。将上面的例子代入就是将每一个博客当作图上的一个顶点,然后根据相似度将这些顶点连起来,最后进行分割。分割后还连在一起的顶点就是同一类了。更具体的例子如下图所示:
在上图中,一共有6个顶点(博客),顶点之间的连线表示两个顶点的相似度,现在要将这图分成两半(两个类),要怎样分割(去掉哪边条)?根据谱聚类的思想,应该去掉的边是用虚线表示的那条。最后,剩下的两半就分别对应两个类了。
根据这个思想,可以得到unnormalized谱聚类和normalized谱聚类,由于前者比后者简单,所以本文介绍unnormalized谱聚类的几个步骤(假设要分K个类):
(a)建立similarity graph,并用 W 表示similarity graph的带权邻接矩阵
(b)计算unnormalized graph Laplacian matrix L(L = D - W, 其中D是degree matrix)
(c)计算L的前K个最小的特征向量
(d)把这k个特征向量排列在一起组成一个N*k的矩阵,将其中每一行看作k维空间中的一个向量,并使用 K-means 算法进行聚类
2. 算法原理解析
这一节主要从大体上解释unnormalized谱聚类的四个步骤是怎么来的,不涉及具体的公式推导。
(a)谱聚类的思想就是要转化为图分割问题。因此,第一步就是将原问题转化为图。转为图有两个问题要解决:一是两个顶点的边要怎样定义;二是要保留哪些边。
对于第一个问题,如果两个点在一定程度上相似,就在两个点之间添加一条边。相似的程度由边的权重表示(上图中边上面的数值就是权重了)。因此,只要是计算相似度的公式都可用,不过常用的是Gaussian similarity function
要保留部分边的原因有:边太多了不好处理;权重太低的边是多余的。常用的保留边的方法是建立k-nearest neighbor graph。在这种图中,每个顶点只与K个相似度最高的点连边。
(b)unnormalized graph Laplacian matrix(以下用L表示)有很多很好的性质,也正是这个原因,才要在第二步中计算这么一个矩阵。最重要的性质是下面这一组性质:
这一组性质将在之后的公式推导中起到决定性作用。
(c)将原问题转化为图后,接下来的工作就是决定怎样分割了。图分割问题实际上就是最小割问题(mincut problem)。最小割问题可定义为最小化以下目标函数:
其中k表示分成k个组,Ai表示第i个组,![]() 表示第Ai的补集,W(A,B)表示第A组与第B组之间的所有边的权重之和。
表示第Ai的补集,W(A,B)表示第A组与第B组之间的所有边的权重之和。
这个式子的直观意义:如果要分成K个组,那么其代价就是进行分割时去掉的边的权重的总和。可惜的是直接最小化这式子通常会导致不好的分割。以分成2类为例,这个式子通常会将图分成这样的两类:一个点为一类,剩下的所有点为另一类。显然,这样的分割是很不好的。因为我们期望着每个类都有合理的大小。所以,要对这个式子进行改进,改进后的公式(称为RatioCut)如下:
其中|A|表示A组中包含的顶点数目。
在RatioCut中,如果某一组包含的顶点数越少,那么它的值就越大。在一个最小化问题中,这相当于是惩罚,也就是不鼓励将组分得太小。现在只要将最小化RatioCut解出来,分割就完成了。不幸的是,这是个NP难问题。想要在多项式时间内解出来,就要对这个问题作一个转化了。在转化的过程中,就用到上面提到的L的那一组性质,经过若干推导,最后可以得到这样的一个问题:
如果H矩阵的元素![]() 不为0,则说明第i个点属于第j个类。也就是说,只要得到H矩阵,就能知道要怎样分割了。可惜的是,这个问题仍然是NP难问题。但是,如果我们让H矩阵的元素能够取任意值,这个问题就变成多项式时间内可解的了,此时问题变为:
不为0,则说明第i个点属于第j个类。也就是说,只要得到H矩阵,就能知道要怎样分割了。可惜的是,这个问题仍然是NP难问题。但是,如果我们让H矩阵的元素能够取任意值,这个问题就变成多项式时间内可解的了,此时问题变为:
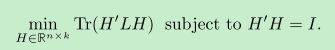
根据Rayleigh-Ritz theorem,这个问题的解是L的前k个最小的特征向量组成的矩阵H,其中特征向量是按列来排,即H的每一列,均为一个特征向量。
(d)在第三步中,我们为了松驰NP难问题,让H矩阵取任意值,因此,解出来的H矩阵不再具有原来的性质——元素值能指出哪个点属于哪一类。尽管如此,对于k-means来说,将H矩阵的每一行当作一个点进行聚类还是挺轻松的。因此,用k-means对H矩阵进行聚类作为谱聚类的最终结果。
3. 谱聚类的实现
以下是unnormalized谱聚类的MATLAB版实现(博客园的代码格式选择中居然没有Matlab的。。。这里选个C++的):
function [ C, L, D, Q, V ] = SpectralClustering(W, k) % spectral clustering algorithm % input: adjacency matrix W; number of cluster k % return: cluster indicator vectors as columns in C; unnormalized Laplacian L; degree matrix D; % eigenvectors matrix Q; eigenvalues matrix V % calculate degree matrix degs = sum(W, 2); D = sparse(1:size(W, 1), 1:size(W, 2), degs); % compute unnormalized Laplacian L = D - W; % compute the eigenvectors corresponding to the k smallest eigenvalues % diagonal matrix V is NcutL's k smallest magnitude eigenvalues % matrix Q whose columns are the corresponding eigenvectors. [Q, V] = eigs(L, k, 'SA'); % use the k-means algorithm to cluster V row-wise % C will be a n-by-1 matrix containing the cluster number for each data point C = kmeans(Q, k); % convert C to a n-by-k matrix containing the k indicator vectors as columns C = sparse(1:size(D, 1), C, 1); end
4. 相关资料
如果想更好地了解谱聚类的话,强烈推荐[1];如果想全面地了解聚类的话,强烈推荐[2]
[1]A Tutorial on Spectral Clustering
[2]漫谈 Clustering 系列