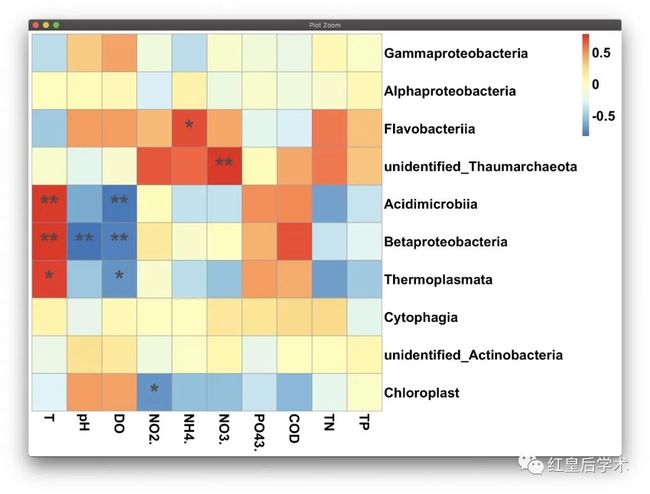
spearman相关性分析_环境因子与主要物种丰度的相关性热图!!
写在前面
本文章旨在给出一个简便易用的环境因子与主要物种丰度的相关性分析及结果展示方式,适用于环境因子和主要物种相对较少的情况。
如果样本数目过多、微生物群落十分复杂,建议使用网络分析评估特定环境因子与不同物种之间的关系,网络分析涉及的内容较多,过一阵应该会专门开个专题来说一说。
我在本文所进行的分析关注的只有物种与环境因子的关系,而对于物种与物种、环境因子和环境因子之间的关系并不关注,因此使用的是不对称的相关矩阵进行可视化,只给出了一种结果可视化的方法。
在R语言中,对于相关性矩阵可视化的方法非常多,不过大多都是基于对称的相关性矩阵,比如cpairs包、corrgram包、corrplot包、ggcorrplot包等等,网上的文章也挺多的,大家可以自行了解一下。
环境因子关联分析
上一节中我介绍了环境因子关联分析中最常用的CCA和RDA,这两种分析都是用来确定环境因子与整体微生物群落之间的相关性,更够得到在微生物群落变化过程中发挥主要作用的环境因子。
在科学研究中,我们通常还想要知道另一个问题,不同的环境因子到底能够调控哪些微生物的变化,当这一问题有了结果之后,我们就可以通过尝试通过调控特定的环境因子,改变特定功能微生物在群落中的丰度,从而达到一些特异性的目的。
如果只通过CCA和RDA是不足以得到这一结果的,此时我们需要使用不同微生物的丰度与环境因子进行相关性分析,从而得到与环境因子相关的物种信息。
一般来说,在进行此项分析之前,首先会对微生物群落数据进行一个初步的筛选,挑选出主要的微生物物种用于分析。
选择主要物种的目的一方面是去除低丰度物种,这些物种在微生物群落中作用有限,我们无需过分关注,另一方面也是由于过多的物种会导致结果太多,不利于结果的展示。
物种的选择没有具体标准,根据实际情况进行选择即可,可以使用门水平的物种也可以使用属水平的物种,可以选择丰度排名前10的物种,也可以选择丰度排名前30的物种。
结果解释
此项分析使用热图进行结果展示,通常情况下行代表不同的物种,列代表不同的环境因子。
色块的颜色表示相关系数也就是R2,一般来说红色代表正相关、蓝色代表负相关 (我说的只是一般情况,要注意比对图例)。
如果某一个环境因子与某一物种丰度之间相关性的p值小于0.05,则在其对应色块中标注星号,表示显著相关,如果p值小于0.01,则标注两个星号,表示极显著相关。
相关性计算
在计算两个变量之间的相关性时,我们最长使用的是两种相关性算法,Pearson和Spearman。
这两种算法的区别在于Pearson要求两个变量均符合正态分布,而当其中一个变量不符合正态分布时,就需要使用Spearman。
因为我们计算的是微生物物种丰度与环境因子的相关性,环境因子多种多样,很容易就会出现不符合正态分布的数据,因此大部分情况下使用的都是Spearman算法。
相关性的计算使用psych包中的corr.test()函数完成,其可以同时得到相关系数和显著性p值。
先来介绍一下corr.test()函数的使用方法。
corr.test(x, y = NULL, use = "pairwise",method="pearson",adjust="holm", alpha=.05,ci=TRUE,minlength=5)各参数意义“
x为一个用于分析的数据框,这里就是物种丰度数据;
y为另一个用于分析的数据框,这里就是环境因子数据;
method表示用于相关性分析的方法,参数包括pearson、spearman和kendall;
adjust表示是否对p值进行校正,参数包括holm、hochberg、hommel、bonferron、BH、BY、fdr和none。
其它参数默认即可。
计算相关性需要物种丰度和环境因子两个文件,要求样本为行,因子为列,并且物种的样本和环境因子的样本要一一对应,同时数据中不能含有NA。
sampledata "class.txt", header = TRUE,row.names = 1,sep = "\t")sampledata sampledata as.data.frame(sampledata)sampledata[is.na(sampledata)] 0envdata "environment.txt", header=TRUE,row.names=1,sep="\t")envdata[is.na(envdata)] 0library(psych)res "spearman",adjust = "holm")得到的结果是一个列表,之后我们将相关系数和显著性p值分别进行保存。
write.table(res$r,"correlation.xls",sep="\t",quote=FALSE,col.names=NA)write.table(res$p,"pvalue.xls",sep="\t",quote=FALSE,col.names=NA)pheatmap实现
接下来根据相关性计算结果进行绘图。
library(pheatmap)pheatmap(res$r, fontsize_number=35,fontsize = 20,cluster_rows = FALSE, display_numbers = matrix(ifelse(res$p <= 0.01, "**", ifelse(res$p <= 0.05 ,"*"," ")), nrow(res$p)), cluster_cols = FALSE,fontface = "bold")pheatmap的相关参数已经在之前的推文中进行了介绍,各位如果想要修改图形参数,可以参考之前的推文。
关注公众号“红皇后学术”,后台回复“相关性热图”获取示例文件和完整代码。
扩展阅读
- 高通量测序基础知识
- 转录组测序技术和结果解读
- 红皇后学术文献解读列表
- 基本分子生物学实验
- PAST:最简便易用的统计学分析软件教程目录
- 每天学习一点R系列
- 微生物群落数据分析系列教程
- 微生物研究相关工具
- 微生物研究投稿期刊简介